-検索条件
-検索結果
検索 (著者・登録者: mclellan & js)の結果120件中、1から50件目までを表示しています

EMDB-42983: 
Structure of the Human Respirovirus 3 Fusion Protein Bound to Camelid Nanobodies 4C03 and 4C06

EMDB-42987: 
Structure of the Human Respirovirus 3 Fusion Protein Bound to Camelid Nanobodies 1D10 and 4C06

EMDB-41374: 
Antibody N3-1 bound to RBDs in the up and down conformations
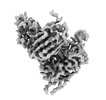
EMDB-41382: 
Antibody N3-1 bound to RBD in the up conformation
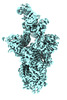
EMDB-41399: 
Antibody N3-1 bound to SARS-CoV-2 spike

EMDB-42940: 
Dimer of Hendra virus prefusion F trimers

EMDB-42942: 
Dimer of Langya virus prefusion F trimers

EMDB-41825: 
Prefusion-stabilized Langya virus F protein, variant G99C/I109C

EMDB-29452: 
Structure Of Respiratory Syncytial Virus Polymerase with Novel Non-Nucleoside Inhibitor

EMDB-40272: 
BtCoV-422 in complex with neutralizing antibody JC57-11

EMDB-40306: 
BtCoV-422 spike in complex with JC57-11 Fab, global map

EMDB-40310: 
BtCoV-422 spike in complex with JC57-11 Fab, local map

EMDB-41089: 
Cryo-EM structure of RSV preF in complex with Fab 2.4K

EMDB-41158: 
CS2it1p2_F7K local refinement for HCMV Trimer in complex with CS2it1p2_F7K Fab and CS4tt1p1_E3K Fab

EMDB-41180: 
Global reconstruction for HCMV Pentamer in complex with CS2pt1p2_A10L Fab and CS3pt1p4_C1L Fab

EMDB-41156: 
HCMV Trimer in complex with CS2it1p2_F7K Fab and CS4tt1p1_E3K Fab

EMDB-41157: 
Global reconstruction for HCMV Trimer in complex with CS2it1p2_F7K Fab and CS4tt1p1_E3K Fab

EMDB-41160: 
CS4tt1p1_E3K local refinement for HCMV Trimer in complex with CS2it1p2_F7K Fab and CS4tt1p1_E3K Fab

EMDB-41161: 
gH base local refinement for HCMV Trimer in complex with CS2it1p2_F7K Fab and CS4tt1p1_E3K Fab

EMDB-41179: 
HCMV Pentamer in complex with CS2pt1p2_A10L Fab and CS3pt1p4_C1L Fab

EMDB-41181: 
CS3pt1p4_C1L local refinement for HCMV Pentamer in complex with CS2pt1p2_A10L Fab and CS3pt1p4_C1L Fab
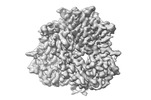
EMDB-27566: 
Prefusion-stabilized Nipah virus fusion protein

EMDB-27577: 
Prefusion-stabilized Hendra virus fusion protein

EMDB-27590: 
Prefusion-stabilized Nipah virus fusion protein, dimer of trimers

EMDB-27562: 
Postfusion Nipah virus fusion protein in complex with Fab 1H1, low resolution reconstruction including the six helix bundle

EMDB-27541: 
Postfusion Nipah virus fusion protein in complex with Fab 1H1

EMDB-26652: 
Prefusion-stabilized Nipah virus fusion protein complexed with Fab 4H3

EMDB-26658: 
Prefusion-stabilized Nipah virus fusion protein complexed with Fab 2D3

EMDB-26659: 
Prefusion-stabilized Nipah virus fusion protein complexed with Fab 1H8

EMDB-26660: 
Prefusion-stabilized Nipah virus fusion protein complexed with Fab 1H1

EMDB-26662: 
Prefusion-stabilized Nipah virus fusion protein complexed with Fab 2B12

EMDB-26668: 
Prefusion-stabilized Nipah virus fusion protein complexed with Fab 1A9
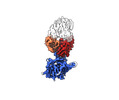
EMDB-25149: 
Antibody A7V3 bound to N-terminal domain of the spike

EMDB-25150: 
Antibody N3-1 bound to SARS-CoV-2 spike

EMDB-25151: 
Antibody A7V3 bound to SARS-CoV-2 spike

EMDB-25929: 
SAN27-14 bound to a antigenic site V on prefusion-stabilized hMPV F

EMDB-25982: 
Cryo-EM structure of hMPV preF bound by Fabs MPE8 and SAN32-2

EMDB-25618: 
SARS-CoV-2 VFLIP spike boung to 2 Ab12 Fab fragments
EMDB-25663: 
SARS-CoV-2 S (Spike Glycoprotein) D614G with Three (3) RBDs Up, Bound to Antibody 2-7 scFv, composite map

EMDB-25689: 
SARS-CoV-2 S (Spike Glycoprotein) D614G with Three (3) RBDs Up, Bound to Antibody 2-7 scFv, global map with poorly-resolved RBDs and scFvs

EMDB-25690: 
SARS-CoV-2 S (Spike Glycoprotein) D614G with Three (3) RBDs Up, Bound to Antibody 2-7 scFv, local refinement map

EMDB-25711: 
SARS-CoV-2 S (Spike Glycoprotein) D614G with One(1) RBD Up

EMDB-26738: 
Integrin alphaM/beta2 ectodomain in complex with adenylate cyclase toxin RTX751 and M1F5 Fab

EMDB-26739: 
Integrin alphaM/beta2 ectodomain

EMDB-27122: 
Global refinement for integrin alphaM/beta2 ectodomain in complex with adenylate cyclase toxin RTX751 and M1F5 Fab

EMDB-27123: 
Tailpiece local refinement for integrin alphaM/beta2 ectodomain in complex with adenylate cyclase toxin RTX751 and M1F5 Fab

EMDB-27124: 
Acylation domain local refinement for integrin alphaM/beta2 ectodomain in complex with adenylate cyclase toxin RTX751 and M1F5 Fab

EMDB-27125: 
Global refinement for integrin alphaM/beta2 ectodomain

EMDB-27126: 
Tailpiece local refinement for integrin alphaM/beta2 ectodomain

EMDB-27024: 
Prefusion-stabilized hMPV fusion protein bound to ADI-61026 and MPE8 Fabs
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
