-検索条件
-検索結果
検索 (著者・登録者: wilkinson & me)の結果84件中、1から50件目までを表示しています

EMDB-42812: 
PNMA2 capsid, overall icosahedral map

EMDB-42815: 
PNMA2 capsid, focussed refinement of a pentamer (C5 symmetry)

EMDB-16876: 
Structure of the Tau-PAM4 Type 1 amyloid fibril

EMDB-16881: 
Structure of the Fmoc-Tau-PAM4 Type 2 amyloid fibril

EMDB-16883: 
Structure of the Fmoc-Tau-PAM4 Type 3 amyloid fibril

EMDB-16886: 
Structure of the Fmoc-Tau-PAM4 Type 4 amyloid fibril

PDB-8oh2: 
Structure of the Tau-PAM4 Type 1 amyloid fibril

PDB-8ohi: 
Structure of the Fmoc-Tau-PAM4 Type 2 amyloid fibril

PDB-8ohp: 
Structure of the Fmoc-Tau-PAM4 Type 3 amyloid fibril
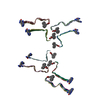
PDB-8oi0: 
Structure of the Fmoc-Tau-PAM4 Type 4 amyloid fibril

EMDB-18301: 
In-tissue cryo electron tomograms of App^NL-G-F amyloid plaques

EMDB-16018: 
Sarkosyl-extracted AppNL-G-F Abeta42 fibril structure

EMDB-16019: 
Sarkosyl-extracted AppNL-G-F Abeta42 fibril structure (Methoxy-X04-labelled mice)

PDB-8bfa: 
Sarkosyl-extracted AppNL-G-F Abeta42 fibril structure
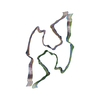
PDB-8bfb: 
Sarkosyl-extracted AppNL-G-F Abeta42 fibril structure (Methoxy-X04-labelled mice)

EMDB-40033: 
Bombyx mori R2 retrotransposon initiating target-primed reverse transcription

EMDB-28064: 
Cas7-11 in complex with Csx29, focus refined on Cas7-11

EMDB-28065: 
Cas7-11 in complex with DR-mismatched target RNA, Csx29 and Csx30

EMDB-28070: 
Cas7-11 in complex with Csx29, focus refined on Csx29

EMDB-28071: 
Cas7-11 in complex with DR-mismatched target RNA, Csx29 and Csx30, focus refined on Cas7-11 INS domain

EMDB-28072: 
Cas7-11 in complex with DR-mismatched target RNA, Csx29 and Csx30, focus refined on Csx29

EMDB-28073: 
Cas7-11 in complex with DR-mismatched target RNA, Csx29 and Csx30, focus refined on Csx29 CHAT domain and Csx30

EMDB-26723: 
Structure of IsrB ternary complex with RNA mutant and target DNA

EMDB-27533: 
Structure of Desulfovirgula thermocuniculi IsrB (DtIsrB) in complex with omega RNA and target DNA

EMDB-27421: 
Avs3 bound to phage PhiV-1 terminase, C2 refinement of Cap4 nuclease domain

EMDB-27422: 
Avs4 bound to phage PhiV-1 portal, C2 refinement of Mrr nuclease domain
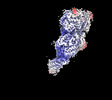
EMDB-27424: 
Avs3 bound to PhiV-1 terminase, symmetry-expanded C1 refinement of TPR-terminase domain
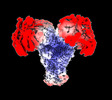
EMDB-27425: 
Avs4 bound to phage PhiV-1 portal, overall C2 reconstruction
EMDB-27426: 
Avs4 bound to phage PhiV-1 portal, symmetry-expanded C1 refinement of TPR-portal domain

EMDB-12108: 
Yeast C complex spliceosome, focused refinement on U2 snRNP

EMDB-12106: 
Yeast C complex spliceosome at 2.8 Angstrom resolution with Prp18/Slu7 bound, composite map

EMDB-12109: 
Yeast C complex spliceosome, focused refinement on NTC

EMDB-12110: 
Yeast C complex spliceosome, focused refinement on Brr2/Prp8-Jab1 and Prp16

EMDB-12111: 
Yeast C complex spliceosome, focused refinement on just Brr2/Prp8-Jab1

EMDB-12112: 
Yeast C complex spliceosome, overall map after focused classification on Prp17

EMDB-12113: 
Yeast C complex spliceosome, overall map after focused classification on Slu7 zinc knuckle

EMDB-12114: 
Yeast C complex spliceosome, overall map after focused classification on Prp18

EMDB-12115: 
Yeast C complex spliceosome, reconstructed signal-subtracted map after focused classification on Prp19

EMDB-12116: 
Yeast C complex spliceosome, overall map after focused classification on Prp19

EMDB-12117: 
Yeast C complex spliceosome, overall map after focused classification on U5 Sm ring

EMDB-12118: 
Yeast C complex spliceosome, overall map after focused classification on Cwc22 NTD

EMDB-12107: 
Yeast C complex spliceosome, focused refinement on core

EMDB-22829: 
Human Tom70 in complex with SARS CoV2 Orf9b

PDB-7kdt: 
Human Tom70 in complex with SARS CoV2 Orf9b

EMDB-10140: 
Post-catalytic P complex spliceosome at 3.3 angstrom resolution from merging data sets

EMDB-4526: 
Human post-catalytic spliceosome (P complex) stalled with DHX8 S717A mutant, overall map

EMDB-4527: 
Human post-catalytic spliceosome (P complex) stalled with DHX8 K594A mutant, focused refinement on core

EMDB-4528: 
Human post-catalytic spliceosome (P complex) stalled with DHX8 S717A mutant, focused refinement on core

EMDB-4529: 
Human post-catalytic spliceosome (P complex), focused refinement on Aquarius and SYF1

EMDB-4530: 
Human post-catalytic spliceosome (P complex), focused refinement on Brr2
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
