-検索条件
-検索結果
検索 (著者・登録者: chiu & w)の結果783件中、1から50件目までを表示しています

EMDB-36849: 
Nipah virus Attachment glycoprotein with 41-6 antibody fragment

PDB-8k3c: 
Nipah virus Attachment glycoprotein with 41-6 antibody fragment

EMDB-36760: 
Cryo-EM structure of conformation 1 of complex of Nipah virus attachment glycoprotein G with 1E5 neutralizing antibody

EMDB-36761: 
Cryo-EM structure of conformation 2 of complex of Nipah virus attachment G with 1E5 neutralizing antibody

PDB-8sjx: 
Structure of lens aquaporin-0 array in sphingomyelin/cholesterol bilayer (2SM:1Chol)

PDB-8sjy: 
Structure of lens aquaporin-0 array in sphingomyelin/cholesterol bilayer (1SM:2Chol)

EMDB-41637: 
Asymmetric cryoEM reconstruction of Mayaro virus

EMDB-28979: 
Cryo-EM structure of Chikungunya virus asymmetric unit

EMDB-41096: 
Cryo-electron tomography of Chikungunya virus pentamer structure

EMDB-41631: 
Cryo-EM structure of Chikungunya virus with asymmetric reconstruction

PDB-8fcg: 
Cryo-EM structure of Chikungunya virus asymmetric unit

EMDB-42820: 
SARS-CoV-2 5' proximal stem-loop 5 and 6 with SL5b extended

EMDB-28138: 
3D reconstruction of the apical complex of Plasmodium falciparum (3D7) free merozoite

EMDB-28141: 
3D reconstruction of the apical complex of Plasmodium falciparum (3D7) free merozoite
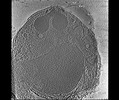
EMDB-28142: 
3D Reconstruction of Plasmodium falciparum (3D7) free merozoite

EMDB-36062: 
Asfv topoisomerase 2 - apo conformer Ia

EMDB-36063: 
Asfv topoisomerase 2 - apo conformer Ib

EMDB-36064: 
Cryo-EM structure of Asfv topoisomerase 2 - apo conformer IIa

EMDB-36065: 
Cryo-EM structure of Asfv topoisomerase 2 - apo conformer IIb

EMDB-36066: 
Cryo-EM structure of Asfv topoisomerase 2 - apo conformer IIIa

EMDB-36067: 
Cryo-EM structure of Asfv topoisomerase 2 - apo conformer IIIb

EMDB-36116: 
Cryo-EM structure of the African swine fever virus topoisomerase 2 complexed with Cut02aDNA and etoposide (EDI-1)

EMDB-36117: 
Cryo-EM structure of the African swine fever virus topoisomerase 2 complexed with Cut02bDNA and etoposide (EDI-2)

EMDB-36118: 
Cryo-EM structure of the African swine fever virus topoisomerase 2 complexed with Cut02aDNA and m-AMSA (EDI-3)
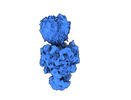
EMDB-36473: 
Cryo-EM structure of the full-length African swine fever virus topoisomerase 2 complexed with Cut02aDNA and etoposide

PDB-8j87: 
Asfv topoisomerase 2 - apo conformer Ia

PDB-8j88: 
Asfv topoisomerase 2 - apo conformer Ib

PDB-8j89: 
Cryo-EM structure of Asfv topoisomerase 2 - apo conformer IIa

PDB-8j8a: 
Cryo-EM structure of Asfv topoisomerase 2 - apo conformer IIb

PDB-8j8b: 
Cryo-EM structure of Asfv topoisomerase 2 - apo conformer IIIa

PDB-8j8c: 
Cryo-EM structure of Asfv topoisomerase 2 - apo conformer IIIb

PDB-8j9v: 
Cryo-EM structure of the African swine fever virus topoisomerase 2 complexed with Cut02aDNA and etoposide (EDI-1)

PDB-8j9w: 
Cryo-EM structure of the African swine fever virus topoisomerase 2 complexed with Cut02bDNA and etoposide (EDI-2)

PDB-8j9x: 
Cryo-EM structure of the African swine fever virus topoisomerase 2 complexed with Cut02aDNA and m-AMSA (EDI-3)

EMDB-41126: 
Cryo-EM structure of the human CLC-2 chloride channel transmembrane domain with bound inhibitor AK-42

EMDB-41127: 
Cryo-EM structure of the human CLC-2 chloride channel transmembrane domain Apo state with resolved N-terminal hairpin

EMDB-41128: 
Cryo-EM structure of the human CLC-2 chloride channel transmembrane domain with symmetric C-terminal

EMDB-41129: 
Title: Cryo-EM structure of the human CLC-2 chloride channel transmembrane domain with asymmetric C-terminal

EMDB-41130: 
Cryo-EM structure of the human CLC-2 chloride channel C-terminal domain

PDB-8ta2: 
Cryo-EM structure of the human CLC-2 chloride channel transmembrane domain with bound inhibitor AK-42

PDB-8ta3: 
Cryo-EM structure of the human CLC-2 chloride channel transmembrane domain Apo state with resolved N-terminal hairpin

PDB-8ta4: 
Cryo-EM structure of the human CLC-2 chloride channel transmembrane domain with symmetric C-terminal

PDB-8ta5: 
Title: Cryo-EM structure of the human CLC-2 chloride channel transmembrane domain with asymmetric C-terminal

PDB-8ta6: 
Cryo-EM structure of the human CLC-2 chloride channel C-terminal domain
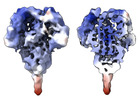
EMDB-29395: 
Subtomogram average of HuCoV-NL63 spike protein from purified intact virions
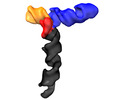
EMDB-42816: 
SARS-CoV-1 5' proximal stem-loop 5

PDB-8uyp: 
SARS-CoV-1 5' proximal stem-loop 5

EMDB-42250: 
Varicella-zoster virus glycoprotein B; H527P prefusion class I.

EMDB-42251: 
Varicella-zoster virus glycoprotein B; H527P prefusion mutant class II.

EMDB-41370: 
Structure of a class A GPCR/Fab complex
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
