+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8pr4 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Dynactin pointed end bound to JIP3 | ||||||||||||
 要素 要素 |
| ||||||||||||
 キーワード キーワード |  MOTOR PROTEIN (モータータンパク質) / MOTOR PROTEIN (モータータンパク質) /  Dynein (ダイニン) / Dynein (ダイニン) /  AAA-Atpase / p150 / AAA-Atpase / p150 /  LIS1 LIS1 | ||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報retrograde axonal transport of mitochondrion /  ダイナクチン / anterograde axonal protein transport / Neutrophil degranulation / MAP-kinase scaffold activity / JUN kinase binding / coronary vasculature development / HSP90 chaperone cycle for steroid hormone receptors (SHR) in the presence of ligand / COPI-independent Golgi-to-ER retrograde traffic / MHC class II antigen presentation ...retrograde axonal transport of mitochondrion / ダイナクチン / anterograde axonal protein transport / Neutrophil degranulation / MAP-kinase scaffold activity / JUN kinase binding / coronary vasculature development / HSP90 chaperone cycle for steroid hormone receptors (SHR) in the presence of ligand / COPI-independent Golgi-to-ER retrograde traffic / MHC class II antigen presentation ...retrograde axonal transport of mitochondrion /  ダイナクチン / anterograde axonal protein transport / Neutrophil degranulation / MAP-kinase scaffold activity / JUN kinase binding / coronary vasculature development / HSP90 chaperone cycle for steroid hormone receptors (SHR) in the presence of ligand / COPI-independent Golgi-to-ER retrograde traffic / MHC class II antigen presentation / COPI-mediated anterograde transport / aorta development / axon regeneration / ventricular septum development / dynein complex binding / axon development / regulation of JNK cascade / ダイナクチン / anterograde axonal protein transport / Neutrophil degranulation / MAP-kinase scaffold activity / JUN kinase binding / coronary vasculature development / HSP90 chaperone cycle for steroid hormone receptors (SHR) in the presence of ligand / COPI-independent Golgi-to-ER retrograde traffic / MHC class II antigen presentation / COPI-mediated anterograde transport / aorta development / axon regeneration / ventricular septum development / dynein complex binding / axon development / regulation of JNK cascade /  kinesin binding / kinesin binding /  stress fiber / axon cytoplasm / vesicle-mediated transport / stress fiber / axon cytoplasm / vesicle-mediated transport /  sarcomere / mitotic spindle organization / positive regulation of JNK cascade / sarcomere / mitotic spindle organization / positive regulation of JNK cascade /  動原体 / signaling receptor complex adaptor activity / 動原体 / signaling receptor complex adaptor activity /  cell body / cell body /  細胞皮質 / 細胞皮質 /  成長円錐 / cytoplasmic vesicle / 成長円錐 / cytoplasmic vesicle /  核膜 / protein stabilization / 核膜 / protein stabilization /  神経繊維 / 神経繊維 /  ゴルジ体 / ゴルジ体 /  中心体 / 中心体 /  樹状突起 / negative regulation of apoptotic process / perinuclear region of cytoplasm / 樹状突起 / negative regulation of apoptotic process / perinuclear region of cytoplasm /  核質 / 核質 /  細胞質 細胞質類似検索 - 分子機能 | ||||||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト)  Sus scrofa (ブタ) Sus scrofa (ブタ) | ||||||||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.5 Å クライオ電子顕微鏡法 / 解像度: 3.5 Å | ||||||||||||
 データ登録者 データ登録者 | Singh, K. / Lau, C.K. / Manigrasso, G. / Gassmann, R. / Carter, A.P. | ||||||||||||
| 資金援助 |  英国, European Union, 3件 英国, European Union, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Science / 年: 2024 ジャーナル: Science / 年: 2024タイトル: Molecular mechanism of dynein-dynactin complex assembly by LIS1. 著者: Kashish Singh / Clinton K Lau / Giulia Manigrasso / José B Gama / Reto Gassmann / Andrew P Carter /   要旨: Cytoplasmic dynein is a microtubule motor vital for cellular organization and division. It functions as a ~4-megadalton complex containing its cofactor dynactin and a cargo-specific coiled-coil ...Cytoplasmic dynein is a microtubule motor vital for cellular organization and division. It functions as a ~4-megadalton complex containing its cofactor dynactin and a cargo-specific coiled-coil adaptor. However, how dynein and dynactin recognize diverse adaptors, how they interact with each other during complex formation, and the role of critical regulators such as lissencephaly-1 (LIS1) protein (LIS1) remain unclear. In this study, we determined the cryo-electron microscopy structure of dynein-dynactin on microtubules with LIS1 and the lysosomal adaptor JIP3. This structure reveals the molecular basis of interactions occurring during dynein activation. We show how JIP3 activates dynein despite its atypical architecture. Unexpectedly, LIS1 binds dynactin's p150 subunit, tethering it along the length of dynein. Our data suggest that LIS1 and p150 constrain dynein-dynactin to ensure efficient complex formation. | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8pr4.cif.gz 8pr4.cif.gz | 253.2 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8pr4.ent.gz pdb8pr4.ent.gz | 183.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8pr4.json.gz 8pr4.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/pr/8pr4 https://data.pdbj.org/pub/pdb/validation_reports/pr/8pr4 ftp://data.pdbj.org/pub/pdb/validation_reports/pr/8pr4 ftp://data.pdbj.org/pub/pdb/validation_reports/pr/8pr4 | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  17834MC  8pqvC  8pqwC  8pqyC  8pqzC  8pr0C  8pr1C  8pr2C  8pr3C  8pr5C  8ptkC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-タンパク質 , 3種, 4分子 JUXx
| #1: タンパク質 | 分子量: 46250.785 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Sus scrofa (ブタ) / 参照: UniProt: I3LHK5 Sus scrofa (ブタ) / 参照: UniProt: I3LHK5 |
|---|---|
| #2: タンパク質 |  ダイナクチン ダイナクチン分子量: 20703.910 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Sus scrofa (ブタ) / 参照: UniProt: D0G6S1 Sus scrofa (ブタ) / 参照: UniProt: D0G6S1 |
| #5: タンパク質 | 分子量: 65975.398 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: MAPK8IP3, JIP3, KIAA1066 Homo sapiens (ヒト) / 遺伝子: MAPK8IP3, JIP3, KIAA1066発現宿主:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ)参照: UniProt: Q9UPT6 |
-Dynactin subunit ... , 2種, 2分子 WY
| #3: タンパク質 |  ダイナクチン ダイナクチン分子量: 20150.533 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Sus scrofa (ブタ) / 参照: UniProt: A0A286ZK88 Sus scrofa (ブタ) / 参照: UniProt: A0A286ZK88 |
|---|---|
| #4: タンパク質 |  ダイナクチン / Dynactin subunit 4 ダイナクチン / Dynactin subunit 4分子量: 52920.434 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Sus scrofa (ブタ) / 参照: UniProt: A0A4X1TB62 Sus scrofa (ブタ) / 参照: UniProt: A0A4X1TB62 |
-非ポリマー , 2種, 4分子 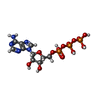


| #6: 化合物 | ChemComp-ATP /  アデノシン三リン酸 アデノシン三リン酸 |
|---|---|
| #7: 化合物 |
-詳細
| 研究の焦点であるリガンドがあるか | N |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Dynactin pointed end bound to JIP3 / タイプ: COMPLEX / Entity ID: #1-#5 / 由来: RECOMBINANT |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 由来(組換発現) | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 緩衝液 | pH: 7.2 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES |
急速凍結 | 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 4000 nm / 最小 デフォーカス(公称値): 500 nm Bright-field microscopy / 最大 デフォーカス(公称値): 4000 nm / 最小 デフォーカス(公称値): 500 nm |
| 撮影 | 電子線照射量: 53 e/Å2 フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) |
- 解析
解析
| EMソフトウェア | 名称: PHENIX / バージョン: 1.20_4459: / カテゴリ: モデル精密化 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||
3次元再構成 | 解像度: 3.5 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 98623 / 対称性のタイプ: POINT | ||||||||||||||||||||||||
| 原子モデル構築 | PDB-ID: 7Z8G Accession code: 7Z8G / Source name: PDB / タイプ: experimental model | ||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj









