+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 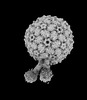 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | AP205 phage Acinetobacter gp16 T4P complex | |||||||||
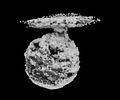
 マップデータ マップデータ | Main Map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  Acinetobacter (アシネトバクター) / SsRNA phage virus / T4P / Acinetobacter (アシネトバクター) / SsRNA phage virus / T4P /  VIRUS (ウイルス) / VIRUS-RNA complex VIRUS (ウイルス) / VIRUS-RNA complex | |||||||||
| 生物種 |   Acinetobacter phage AP205 (ファージ) Acinetobacter phage AP205 (ファージ) | |||||||||
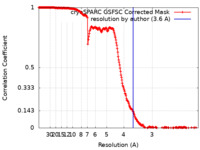
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.6 Å クライオ電子顕微鏡法 / 解像度: 3.6 Å | |||||||||
 データ登録者 データ登録者 | Meng R / Xing Z / Zhang J | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Structural basis of Acinetobacter type IV pili targeting by an RNA virus. 著者: Ran Meng / Zhongliang Xing / Jeng-Yih Chang / Zihao Yu / Jirapat Thongchol / Wen Xiao / Yuhang Wang / Karthik Chamakura / Zhiqi Zeng / Fengbin Wang / Ry Young / Lanying Zeng / Junjie Zhang /  要旨: Acinetobacters pose a significant threat to human health, especially those with weakened immune systems. Type IV pili of acinetobacters play crucial roles in virulence and antibiotic resistance. ...Acinetobacters pose a significant threat to human health, especially those with weakened immune systems. Type IV pili of acinetobacters play crucial roles in virulence and antibiotic resistance. Single-stranded RNA bacteriophages target the bacterial retractile pili, including type IV. Our study delves into the interaction between Acinetobacter phage AP205 and type IV pili. Using cryo-electron microscopy, we solve structures of the AP205 virion with an asymmetric dimer of maturation proteins, the native Acinetobacter type IV pili bearing a distinct post-translational pilin cleavage, and the pili-bound AP205 showing its maturation proteins adapted to pilin modifications, allowing each phage to bind to one or two pili. Leveraging these results, we develop a 20-kilodalton AP205-derived protein scaffold targeting type IV pili in situ, with potential for research and diagnostics. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
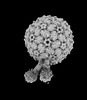
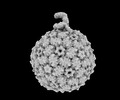
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_41646.map.gz emd_41646.map.gz | 652.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-41646-v30.xml emd-41646-v30.xml emd-41646.xml emd-41646.xml | 16.2 KB 16.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_41646_fsc.xml emd_41646_fsc.xml | 23.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_41646.png emd_41646.png | 59.4 KB | ||
| Filedesc metadata |  emd-41646.cif.gz emd-41646.cif.gz | 4.4 KB | ||
| その他 |  emd_41646_half_map_1.map.gz emd_41646_half_map_1.map.gz emd_41646_half_map_2.map.gz emd_41646_half_map_2.map.gz | 1.2 GB 1.2 GB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41646 http://ftp.pdbj.org/pub/emdb/structures/EMD-41646 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41646 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41646 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_41646.map.gz / 形式: CCP4 / 大きさ: 1.3 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_41646.map.gz / 形式: CCP4 / 大きさ: 1.3 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Main Map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: half map a
| ファイル | emd_41646_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map a | ||||||||||||
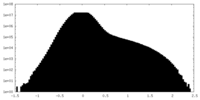
| 投影像・断面図 |
| ||||||||||||
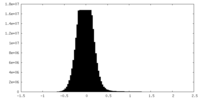
| 密度ヒストグラム |
-ハーフマップ: half map b
| ファイル | emd_41646_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half map b | ||||||||||||
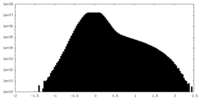
| 投影像・断面図 |
| ||||||||||||
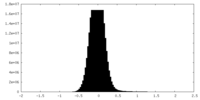
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Acinetobacter phage AP205
| 全体 | 名称:   Acinetobacter phage AP205 (ファージ) Acinetobacter phage AP205 (ファージ) |
|---|---|
| 要素 |
|
-超分子 #1: Acinetobacter phage AP205
| 超分子 | 名称: Acinetobacter phage AP205 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: #1-#5 / NCBI-ID: 154784 / 生物種: Acinetobacter phage AP205 / ウイルスタイプ: VIRION / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: Yes / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  Acinetobacter genomosp. 16BJ (バクテリア) Acinetobacter genomosp. 16BJ (バクテリア) |
| 分子量 | 理論値: 4.54 MDa |
| ウイルス殻 | Shell ID: 1 / 名称: Coat Capsid / 直径: 290.0 Å / T番号(三角分割数): 3 |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 構成要素:
詳細: 20mM Tris-HCl, 150mM NaCl, pH 8.0 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK III / 詳細: 3uL sample applied to a 300-mesh 2/1 copper grid. | |||||||||
| 詳細 | AP205 virion particle |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 130000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 130000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: COUNTING / 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: RIGID BODY FIT |
|---|
 ムービー
ムービー コントローラー
コントローラー














 Z
Z Y
Y X
X