+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Outer Mat-T4P complex | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  Acinetobacter (アシネトバクター) / SsRNA phage virus / T4P / Acinetobacter (アシネトバクター) / SsRNA phage virus / T4P /  VIRUS (ウイルス) VIRUS (ウイルス) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報virion attachment to host cell pilus /  protein secretion by the type II secretion system / protein secretion by the type II secretion system /  type II protein secretion system complex / type II protein secretion system complex /  性繊毛 / 性繊毛 /  virion component / virion component /  細胞接着 / 細胞接着 /  生体膜 生体膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Acinetobacter phage AP205 (ファージ) / Acinetobacter phage AP205 (ファージ) /  Acinetobacter genomosp. 16BJ (バクテリア) Acinetobacter genomosp. 16BJ (バクテリア) | |||||||||
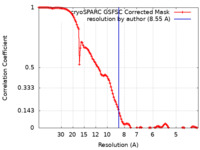
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 8.55 Å クライオ電子顕微鏡法 / 解像度: 8.55 Å | |||||||||
 データ登録者 データ登録者 | Meng R / Xing Z / Thongchol J / Zhang J | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Structural basis of Acinetobacter type IV pili targeting by an RNA virus. 著者: Ran Meng / Zhongliang Xing / Jeng-Yih Chang / Zihao Yu / Jirapat Thongchol / Wen Xiao / Yuhang Wang / Karthik Chamakura / Zhiqi Zeng / Fengbin Wang / Ry Young / Lanying Zeng / Junjie Zhang /  要旨: Acinetobacters pose a significant threat to human health, especially those with weakened immune systems. Type IV pili of acinetobacters play crucial roles in virulence and antibiotic resistance. ...Acinetobacters pose a significant threat to human health, especially those with weakened immune systems. Type IV pili of acinetobacters play crucial roles in virulence and antibiotic resistance. Single-stranded RNA bacteriophages target the bacterial retractile pili, including type IV. Our study delves into the interaction between Acinetobacter phage AP205 and type IV pili. Using cryo-electron microscopy, we solve structures of the AP205 virion with an asymmetric dimer of maturation proteins, the native Acinetobacter type IV pili bearing a distinct post-translational pilin cleavage, and the pili-bound AP205 showing its maturation proteins adapted to pilin modifications, allowing each phage to bind to one or two pili. Leveraging these results, we develop a 20-kilodalton AP205-derived protein scaffold targeting type IV pili in situ, with potential for research and diagnostics. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
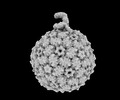
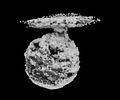
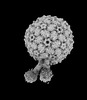
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_41635.map.gz emd_41635.map.gz | 81 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-41635-v30.xml emd-41635-v30.xml emd-41635.xml emd-41635.xml | 18.7 KB 18.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_41635_fsc.xml emd_41635_fsc.xml | 11.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_41635.png emd_41635.png | 42.9 KB | ||
| Filedesc metadata |  emd-41635.cif.gz emd-41635.cif.gz | 6.1 KB | ||
| その他 |  emd_41635_half_map_1.map.gz emd_41635_half_map_1.map.gz emd_41635_half_map_2.map.gz emd_41635_half_map_2.map.gz | 151.6 MB 151.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41635 http://ftp.pdbj.org/pub/emdb/structures/EMD-41635 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41635 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41635 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_41635.map.gz / 形式: CCP4 / 大きさ: 163.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_41635.map.gz / 形式: CCP4 / 大きさ: 163.6 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 2.12 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
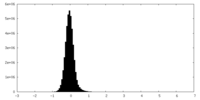
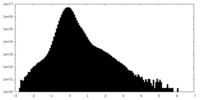
-ハーフマップ: #2
| ファイル | emd_41635_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
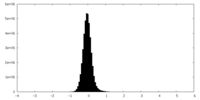
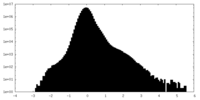
-ハーフマップ: #1
| ファイル | emd_41635_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Acinetobacter phage AP205
| 全体 | 名称:   Acinetobacter phage AP205 (ファージ) Acinetobacter phage AP205 (ファージ) |
|---|---|
| 要素 |
|
-超分子 #1: Acinetobacter phage AP205
| 超分子 | 名称: Acinetobacter phage AP205 / タイプ: virus / ID: 1 / 親要素: 0 / 含まれる分子: all / NCBI-ID: 154784 / 生物種: Acinetobacter phage AP205 / ウイルスタイプ: VIRION / ウイルス・単離状態: SPECIES / ウイルス・エンベロープ: Yes / ウイルス・中空状態: No |
|---|---|
| 宿主 | 生物種:  Acinetobacter genomosp. 16BJ (バクテリア) Acinetobacter genomosp. 16BJ (バクテリア) |
| 分子量 | 理論値: 378 KDa |
| ウイルス殻 | Shell ID: 1 / 名称: Coat Capsid / 直径: 290.0 Å / T番号(三角分割数): 3 |
-分子 #1: Maturation protein
| 分子 | 名称: Maturation protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Acinetobacter phage AP205 (ファージ) Acinetobacter phage AP205 (ファージ) |
| 分子量 | 理論値: 61.063852 KDa |
| 配列 | 文字列: MNMYKWVPES IRDSGEGQPS YSNNGDYAPS GPWVAAGIHT MPQSLRDSMR NSIMVTAQAR RDVIGPEWGP DGRFTGYASV IGTPDPKPA DIVNKFTVER RPVSNGNFQQ RVKAGDIVVA PYTSDGKITV KLVAGQKDIS STPDYDYRID SSLASSAGFV V AGERWYYT ...文字列: MNMYKWVPES IRDSGEGQPS YSNNGDYAPS GPWVAAGIHT MPQSLRDSMR NSIMVTAQAR RDVIGPEWGP DGRFTGYASV IGTPDPKPA DIVNKFTVER RPVSNGNFQQ RVKAGDIVVA PYTSDGKITV KLVAGQKDIS STPDYDYRID SSLASSAGFV V AGERWYYT KRHFIIPRYF QNWRMRRRKY VTGWVMPTFY SPKEIFNRLK DSLVPDTGLV TQVWADNNTK RMDFLTAMAE IP QTLSSFL DALGYLGSLI KDFKRRRFFL NKAHQRIRNK LGVSFAERRS QIVSKYDRKI ASARKPAIIV KLRQRKEKAL KAL DKMRVR EEKKMIREFA TQAASLWLSF RYEIMPLYYQ SQDVLDVIAN STSEFMTSRD FVAKAINIGI PLEWNLDQEN LVSQ PRHNV MVKSKLSPEN NIGKTLSVNP FTTAWELLTL SFVVDWFVNF GDVIAGFTGG YSDDSGATAS WRFDDKKVFH LKNIP SAMV IVDINFYTRQ VIDPRLCGGL AFSPKLNLFR YLDAMSLSWN RSRLKISRAT UniProtKB: Maturation protein |
-分子 #2: Fimbrial protein
| 分子 | 名称: Fimbrial protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 20 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Acinetobacter genomosp. 16BJ (バクテリア) Acinetobacter genomosp. 16BJ (バクテリア) |
| 分子量 | 理論値: 7.470747 KDa |
| 配列 | 文字列: TLIELMIVVA IIGILAAIAI PQYQNYIAKS QVSRVMSETG SLKTVIETCI LDGKTAANCE LGWTNSNLLG UniProtKB: Fimbrial protein |
-分子 #3: Fimbrial protein
| 分子 | 名称: Fimbrial protein / タイプ: protein_or_peptide / ID: 3 / コピー数: 20 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Acinetobacter genomosp. 16BJ (バクテリア) Acinetobacter genomosp. 16BJ (バクテリア) |
| 分子量 | 理論値: 6.999778 KDa |
| 配列 | 文字列: STAAVTGQTG LTITYPASAT ESAAIQGTFG NSAAIKIKNQ TLTWTRTPEG AWSCATTVEA KFKPAGCAS UniProtKB: Fimbrial protein |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 構成要素:
詳細: 20mM Tris-HCl, 150mM NaCl, pH 8.0 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK III / 詳細: 3uL sample applied to a 300-mesh 2/1 copper grid. | |||||||||
| 詳細 | AP205 virion particle |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 130000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 130000 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 検出モード: COUNTING / 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | プロトコル: RIGID BODY FIT |
|---|---|
| 得られたモデル |  PDB-8tva: |
 ムービー
ムービー コントローラー
コントローラー














 Z
Z Y
Y X
X