+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of Nal1 protein, allele IR64, from Oryza sativa indica cultivar | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  Serine protease (セリンプロテアーゼ) / Serine protease (セリンプロテアーゼ) /  PLANT PROTEIN (植物) PLANT PROTEIN (植物) | |||||||||
| 機能・相同性 | stem vascular tissue pattern formation / internode patterning / regulation of leaf development / leaf vascular tissue pattern formation / Peptidase S1, PA clan /  核質 / 核質 /  細胞質 / Protein NARROW LEAF 1 細胞質 / Protein NARROW LEAF 1 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Oryza sativa Japonica Group (イネ) / Oryza sativa Japonica Group (イネ) /   Oryza sativa Indica Group (イネ) Oryza sativa Indica Group (イネ) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.63 Å クライオ電子顕微鏡法 / 解像度: 2.63 Å | |||||||||
 データ登録者 データ登録者 | Huang LY / Rety S / Xi XG | |||||||||
| 資金援助 |  中国, 中国,  フランス, 2件 フランス, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Plants / 年: 2024 ジャーナル: Nat Plants / 年: 2024タイトル: The catalytic triad of rice NARROW LEAF1 involves H234. 著者: Ling-Yun Huang / Na-Nv Liu / Wei-Fei Chen / Xia Ai / Hai-Hong Li / Ze-Lin Zhang / Xi-Miao Hou / Philippe Fossé / Olivier Mauffret / Dong-Sheng Lei / Stephane Rety / Xu-Guang Xi /   要旨: NARROW LEAF1 (NAL1) exerts a multifaceted influence on leaf morphology and crop yield. Recent crystal study proposed that histidine 233 (H233) is part of the catalytic triad. Here we report that ...NARROW LEAF1 (NAL1) exerts a multifaceted influence on leaf morphology and crop yield. Recent crystal study proposed that histidine 233 (H233) is part of the catalytic triad. Here we report that unlike suggested previously, H234 instead of H233 is a component of the catalytic triad alongside residues D291 and S385 in NAL1. Remarkably, residue 233 unexpectedly plays a pivotal role in regulating NAL1's proteolytic activity. These findings establish a strong foundation for utilizing NAL1 in breeding programs aimed at improving crop yield. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_17768.map.gz emd_17768.map.gz | 203.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-17768-v30.xml emd-17768-v30.xml emd-17768.xml emd-17768.xml | 17 KB 17 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_17768_fsc.xml emd_17768_fsc.xml | 12.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_17768.png emd_17768.png | 134.1 KB | ||
| Filedesc metadata |  emd-17768.cif.gz emd-17768.cif.gz | 6.5 KB | ||
| その他 |  emd_17768_half_map_1.map.gz emd_17768_half_map_1.map.gz emd_17768_half_map_2.map.gz emd_17768_half_map_2.map.gz | 200.2 MB 200.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-17768 http://ftp.pdbj.org/pub/emdb/structures/EMD-17768 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17768 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-17768 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8pn2MC  8pmeC  8pmgC  8pmiC  8pmlC  8pmmC  8pn1C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_17768.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_17768.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.82 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_17768_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
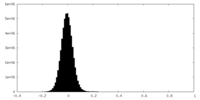
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_17768_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Nal1
| 全体 | 名称: Nal1 |
|---|---|
| 要素 |
|
-超分子 #1: Nal1
| 超分子 | 名称: Nal1 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Oryza sativa Japonica Group (イネ) Oryza sativa Japonica Group (イネ) |
-分子 #1: Protein NARROW LEAF 1
| 分子 | 名称: Protein NARROW LEAF 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Oryza sativa Indica Group (イネ) Oryza sativa Indica Group (イネ) |
| 分子量 | 理論値: 63.822469 KDa |
| 組換発現 | 生物種:   Escherichia coli K-12 (大腸菌) Escherichia coli K-12 (大腸菌) |
| 配列 | 文字列: FHMKPSDDKA QLSGLAQSEE SSLDVDHQSF PCSPSIQPVA SGCTHTENSA AYFLWPTSNL QHCAAEGRAN YFGNLQKGLL PRHPGRLPK GQQANSLLDL MTIRAFHSKI LRRFSLGTAV GFRIRKGDLT DIPAILVFVA RKVHKKWLNP AQCLPAILEG P GGVWCDVD ...文字列: FHMKPSDDKA QLSGLAQSEE SSLDVDHQSF PCSPSIQPVA SGCTHTENSA AYFLWPTSNL QHCAAEGRAN YFGNLQKGLL PRHPGRLPK GQQANSLLDL MTIRAFHSKI LRRFSLGTAV GFRIRKGDLT DIPAILVFVA RKVHKKWLNP AQCLPAILEG P GGVWCDVD VVEFSYYGAP AQTPKEQMFS ELVDKLCGSD ECIGSGSQVA SHETFGTLGA IVKRRTGNKQ VGFLTNRHVA VD LDYPNQK MFHPLPPNLG PGVYLGAVER ATSFITDDVW YGIYAGTNPE TFVRADGAFI PFADDFDIST VTTVVRGVGD IGD VKVIDL QCPLNSLIGR QVCKVGRSSG HTTGTVMAYA LEYNDEKGIC FFTDILVVGE NRQTFDLEGD SGSLIILTSQ DGEK PRPIG IIWGGTANRG RLKLTSDHGP ENWTSGVDLG RLLDRLELDI IITNESLQDA VQQQRFALVA AVTSAVGESS GAPVA IPEE KVEEIFEPLG IQIQQLPRHD VAASGTEGEE ASNTVVNVEE HQFISNFVGM SPVRDDQDAP RSITNLNNPS EEELAM SLH LGDREPKRLR SDSGSSLDLE KLE UniProtKB: Protein NARROW LEAF 1 |
-分子 #2: ADENOSINE-5'-TRIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-TRIPHOSPHATE / タイプ: ligand / ID: 2 / コピー数: 6 / 式: ATP |
|---|---|
| 分子量 | 理論値: 507.181 Da |
| Chemical component information | 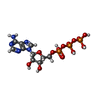 ChemComp-ATP: |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 6 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 / 詳細: 20mM Tris-HCl 100mM NaCl |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 282 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 50.0 µm / 最小 デフォーカス(公称値): 5.0 µm Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 50.0 µm / 最小 デフォーカス(公称値): 5.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| ソフトウェア | 名称: SerialEM (ver. 3.8.4) |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: SUPER-RESOLUTION / 実像数: 700 / 平均電子線量: 49.02 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X