| 登録情報 | データベース: PDB / ID: 6i0j
|
|---|
| タイトル | Crystal structure of human carbonic anhydrase I in complex with the 4-({[4-chloro-3-(trifluoromethyl)phenyl]carbamoyl}amino)phenyl sulfamate inhibitor |
|---|
 要素 要素 | Carbonic anhydrase 1 炭酸脱水酵素 炭酸脱水酵素 |
|---|
 キーワード キーワード |  LYASE (リアーゼ) / LYASE (リアーゼ) /  carbonic anhydrase I (炭酸脱水酵素) / carbonic anhydrase I (炭酸脱水酵素) /  inhibitor (酵素阻害剤) inhibitor (酵素阻害剤) |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 |
|---|
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  フーリエ合成 / 解像度: 1.35 Å フーリエ合成 / 解像度: 1.35 Å |
|---|
 データ登録者 データ登録者 | Ferraroni, M. / Supuran, C.T. / Bozdag, M. / Chiapponi, D. |
|---|
 引用 引用 |  ジャーナル: Eur.J.Med.Chem. / 年: 2019 ジャーナル: Eur.J.Med.Chem. / 年: 2019
タイトル: Carbonic anhydrase inhibitors based on sorafenib scaffold: Design, synthesis, crystallographic investigation and effects on primary breast cancer cells.
著者: Bozdag, M. / Ferraroni, M. / Ward, C. / Carta, F. / Bua, S. / Angeli, A. / Langdon, S.P. / Kunkler, I.H. / Al-Tamimi, A.S. / Supuran, C.T. |
|---|
| 履歴 | | 登録 | 2018年10月26日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2019年11月20日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2020年12月2日 | Group: Database references / Derived calculations / カテゴリ: citation / citation_author / struct_conn
Item: _citation.country / _citation.journal_abbrev ..._citation.country / _citation.journal_abbrev / _citation.journal_id_ASTM / _citation.journal_id_CSD / _citation.journal_id_ISSN / _citation.journal_volume / _citation.page_first / _citation.page_last / _citation.pdbx_database_id_DOI / _citation.pdbx_database_id_PubMed / _citation.title / _citation.year / _struct_conn.pdbx_dist_value / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id |
|---|
| 改定 1.2 | 2024年1月24日 | Group: Data collection / Database references / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 炭酸脱水酵素
炭酸脱水酵素  キーワード
キーワード LYASE (リアーゼ) /
LYASE (リアーゼ) /  carbonic anhydrase I (炭酸脱水酵素) /
carbonic anhydrase I (炭酸脱水酵素) /  inhibitor (酵素阻害剤)
inhibitor (酵素阻害剤) 機能・相同性情報
機能・相同性情報 hydro-lyase activity / Gene and protein expression by JAK-STAT signaling after Interleukin-12 stimulation /
hydro-lyase activity / Gene and protein expression by JAK-STAT signaling after Interleukin-12 stimulation /  cyanamide hydratase /
cyanamide hydratase /  cyanamide hydratase activity /
cyanamide hydratase activity /  arylesterase activity / Reversible hydration of carbon dioxide /
arylesterase activity / Reversible hydration of carbon dioxide /  炭酸脱水酵素 /
炭酸脱水酵素 /  carbonate dehydratase activity / Erythrocytes take up oxygen and release carbon dioxide / Erythrocytes take up carbon dioxide and release oxygen ...
carbonate dehydratase activity / Erythrocytes take up oxygen and release carbon dioxide / Erythrocytes take up carbon dioxide and release oxygen ... hydro-lyase activity / Gene and protein expression by JAK-STAT signaling after Interleukin-12 stimulation /
hydro-lyase activity / Gene and protein expression by JAK-STAT signaling after Interleukin-12 stimulation /  cyanamide hydratase /
cyanamide hydratase /  cyanamide hydratase activity /
cyanamide hydratase activity /  arylesterase activity / Reversible hydration of carbon dioxide /
arylesterase activity / Reversible hydration of carbon dioxide /  炭酸脱水酵素 /
炭酸脱水酵素 /  carbonate dehydratase activity / Erythrocytes take up oxygen and release carbon dioxide / Erythrocytes take up carbon dioxide and release oxygen / one-carbon metabolic process / extracellular exosome / zinc ion binding /
carbonate dehydratase activity / Erythrocytes take up oxygen and release carbon dioxide / Erythrocytes take up carbon dioxide and release oxygen / one-carbon metabolic process / extracellular exosome / zinc ion binding /  細胞質基質
細胞質基質
 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  フーリエ合成 / 解像度: 1.35 Å
フーリエ合成 / 解像度: 1.35 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Eur.J.Med.Chem. / 年: 2019
ジャーナル: Eur.J.Med.Chem. / 年: 2019 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 6i0j.cif.gz
6i0j.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb6i0j.ent.gz
pdb6i0j.ent.gz PDB形式
PDB形式 6i0j.json.gz
6i0j.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/i0/6i0j
https://data.pdbj.org/pub/pdb/validation_reports/i0/6i0j ftp://data.pdbj.org/pub/pdb/validation_reports/i0/6i0j
ftp://data.pdbj.org/pub/pdb/validation_reports/i0/6i0j リンク
リンク 集合体
集合体


 要素
要素 炭酸脱水酵素 / Carbonate dehydratase I / Carbonic anhydrase B / CAB / Carbonic anhydrase I / CA-I
炭酸脱水酵素 / Carbonate dehydratase I / Carbonic anhydrase B / CAB / Carbonic anhydrase I / CA-I
 Homo sapiens (ヒト) / 参照: UniProt: P00915,
Homo sapiens (ヒト) / 参照: UniProt: P00915,  炭酸脱水酵素
炭酸脱水酵素
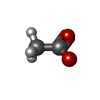







 酢酸塩
酢酸塩 グリセリン
グリセリン 水
水 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製
 シンクロトロン / サイト:
シンクロトロン / サイト:  ESRF
ESRF  / ビームライン: MASSIF-1 / 波長: 0.966 Å
/ ビームライン: MASSIF-1 / 波長: 0.966 Å : 0.966 Å / 相対比: 1
: 0.966 Å / 相対比: 1  解析
解析 :
:  フーリエ合成
フーリエ合成 ムービー
ムービー コントローラー
コントローラー























 PDBj
PDBj






