+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of Asfv topoisomerase 2 - apo conformer IIIa | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  Topoisomerase (DNAトポイソメラーゼ) / Topoisomerase (DNAトポイソメラーゼ) /  ASFV (アフリカ豚熱ウイルス) / ASFV (アフリカ豚熱ウイルス) /  VIRAL PROTEIN (ウイルスタンパク質) / VIRAL PROTEIN (ウイルスタンパク質) /  ISOMERASE (異性化酵素) ISOMERASE (異性化酵素) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報sister chromatid segregation / DNA topoisomerase type II (double strand cut, ATP-hydrolyzing) activity /  DNA topoisomerase (ATP-hydrolysing) / DNA topological change / host cell cytoplasm / DNA topoisomerase (ATP-hydrolysing) / DNA topological change / host cell cytoplasm /  DNA binding / DNA binding /  ATP binding / ATP binding /  metal ion binding / metal ion binding /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |    African swine fever virus (アフリカ豚コレラウイルス) African swine fever virus (アフリカ豚コレラウイルス) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.43 Å クライオ電子顕微鏡法 / 解像度: 2.43 Å | |||||||||
 データ登録者 データ登録者 | Chang C-W / Tsai M-D | |||||||||
| 資金援助 |  台湾, 1件 台湾, 1件
| |||||||||
 引用 引用 |  ジャーナル: Commun Chem / 年: 2024 ジャーナル: Commun Chem / 年: 2024タイトル: A unified view on enzyme catalysis by cryo-EM study of a DNA topoisomerase. 著者: Chiung-Wen Mary Chang / Shun-Chang Wang / Chun-Hsiung Wang / Allan H Pang / Cheng-Han Yang / Yao-Kai Chang / Wen-Jin Wu / Ming-Daw Tsai /   要旨: The theories for substrate recognition in enzyme catalysis have evolved from lock-key to induced fit, then conformational selection, and conformational selection followed by induced fit. However, the ...The theories for substrate recognition in enzyme catalysis have evolved from lock-key to induced fit, then conformational selection, and conformational selection followed by induced fit. However, the prevalence and consensus of these theories require further examination. Here we use cryogenic electron microscopy and African swine fever virus type 2 topoisomerase (AsfvTop2) to demonstrate substrate binding theories in a joint and ordered manner: catalytic selection by the enzyme, conformational selection by the substrates, then induced fit. The apo-AsfvTop2 pre-exists in six conformers that comply with the two-gate mechanism directing DNA passage and release in the Top2 catalytic cycle. The structures of AsfvTop2-DNA-inhibitor complexes show that substantial induced-fit changes occur locally from the closed apo-conformer that however is too far-fetched for the open apo-conformer. Furthermore, the ATPase domain of AsfvTop2 in the MgAMP-PNP-bound crystal structures coexist in reduced and oxidized forms involving a disulfide bond, which can regulate the AsfvTop2 function. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_36066.map.gz emd_36066.map.gz | 122.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-36066-v30.xml emd-36066-v30.xml emd-36066.xml emd-36066.xml | 14.9 KB 14.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_36066_fsc.xml emd_36066_fsc.xml | 14.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_36066.png emd_36066.png | 58.7 KB | ||
| Filedesc metadata |  emd-36066.cif.gz emd-36066.cif.gz | 6.1 KB | ||
| その他 |  emd_36066_half_map_1.map.gz emd_36066_half_map_1.map.gz emd_36066_half_map_2.map.gz emd_36066_half_map_2.map.gz | 226.6 MB 226.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-36066 http://ftp.pdbj.org/pub/emdb/structures/EMD-36066 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36066 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-36066 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8j8bMC  8j87C  8j88C  8j89C  8j8aC  8j8cC  8j9vC  8j9wC  8j9xC  8ja1C  8ja2C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_36066.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_36066.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.83 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
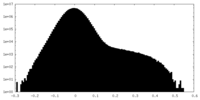
| ファイル | emd_36066_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
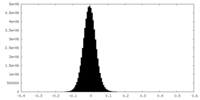
| ファイル | emd_36066_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : ASFV Topoisomerase 2
| 全体 | 名称: ASFV Topoisomerase 2 |
|---|---|
| 要素 |
|
-超分子 #1: ASFV Topoisomerase 2
| 超分子 | 名称: ASFV Topoisomerase 2 / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:    African swine fever virus (アフリカ豚コレラウイルス) African swine fever virus (アフリカ豚コレラウイルス) |
-分子 #1: DNA topoisomerase 2
| 分子 | 名称: DNA topoisomerase 2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:    African swine fever virus (アフリカ豚コレラウイルス) African swine fever virus (アフリカ豚コレラウイルス) |
| 分子量 | 理論値: 136.422578 KDa |
| 組換発現 | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 配列 | 文字列: MEAFEISDFK EHAKKKSMWA GALNKVTISG LMGVFTEDED LMALPIHRDH CPALLKIFDE LIVNATDHER ACHSKTKKVT YIKISFDKG VFSCENDGPG IPIAKHEQAS LIAKRDVYVP EVASCFFLAG TNINKAKDCI KGGTNGVGLK LAMVHSQWAI L TTADGAQK ...文字列: MEAFEISDFK EHAKKKSMWA GALNKVTISG LMGVFTEDED LMALPIHRDH CPALLKIFDE LIVNATDHER ACHSKTKKVT YIKISFDKG VFSCENDGPG IPIAKHEQAS LIAKRDVYVP EVASCFFLAG TNINKAKDCI KGGTNGVGLK LAMVHSQWAI L TTADGAQK YVQQINQRLD IIEPPTITPS REMFTRIELM PVYQELGYAE PLSETEQADL SAWIYLRACQ CAAYVGKGTT IY YNDKPCR TGSVMALAKM YTLLSAPNST IHTATIKADA KPYSLHPLQV AAVVSPKFKK FEHVSIINGV NCVKGEHVTF LKK TINEMV IKKFQQTIKD KNRKTTLRDS CSNIFVVIVG SIPGIEWTGQ RKDELSIAEN VFKTHYSIPS SFLTSMTRSI VDIL LQSIS KKDNHKQVDV DKYTRARNAG GKRAQDCMLL AAEGDSALSL LRTGLTLGKS NPSGPSFDFC GMISLGGVIM NACKK VTNI TTDSGETIMV RNEQLTNNKV LQGIVQVLGL DFNCHYKTQE ERAKLRYGCI VACVDQDLDG CGKILGLLLA YFHLFW PQL IIHGFVKRLL TPLIRVYEKG KTMPVEFYYE QEFDAWAKKQ TSLVNHTVKY YKGLAAHDTH EVKSMFKHFD NMVYTFT LD DSAKELFHIY FGGESELRKR ELCTGVVPLT ETQTQSIHSV RRIPCSLHLQ VDTKAYKLDA IERQIPNFLD GMTRARRK I LAGGVKCFAS NNRERKVFQF GGYVADHMFY HHGDMSLNTS IIKAAQYYPG SSHLYPVFIG IGSFGSRHLG GKDAGSPRY ISVQLASEFI KTMFPAEDSW LLPYVFEDGQ RAEPEYYVPV LPLAIMEYGA NPSEGWKYTT WARQLEDILA LVRAYVDKDN PKHELLHYA IKHKITILPL RPSNYNFKGH LKRFGQYYYS YGTYDISEQR NIITITELPL RVPTVAYIES IKKSSNRMTF I EEIIDYSS SETIEILVKL KPNSLNRIVE EFKETEEQDS IENFLRLRNC LHSHLNFVKP KGGIIEFNSY YEILYAWLPY RR ELYQKRL MREHAVLKLR IIMETAIVRY INESAELNLS HYEDEKEASR ILSEHGFPPL NHTLIISPEF ASIEELNQKA LQG CYTYIL SLQARELLIA AKTRRVEKIK KMQARLDKVE QLLQESPFPG ASVWLEEIDA VEKAIIKGRN TQWKFHHHHH H UniProtKB:  DNA topoisomerase 2 DNA topoisomerase 2 |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.3 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm Bright-field microscopy / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 8900 / 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | Chain - Source name: Other / Chain - Initial model type: experimental model / 詳細: AsfvTop2 conformer IIa |
|---|---|
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT |
| 得られたモデル |  PDB-8j8b: |
 ムービー
ムービー コントローラー
コントローラー














 Z
Z Y
Y X
X