-検索条件
-検索結果
検索 (著者・登録者: zhang & jl)の結果158件中、1から50件目までを表示しています

EMDB-40797: 
BG505 GT1.1 SOSIP in complex with NHP Fabs 21N13, 21M20 and RM20A3

EMDB-35901: 
GK tetramer with adjacent hooks at reaction state

EMDB-35946: 
GK monomer complexes with ADP

EMDB-35900: 
GK monomer complexes with catalytic intermediate

EMDB-35902: 
GK monomer complexes with glutamate and ATP

EMDB-35947: 
GK monomer complexes with G5P and ADP

EMDB-38864: 
RSF-38N38NCP complex Class0

EMDB-29943: 
Cryo-EM Structure of the Prostaglandin E2 Receptor 4 Coupled to G Protein

EMDB-29944: 
Cryo-EM Structure of the Prostaglandin E2 Receptor 4 Coupled to G Protein

EMDB-29945: 
Cryo-EM Structure of the Prostaglandin E2 Receptor 4 Coupled to G Protein

EMDB-29946: 
Cryo-EM Structure of the Prostaglandin E2 Receptor 3 Coupled to G Protein

EMDB-29935: 
Cryo-EM Structure of the Prostaglandin E Receptor EP4 Coupled to G Protein

EMDB-29940: 
Cryo-EM Structure of the Prostaglandin E2 Receptor 4 Coupled to G Protein

EMDB-42124: 
Cryo-EM structure of human STEAP1 in complex with AMG 509 Fab

EMDB-29281: 
Cryo-EM structure of STING oligomer bound to cGAMP and NVS-STG2

EMDB-29282: 
Cryo-EM structure of STING oligomer bound to cGAMP, NVS-STG2 and C53
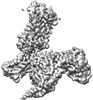
EMDB-34948: 
Cryo-EM structure of the apo-GPR132-Gi

EMDB-34950: 
Activation mechanism of GPR132 by NPGLY

EMDB-34951: 
Activation mechanism of GPR132 by 9(S)-HODE

EMDB-35044: 
Activation mechanism of GPR132 by compound NOX-6-7

EMDB-33883: 
Structural basis of human PRPS2 filaments

EMDB-33756: 
Structure of PfNT1(Y190A)-GFP in complex with GSK4

EMDB-33788: 
Structure of GluN1a-GluN2D NMDA receptor in complex with agonist glycine and competitive antagonist CPP.

EMDB-33792: 
Structure of GluN1a-GluN2D NMDA receptor in complex with agonists glycine and glutamate.

EMDB-33795: 
Structure of GluN1a E698C-GluN2D NMDA receptor in cystines crosslinked state.

EMDB-33798: 
Structure of GluN1a E698C-GluN2D NMDA receptor in cystines non-crosslinked state.

EMDB-33793: 
Structure of GluN1b-GluN2D NMDA receptor in complex with agonists glycine and glutamate.

EMDB-31532: 
Cryo-EM structure of the Potassium channel AKT1 mutant from Arabidopsis thaliana

EMDB-32769: 
Cryo-EM structure of the Potassium channel AKT1 from Arabidopsis thaliana

EMDB-33467: 
Cryo-EM structure of the AKT1-AtKC1 complex from Arabidopsis thaliana

EMDB-33305: 
E.coli phosphoribosylpyrophosphate (PRPP) synthetase type A filament bound with ADP, Pi and R5P

EMDB-33306: 
E.coli phosphoribosylpyrophosphate (PRPP) synthetase type A(AMP/ADP) filament bound with ADP, AMP and R5P

EMDB-33309: 
E.coli phosphoribosylpyrophosphate (PRPP) synthetase type B filament bound with Pi

EMDB-26522: 
SARS-CoV-2 6P Mut7 in complex with K398.25 Fab

EMDB-26523: 
SARS-CoV-1 in complex with K398.25 Fab
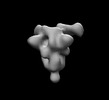
EMDB-26524: 
SARS-CoV-2 6P Mut7 in complex with K398.16 Fab
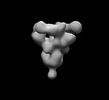
EMDB-26525: 
SARS-CoV-2 6P Mut7 in complex with K398.16 Fab (3 bound)

EMDB-26526: 
SARS-CoV-1 in complex with K398.16 Fab
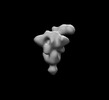
EMDB-26527: 
SARS-CoV-2 6P Mut7 in complex with K288.2 Fab

EMDB-26528: 
SARS-CoV-2 6P Mut7 in complex with K398.8 Fab

EMDB-26529: 
SARS-CoV-2 6P Mut7 in complex with K398.8 Fab (2 bound)

EMDB-26530: 
SARS-CoV-2 6P Mut7 in complex with K398.18 Fab
EMDB-26531: 
SARS-CoV-2 6P Mut7 in complex with K398.18 Fabs (2 bound)

EMDB-26532: 
SARS-CoV-2 6P Mut7 in complex with K398.22 Fab
EMDB-26533: 
SARS-CoV-2 6P Mut7 in complex with K398.22 Fab (2 bound)

EMDB-26534: 
SARS-CoV-1 in complex with K398.8 Fab

EMDB-26535: 
SARS-CoV-1 in complex with K398.8 Fab (2 bound)

EMDB-26536: 
SARS-CoV-1 in complex with K398.18 Fab (2 bound)

EMDB-26537: 
SARS-CoV-1 in complex with K398.18 Fab (3 bound)

EMDB-26538: 
SARS-CoV-1 in complex with K288.2 Fab
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
