+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structural basis of human PRPS2 filaments | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | phosphoribosylpyrophosphate synthetase /  complex / complex /  filament / filament /  TRANSFERASE (転移酵素) TRANSFERASE (転移酵素) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報AMP biosynthetic process / 5-Phosphoribose 1-diphosphate biosynthesis / ribose phosphate diphosphokinase complex /  リボースリン酸ジホスホキナーゼ / ribose phosphate diphosphokinase activity / 5-phosphoribose 1-diphosphate biosynthetic process / pentose-phosphate shunt / purine nucleotide biosynthetic process / nucleobase-containing compound metabolic process / AMP binding ...AMP biosynthetic process / 5-Phosphoribose 1-diphosphate biosynthesis / ribose phosphate diphosphokinase complex / リボースリン酸ジホスホキナーゼ / ribose phosphate diphosphokinase activity / 5-phosphoribose 1-diphosphate biosynthetic process / pentose-phosphate shunt / purine nucleotide biosynthetic process / nucleobase-containing compound metabolic process / AMP binding ...AMP biosynthetic process / 5-Phosphoribose 1-diphosphate biosynthesis / ribose phosphate diphosphokinase complex /  リボースリン酸ジホスホキナーゼ / ribose phosphate diphosphokinase activity / 5-phosphoribose 1-diphosphate biosynthetic process / pentose-phosphate shunt / purine nucleotide biosynthetic process / nucleobase-containing compound metabolic process / AMP binding / animal organ regeneration / リボースリン酸ジホスホキナーゼ / ribose phosphate diphosphokinase activity / 5-phosphoribose 1-diphosphate biosynthetic process / pentose-phosphate shunt / purine nucleotide biosynthetic process / nucleobase-containing compound metabolic process / AMP binding / animal organ regeneration /  ADP binding / GDP binding / ADP binding / GDP binding /  kinase activity / kinase activity /  carbohydrate binding / carbohydrate binding /  リン酸化 / magnesium ion binding / protein homodimerization activity / リン酸化 / magnesium ion binding / protein homodimerization activity /  ATP binding / identical protein binding / ATP binding / identical protein binding /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.08 Å クライオ電子顕微鏡法 / 解像度: 3.08 Å | |||||||||
 データ登録者 データ登録者 | Lu GM / Hu HH / Liu JL | |||||||||
| 資金援助 |  中国, 2件 中国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Cell Biosci / 年: 2023 ジャーナル: Cell Biosci / 年: 2023タイトル: Structural basis of human PRPS2 filaments. 著者: Guang-Ming Lu / Huan-Huan Hu / Chia-Chun Chang / Jiale Zhong / Xian Zhou / Chen-Jun Guo / Tianyi Zhang / Yi-Lan Li / Boqi Yin / Ji-Long Liu /   要旨: BACKGROUND: PRPP synthase (PRPS) transfers the pyrophosphate groups from ATP to ribose-5-phosphate to produce 5-phosphate ribose-1-pyrophosphate (PRPP), a key intermediate in the biosynthesis of ...BACKGROUND: PRPP synthase (PRPS) transfers the pyrophosphate groups from ATP to ribose-5-phosphate to produce 5-phosphate ribose-1-pyrophosphate (PRPP), a key intermediate in the biosynthesis of several metabolites including nucleotides, dinucleotides and some amino acids. There are three PRPS isoforms encoded in human genome. While human PRPS1 (hPRPS1) and human PRPS2 (hPRPS2) are expressed in most tissues, human PRPS3 (hPRPS3) is exclusively expressed in testis. Although hPRPS1 and hPRPS2 share 95% sequence identity, hPRPS2 has been shown to be less sensitive to allosteric inhibition and specifically upregulated in certain cancers in the translational level. Recent studies demonstrate that PRPS can form a subcellular compartment termed the cytoophidium in multiple organisms across prokaryotes and eukaryotes. Forming filaments and cytoophidia is considered as a distinctive mechanism involving the polymerization of the protein. Previously we solved the filament structures of Escherichia coli PRPS (ecPRPS) using cryo-electron microscopy (cryo-EM) . RESULTS: Order to investigate the function and molecular mechanism of hPRPS2 polymerization, here we solve the polymer structure of hPRPS2 at 3.08 Å resolution. hPRPS2 hexamers stack into polymers ...RESULTS: Order to investigate the function and molecular mechanism of hPRPS2 polymerization, here we solve the polymer structure of hPRPS2 at 3.08 Å resolution. hPRPS2 hexamers stack into polymers in the conditions with the allosteric/competitive inhibitor ADP. The binding modes of ADP at the canonical allosteric site and at the catalytic active site are clearly determined. A point mutation disrupting the inter-hexamer interaction prevents hPRPS2 polymerization and results in significantly reduced catalytic activity. CONCLUSION: Findings suggest that the regulation of hPRPS2 polymer is distinct from ecPRPS polymer and provide new insights to the regulation of hPRPS2 with structural basis. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_33883.map.gz emd_33883.map.gz | 59.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-33883-v30.xml emd-33883-v30.xml emd-33883.xml emd-33883.xml | 17 KB 17 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_33883_fsc.xml emd_33883_fsc.xml | 9.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_33883.png emd_33883.png | 63.1 KB | ||
| マスクデータ |  emd_33883_msk_1.map emd_33883_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| その他 |  emd_33883_half_map_1.map.gz emd_33883_half_map_1.map.gz emd_33883_half_map_2.map.gz emd_33883_half_map_2.map.gz | 49.6 MB 49.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-33883 http://ftp.pdbj.org/pub/emdb/structures/EMD-33883 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33883 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33883 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7yk1MC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_33883.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_33883.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.06 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_33883_msk_1.map emd_33883_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_33883_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_33883_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Filament structure of human phosphoribosylpyrophosphate synthetase2.
| 全体 | 名称: Filament structure of human phosphoribosylpyrophosphate synthetase2. |
|---|---|
| 要素 |
|
-超分子 #1: Filament structure of human phosphoribosylpyrophosphate synthetase2.
| 超分子 | 名称: Filament structure of human phosphoribosylpyrophosphate synthetase2. タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Ribose-phosphate pyrophosphokinase 2
| 分子 | 名称: Ribose-phosphate pyrophosphokinase 2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO / EC番号:  リボースリン酸ジホスホキナーゼ リボースリン酸ジホスホキナーゼ |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 35.640969 KDa |
| 組換発現 | 生物種:   Escherichia coli 'BL21-Gold(DE3)pLysS AG' (大腸菌) Escherichia coli 'BL21-Gold(DE3)pLysS AG' (大腸菌) |
| 配列 | 文字列: MPNIVLFSGS SHQDLSQRVA DRLGLELGKV VTKKFSNQET SVEIGESVRG EDVYIIQSGC GEINDNLMEL LIMINACKIA SSSRVTAVI PCFPYARQDK KDKSRAPISA KLVANMLSVA GADHIITMDL HASQIQGFFD IPVDNLYAEP AVLQWIRENI A EWKNCIIV ...文字列: MPNIVLFSGS SHQDLSQRVA DRLGLELGKV VTKKFSNQET SVEIGESVRG EDVYIIQSGC GEINDNLMEL LIMINACKIA SSSRVTAVI PCFPYARQDK KDKSRAPISA KLVANMLSVA GADHIITMDL HASQIQGFFD IPVDNLYAEP AVLQWIRENI A EWKNCIIV SPDAGGAKRV TSIADRLNVE FALIHKERKK ANEVDRMVLV GDVKDRVAIL VDDMADTCGT ICHAADKLLS AG ATKVYAI LTHGIFSGPA ISRINNAAFE AVVVTNTIPQ EDKMKHCTKI QVIDISMILA EAIRRTHNGE SVSYLFSHVP LHH HHHH UniProtKB: Ribose-phosphate pyrophosphokinase 2 |
-分子 #2: PHOSPHATE ION
| 分子 | 名称: PHOSPHATE ION / タイプ: ligand / ID: 2 / コピー数: 6 / 式: PO4 |
|---|---|
| 分子量 | 理論値: 94.971 Da |
| Chemical component information | 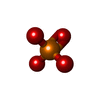 ChemComp-PO4: |
-分子 #3: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 3 / コピー数: 6 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #4: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 4 / コピー数: 12 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information | 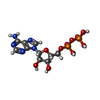 ChemComp-ADP: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | filament |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 構成要素:
| |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: UltrAuFoil R1.2/1.3 / 材質: GOLD / 支持フィルム - 材質: GOLD | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 22500 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 22500 |
| 試料ステージ | ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 2403 / 平均露光時間: 4.0 sec. / 平均電子線量: 60.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X


























