-検索条件
-検索結果
検索 (著者・登録者: huang & cy)の結果177件中、1から50件目までを表示しています

EMDB-36800: 
Potassium transporter KtrAB from Bacillus subtilis in ADP-bound state
手法: 単粒子 / : Chang YK, Chiang WT, Hu NJ, Tsai MD

EMDB-36801: 
Potassium transporter KtrAB from Bacillus subtilis in ADP-bound state, focused refined on KtrA octamer
手法: 単粒子 / : Chang YK, Chiang WT, Hu NJ, Tsai MD

EMDB-36802: 
Potassium transporter KtrAB from Bacillus subtilis in ADP-bound state, focused refined on KtrB dimer
手法: 単粒子 / : Chang YK, Chiang WT, Hu NJ, Tsai MD

EMDB-36803: 
Potassium transporter KtrAB from Bacillus subtilis in ATP-bound state with addition of MgCl2
手法: 単粒子 / : Chang YK, Chiang WT, Hu NJ, Tsai MD

EMDB-36804: 
Potassium transporter KtrAB from Bacillus subtilis in ATP-bound state with addition of EDTA and EGTA
手法: 単粒子 / : Chang YK, Chiang WT, Hu NJ, Tsai MD

EMDB-38477: 
Potassium transporter KtrAB from Bacillus subtilis in ATP-bound state with addition of EDTA and EGTA, vertical C2 symmetry axis
手法: 単粒子 / : Chang YK, Chiang WT, Hu NJ, Tsai MD

EMDB-38478: 
Potassium transporter KtrAB from Bacillus subtilis in ATP-bound state with addition of EDTA and EGTA, C1 symmetry
手法: 単粒子 / : Chang YK, Chiang WT, Hu NJ, Tsai MD

EMDB-41374: 
Antibody N3-1 bound to RBDs in the up and down conformations
手法: 単粒子 / : Hsieh CL, McLellan JS
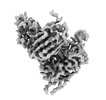
EMDB-41382: 
Antibody N3-1 bound to RBD in the up conformation
手法: 単粒子 / : Hsieh CL, McLellan JS
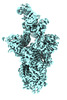
EMDB-41399: 
Antibody N3-1 bound to SARS-CoV-2 spike
手法: 単粒子 / : Hsieh CL, McLellan JS

PDB-8tm1: 
Antibody N3-1 bound to RBDs in the up and down conformations
手法: 単粒子 / : Hsieh CL, McLellan JS

PDB-8tma: 
Antibody N3-1 bound to RBD in the up conformation
手法: 単粒子 / : Hsieh CL, McLellan JS

EMDB-36229: 
CryoEM structure of Gi-coupled MRGPRX1 with peptide agonist CNF-Tx2
手法: 単粒子 / : Sun JP, Xu HE, Yang F, Liu ZM, Guo LL, Zhang YM, Fang GX, Tie L, Zhuang YM, Xue CY

EMDB-36232: 
CryoEM structure of Gq-coupled MRGPRX1 with peptide agonist BAM8-22
手法: 単粒子 / : Sun JP, Xu HE, Yang F, Liu ZM, Guo LL, Zhang YM, Fang GX, Tie L, Zhuang YM, Xue CY

EMDB-36233: 
CryoEM structure of Gi-coupled MRGPRX1 with peptide agonist BAM8-22
手法: 単粒子 / : Sun JP, Xu HE, Yang F, Liu ZM, Guo LL, Zhang YM, Fang GX, Tie L, Zhuang YM, Xue CY

EMDB-35384: 
Cryo-EM structure of ATP13A2 in the E1-ATP state
手法: 単粒子 / : Liu ZM, Mu JQ, Xue CY
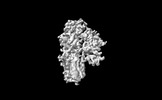
EMDB-35385: 
Cryo-EM structure of ATP13A2 in the E1-like state
手法: 単粒子 / : Liu ZM, Mu JQ, Xue CY

EMDB-35386: 
Cryo-EM structure of ATP13A2 in the E2P state
手法: 単粒子 / : Liu ZM, Mu JQ, Xue CY

EMDB-35387: 
Cryo-EM structure of ATP13A2 in the E2-Pi state
手法: 単粒子 / : Liu ZM, Mu JQ, Xue CY

EMDB-35388: 
Cryo-EM structure of ATP13A2 in the nominal E1P state
手法: 単粒子 / : Liu ZM, Mu JQ, Xue CY
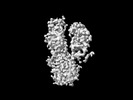
EMDB-35391: 
Cryo-EM structure of ATP13A2 in the putative of E2 state
手法: 単粒子 / : Liu ZM, Mu JQ, Xue CY
EMDB-35392: 
Cryo-EM structure of ATP13A2 in the E1P-ADP state
手法: 単粒子 / : Liu ZM, Mu JQ, Xue CY

EMDB-36661: 
Cryo-EM structure of SIDT1 in complex with phosphatidic acid
手法: 単粒子 / : Sun CR, Xu D, Li Q, Zhou CZ, Chen Y

EMDB-37237: 
Cryo-EM structure of the GPR174-Gs complex bound to endogenous lysoPS
手法: 単粒子 / : Nie Y, Qiu Z, Zheng S, Chen S

PDB-8kh5: 
Cryo-EM structure of the GPR174-Gs complex bound to endogenous lysoPS
手法: 単粒子 / : Nie Y, Qiu Z, Zheng S, Chen S

EMDB-37224: 
Cryo-EM structure of the GPR61-Gs complex
手法: 単粒子 / : Nie Y, Qiu Z, Zheng S

EMDB-37236: 
Cryo-EM structure of the GPR161-Gs complex
手法: 単粒子 / : Nie Y, Qiu Z, Zheng S, Chen S
EMDB-28617: 
Cryo-EM structure of HIV-1 BG505 DS-SOSIP ENV trimer bound to VRC34.01 FAB
手法: 単粒子 / : Pletnev S, Kwong P

EMDB-28618: 
Cryo-EM structure of HIV-1 BG505 DS-SOSIP ENV trimer bound to VRC34.01-COMBO1 FAB
手法: 単粒子 / : Pletnev S, Kwong P

EMDB-28619: 
Cryo-EM structure of HIV-1 BG505 DS-SOSIP ENV trimer bound to VRC34.01-MM28 FAB
手法: 単粒子 / : Pletnev S, Kwong P

PDB-8euu: 
Cryo-EM structure of HIV-1 BG505 DS-SOSIP ENV trimer bound to VRC34.01 FAB
手法: 単粒子 / : Pletnev S, Kwong P

PDB-8euv: 
Cryo-EM structure of HIV-1 BG505 DS-SOSIP ENV trimer bound to VRC34.01-COMBO1 FAB
手法: 単粒子 / : Pletnev S, Kwong P

PDB-8euw: 
Cryo-EM structure of HIV-1 BG505 DS-SOSIP ENV trimer bound to VRC34.01-MM28 FAB
手法: 単粒子 / : Pletnev S, Kwong P

EMDB-28036: 
Cryo-EM structure of the full-length human NF1 dimer
手法: 単粒子 / : Darling JE, Merk A, Grisshammer R, Ognjenovic J

PDB-8edm: 
Cryo-EM structure of the full-length human NF1 dimer
手法: 単粒子 / : Darling JE, Merk A, Grisshammer R, Ognjenovic J

EMDB-15786: 
Cryo-EM structure of apolipoprotein N-acyltransferase Lnt from E. coli (Apo form)
手法: 単粒子 / : Degtjarik O, Smithers L, Boland C, Caffrey M, Shalev Benami M

EMDB-15787: 
Cryo-EM structure of apolipoprotein N-acyltransferase Lnt from E. coli in complex with PE
手法: 単粒子 / : Degtjarik O, Smithers L, Boland C, Caffrey M, Shalev Benami M

EMDB-15788: 
Cryo-EM structure of apolipoprotein N-acyltransferase Lnt from E. coli in complex with PE (C387S mutant)
手法: 単粒子 / : Degtjarik O, Smithers L, Boland C, Caffrey M, Shalev Benami M

EMDB-15789: 
Cryo-EM structure of apolipoprotein N-acyltransferase Lnt from E. coli in complex with Lyso-PE
手法: 単粒子 / : Degtjarik O, Smithers L, Boland C, Caffrey M, Shalev Benami M

EMDB-15790: 
Cryo-EM structure apolipoprotein N-acyltransferase Lnt from E.coli in complex with FP3
手法: 単粒子 / : Degtjarik O, Smithers L, Boland C, Caffrey M, Shalev Benami M

EMDB-15791: 
Cryo-EM structure of apolipoprotein N-acyltransferase Lnt from E. coli in complex with Pam3
手法: 単粒子 / : Degtjarik O, Smithers L, Boland C, Caffrey M, Shalev Benami M

EMDB-33845: 
Cryo-EM map of Rpd3S complex
手法: 単粒子 / : Li HT, Yan CY, Guan HP, Wang P

EMDB-33846: 
Cryo-EM map of Rpd3S:head-bridge-right arm
手法: 単粒子 / : Li HT, Yan CY, Guan HP, Wang P

EMDB-33847: 
Cryo-EM map of Rpd3S:bridge-left arm
手法: 単粒子 / : Li HT, Yan CY, Guan HP, Wang P

EMDB-33848: 
Cryo-EM map of Eaf3 CHD bound to H3K36me3 nucleosome
手法: 単粒子 / : Li HT, Yan CY, Guan HP, Wang P

EMDB-33849: 
Cryo-EM map of Rpd3S in loose-state Rpd3S-NCP complex
手法: 単粒子 / : Li HT, Yan CY, Guan HP, Wang P

EMDB-33850: 
Cryo-EM map of Rpd3S in close-state Rpd3S-NCP complex
手法: 単粒子 / : Li HT, Yan CY, Guan HP, Wang P
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について






 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
