+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of Walker B mutated MCM8/9 heterohexamer complex with ADP | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Helicase DNA Replication DNA repair /  DNA BINDING PROTEIN (DNA結合タンパク質) DNA BINDING PROTEIN (DNA結合タンパク質) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報MutLbeta complex binding / MutSbeta complex binding / recombinational interstrand cross-link repair / MCM8-MCM9 complex / male gamete generation / mismatch repair involved in maintenance of fidelity involved in DNA-dependent DNA replication / CDC6 association with the ORC:origin complex / MutSalpha complex binding / E2F-enabled inhibition of pre-replication complex formation / female gamete generation ...MutLbeta complex binding / MutSbeta complex binding / recombinational interstrand cross-link repair / MCM8-MCM9 complex / male gamete generation / mismatch repair involved in maintenance of fidelity involved in DNA-dependent DNA replication / CDC6 association with the ORC:origin complex / MutSalpha complex binding / E2F-enabled inhibition of pre-replication complex formation / female gamete generation / Unwinding of DNA / MCM complex / DNA duplex unwinding / Activation of the pre-replicative complex / Activation of ATR in response to replication stress / protein localization to chromatin /  DNA helicase activity / DNA helicase activity /  helicase activity / double-strand break repair via homologous recombination / Orc1 removal from chromatin / helicase activity / double-strand break repair via homologous recombination / Orc1 removal from chromatin /  single-stranded DNA binding / single-stranded DNA binding /  染色体 / 染色体 /  ヘリカーゼ / protein stabilization / ヘリカーゼ / protein stabilization /  細胞周期 / DNA damage response / 細胞周期 / DNA damage response /  chromatin binding / protein-containing complex binding / chromatin binding / protein-containing complex binding /  enzyme binding / enzyme binding /  ATP hydrolysis activity / ATP hydrolysis activity /  核質 / 核質 /  ATP binding / ATP binding /  細胞核 細胞核類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.3 Å クライオ電子顕微鏡法 / 解像度: 4.3 Å | |||||||||
 データ登録者 データ登録者 | Li C / Gao Y | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nucleic Acids Res / 年: 2023 ジャーナル: Nucleic Acids Res / 年: 2023タイトル: Activity, substrate preference and structure of the HsMCM8/9 helicase. 著者: David R McKinzey / Chuxuan Li / Yang Gao / Michael A Trakselis /  要旨: The minichromosomal maintenance proteins, MCM8 and MCM9, are more recent evolutionary additions to the MCM family, only cooccurring in selected higher eukaryotes. Mutations in these genes are ...The minichromosomal maintenance proteins, MCM8 and MCM9, are more recent evolutionary additions to the MCM family, only cooccurring in selected higher eukaryotes. Mutations in these genes are directly linked to ovarian insufficiency, infertility, and several cancers. MCM8/9 appears to have ancillary roles in fork progression and recombination of broken replication forks. However, the biochemical activity, specificities and structures have not been adequately illustrated, making mechanistic determination difficult. Here, we show that human MCM8/9 (HsMCM8/9) is an ATP dependent DNA helicase that unwinds fork DNA substrates with a 3'-5' polarity. High affinity ssDNA binding occurs in the presence of nucleoside triphosphates, while ATP hydrolysis weakens the interaction with DNA. The cryo-EM structure of the HsMCM8/9 heterohexamer was solved at 4.3 Å revealing a trimer of heterodimer configuration with two types of interfacial AAA+ nucleotide binding sites that become more organized upon binding ADP. Local refinements of the N or C-terminal domains (NTD or CTD) improved the resolution to 3.9 or 4.1 Å, respectively, and shows a large displacement in the CTD. Changes in AAA+ CTD upon nucleotide binding and a large swing between the NTD and CTD likely implies that MCM8/9 utilizes a sequential subunit translocation mechanism for DNA unwinding. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
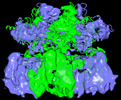
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_40234.map.gz emd_40234.map.gz | 117.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-40234-v30.xml emd-40234-v30.xml emd-40234.xml emd-40234.xml | 17 KB 17 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_40234.png emd_40234.png | 183 KB | ||
| その他 |  emd_40234_half_map_1.map.gz emd_40234_half_map_1.map.gz emd_40234_half_map_2.map.gz emd_40234_half_map_2.map.gz | 115.8 MB 115.8 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-40234 http://ftp.pdbj.org/pub/emdb/structures/EMD-40234 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40234 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-40234 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8s91MC  8s92C  8s94C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_40234.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_40234.map.gz / 形式: CCP4 / 大きさ: 125 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.11 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_40234_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_40234_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Structure of Walker B mutated MCM8/9 heterohexamer complex with ADP
| 全体 | 名称: Structure of Walker B mutated MCM8/9 heterohexamer complex with ADP |
|---|---|
| 要素 |
|
-超分子 #1: Structure of Walker B mutated MCM8/9 heterohexamer complex with ADP
| 超分子 | 名称: Structure of Walker B mutated MCM8/9 heterohexamer complex with ADP タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 500 KDa |
-分子 #1: DNA helicase MCM8
| 分子 | 名称: DNA helicase MCM8 / タイプ: protein_or_peptide / ID: 1 / コピー数: 3 / 光学異性体: LEVO / EC番号:  ヘリカーゼ ヘリカーゼ |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 93.817703 KDa |
| 組換発現 | 生物種:   Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MNGEYRGRGF GRGRFQSWKR GRGGGNFSGK WREREHRPDL SKTTGKRTSE QTPQFLLSTK TPQSMQSTLD RFIPYKGWKL YFSEVYSDS SPLIEKIQAF EKFFTRHIDL YDKDEIERKG SILVDFKELT EGGEVTNLIP DIATELRDAP EKTLACMGLA I HQVLTKDL ...文字列: MNGEYRGRGF GRGRFQSWKR GRGGGNFSGK WREREHRPDL SKTTGKRTSE QTPQFLLSTK TPQSMQSTLD RFIPYKGWKL YFSEVYSDS SPLIEKIQAF EKFFTRHIDL YDKDEIERKG SILVDFKELT EGGEVTNLIP DIATELRDAP EKTLACMGLA I HQVLTKDL ERHAAELQAQ EGLSNDGETM VNVPHIHARV YNYEPLTQLK NVRANYYGKY IALRGTVVRV SNIKPLCTKM AF LCAACGE IQSFPLPDGK YSLPTKCPVP VCRGRSFTAL RSSPLTVTMD WQSIKIQELM SDDQREAGRI PRTIECELVH DLV DSCVPG DTVTITGIVK VSNAEEGSRN KNDKCMFLLY IEANSISNSK GQKTKSSEDG CKHGMLMEFS LKDLYAIQEI QAEE NLFKL IVNSLCPVIF GHELVKAGLA LALFGGSQKY ADDKNRIPIR GDPHILVVGD PGLGKSQMLQ AACNVAPRGV YVCGN TTTT SGLTVTLSKD SSSGDFALEA GALVLGDQGI CGIDQFDKMG NQHQALLEAM EQQSISLAKA GVVCSLPART SIIAAA NPV GGHYNKAKTV SENLKMGSAL LSRFDLVFIL LDTPNEHHDH LLSEHVIAIR AGKQRTISSA TVARMNSQDS NTSVLEV VS EKPLSERLKV VPGETIDPIP HQLLRKYIGY ARQYVYPRLS TEAARVLQDF YLELRKQSQR LNSSPITTRQ LESLIRLT E ARARLELREE ATKEDAEDIV EIMKYSMLGT YSDEFGNLDF ERSQHGSGMS NRSTAKRFIS ALNNVAERTY NNIFQFHQL RQIAKELNIQ VADFENFIGS LNDQGYLLKK GPKVYQLQTM UniProtKB: DNA helicase MCM8 |
-分子 #2: DNA helicase MCM9
| 分子 | 名称: DNA helicase MCM9 / タイプ: protein_or_peptide / ID: 2 / コピー数: 3 / 光学異性体: LEVO / EC番号:  ヘリカーゼ ヘリカーゼ |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 127.485594 KDa |
| 組換発現 | 生物種:   Trichoplusia ni (イラクサキンウワバ) Trichoplusia ni (イラクサキンウワバ) |
| 配列 | 文字列: MNSDQVTLVG QVFESYVSEY HKNDILLILK ERDEDAHYPV VVNAMTLFET NMEIGEYFNM FPSEVLTIFD SALRRSALTI LQSLSQPEA VSMKQNLHAR ISGLPVCPEL VREHIPKTKD VGHFLSVTGT VIRTSLVKVL EFERDYMCNK CKHVFVIKAD F EQYYTFCR ...文字列: MNSDQVTLVG QVFESYVSEY HKNDILLILK ERDEDAHYPV VVNAMTLFET NMEIGEYFNM FPSEVLTIFD SALRRSALTI LQSLSQPEA VSMKQNLHAR ISGLPVCPEL VREHIPKTKD VGHFLSVTGT VIRTSLVKVL EFERDYMCNK CKHVFVIKAD F EQYYTFCR PSSCPSLESC DSSKFTCLSG LSSSPTRCRD YQEIKIQEQV QRLSVGSIPR SMKVILEDDL VDSCKSGDDL TI YGIVMQR WKPFQQDVRC EVEIVLKANY IQVNNEQSSG IIMDEEVQKE FEDFWEYYKS DPFAGRNVIL ASLCPQVFGM YLV KLAVAM VLAGGIQRTD ATGTRVRGES HLLLVGDPGT GKSQFLKYAA KITPRSVLTT GIGSTSAGLT VTAVKDSGEW NLEA GALVL ADAGLCCIDQ FNSLKEHDRT SIHEAMEQQT ISVAKAGLVC KLNTRTTILA ATNPKGQYDP QESVSVNIAL GSPLL SRFD LILVLLDTKN EDWDRIISSF ILENKGYPSK SEKLWSMEKM KTYFCLIRNL QPTLSDVGNQ VLLRYYQMQR QSDCRN AAR TTIRLLESLI RLAEAHARLM FRDTVTLEDA ITVVSVMESS MQGGALLGGV NALHTSFPEN PGEQYQRQCE LILEKLE LQ SLLSEELRRL ERLQNQSVHQ SQPRVLEVET TPGSLRNGPG EESNFRTSSQ QEINYSTHIF SPGGSPEGSP VLDPPPHL E PNRSTSRKHS AQHKNNRDDS LDWFDFMATH QSEPKNTVVV SPHPKTSGEN MASKISNSTS QGKEKSEPGQ RSKVDIGLL PSPGETGVPW RADNVESNKK KRLALDSEAA VSADKPDSVL THHVPRNLQK LCKERAQKLC RNSTRVPAQC TVPSHPQSTP VHSPDRMLD SPKRKRPKSL AQVEEPAIEN VKPPGSPVAK LAKFTFKQKS KLIHSFEDHS HVSPGATKIA VHSPKISQRR T RRDAALPV KRPGKLTSTP GNQISSQPQG ETKEVSQQPP EKHGPREKVM CAPEKRIIQP ELELGNETGC AHLTCEGDKK EE VSGSNKS GKVHACTLAR LANFCFTPPS ESKSKSPPPE RKNRGERGPS SPPTTTAPMR VSKRKSFQLR GSTEKLIVSK ESL FTLPEL GDEAFDCDWD EEMRKKS UniProtKB: DNA helicase MCM9 |
-分子 #3: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 3 / コピー数: 6 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information | 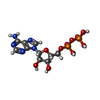 ChemComp-ADP: |
-分子 #4: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 4 / コピー数: 6 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.5 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 298 K / 装置: FEI VITROBOT MARK I |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 3.0 µm Bright-field microscopy / 最大 デフォーカス(公称値): 3.0 µm最小 デフォーカス(公称値): 0.7000000000000001 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 平均電子線量: 49.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 4.3 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 227818 |
 ムービー
ムービー コントローラー
コントローラー









 Z
Z Y
Y X
X