+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of a bacterial gasdermin large oval pore assembly | |||||||||
 マップデータ マップデータ | ~50mer bacterial gasdermin oval map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | gasdermin /  pore-forming protein (膜孔形成毒素) / pore-forming protein (膜孔形成毒素) /  pyroptosis / pyroptosis /  bacteria (細菌) / bacteria (細菌) /  immunity / immunity /  cell death (細胞死) / cell death (細胞死) /  IMMUNE SYSTEM (免疫系) IMMUNE SYSTEM (免疫系) | |||||||||
| 機能・相同性 | defense response to virus /  細胞膜 / 細胞膜 /  細胞質 / Gasdermin bGSDM 細胞質 / Gasdermin bGSDM 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |  Vitiosangium sp. GDMCC 1.1324 (バクテリア) Vitiosangium sp. GDMCC 1.1324 (バクテリア) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 6.1 Å クライオ電子顕微鏡法 / 解像度: 6.1 Å | |||||||||
 データ登録者 データ登録者 | Johnson AG / Mayer ML / Kranzusch PJ | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2024 ジャーナル: Nature / 年: 2024タイトル: Structure and assembly of a bacterial gasdermin pore. 著者: Alex G Johnson / Megan L Mayer / Stefan L Schaefer / Nora K McNamara-Bordewick / Gerhard Hummer / Philip J Kranzusch /   要旨: In response to pathogen infection, gasdermin (GSDM) proteins form membrane pores that induce a host cell death process called pyroptosis. Studies of human and mouse GSDM pores have revealed the ...In response to pathogen infection, gasdermin (GSDM) proteins form membrane pores that induce a host cell death process called pyroptosis. Studies of human and mouse GSDM pores have revealed the functions and architectures of assemblies comprising 24 to 33 protomers, but the mechanism and evolutionary origin of membrane targeting and GSDM pore formation remain unknown. Here we determine a structure of a bacterial GSDM (bGSDM) pore and define a conserved mechanism of pore assembly. Engineering a panel of bGSDMs for site-specific proteolytic activation, we demonstrate that diverse bGSDMs form distinct pore sizes that range from smaller mammalian-like assemblies to exceptionally large pores containing more than 50 protomers. We determine a cryo-electron microscopy structure of a Vitiosangium bGSDM in an active 'slinky'-like oligomeric conformation and analyse bGSDM pores in a native lipid environment to create an atomic-level model of a full 52-mer bGSDM pore. Combining our structural analysis with molecular dynamics simulations and cellular assays, our results support a stepwise model of GSDM pore assembly and suggest that a covalently bound palmitoyl can leave a hydrophobic sheath and insert into the membrane before formation of the membrane-spanning β-strand regions. These results reveal the diversity of GSDM pores found in nature and explain the function of an ancient post-translational modification in enabling programmed host cell death. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_43510.map.gz emd_43510.map.gz | 75.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-43510-v30.xml emd-43510-v30.xml emd-43510.xml emd-43510.xml | 15.1 KB 15.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_43510_fsc.xml emd_43510_fsc.xml | 11.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_43510.png emd_43510.png | 54.2 KB | ||
| マスクデータ |  emd_43510_msk_1.map emd_43510_msk_1.map | 149.9 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-43510.cif.gz emd-43510.cif.gz | 5 KB | ||
| その他 |  emd_43510_half_map_1.map.gz emd_43510_half_map_1.map.gz emd_43510_half_map_2.map.gz emd_43510_half_map_2.map.gz | 139.3 MB 139.3 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-43510 http://ftp.pdbj.org/pub/emdb/structures/EMD-43510 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-43510 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-43510 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_43510.map.gz / 形式: CCP4 / 大きさ: 149.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_43510.map.gz / 形式: CCP4 / 大きさ: 149.9 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | ~50mer bacterial gasdermin oval map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 2.66 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_43510_msk_1.map emd_43510_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
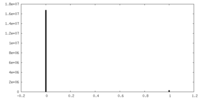
| 投影像・断面図 |
| ||||||||||||
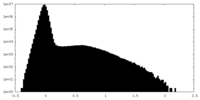
| 密度ヒストグラム |
-ハーフマップ: ~50mer bacterial gasdermin oval half map
| ファイル | emd_43510_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | ~50mer bacterial gasdermin oval half map | ||||||||||||
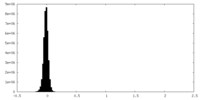
| 投影像・断面図 |
| ||||||||||||
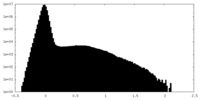
| 密度ヒストグラム |
-ハーフマップ: ~50mer bacterial gasdermin oval half map
| ファイル | emd_43510_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | ~50mer bacterial gasdermin oval half map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Vitiosangium bGSDM in a large oval pore assembly
| 全体 | 名称: Vitiosangium bGSDM in a large oval pore assembly |
|---|---|
| 要素 |
|
-超分子 #1: Vitiosangium bGSDM in a large oval pore assembly
| 超分子 | 名称: Vitiosangium bGSDM in a large oval pore assembly / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:  Vitiosangium sp. GDMCC 1.1324 (バクテリア) Vitiosangium sp. GDMCC 1.1324 (バクテリア) |
-分子 #1: Vitiosangium bacterial gasdermin
| 分子 | 名称: Vitiosangium bacterial gasdermin / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:  Vitiosangium sp. GDMCC 1.1324 (バクテリア) Vitiosangium sp. GDMCC 1.1324 (バクテリア) |
| 組換発現 | 生物種:   Escherichia coli 'BL21-Gold(DE3)pLysS AG' (大腸菌) Escherichia coli 'BL21-Gold(DE3)pLysS AG' (大腸菌) |
| 配列 | 文字列: SGLCSDPAIT YLKRLGYNVV RLPREGIQPL HLLGQQRGTV EYLGSLEKLI TQPPSEPPAI TRDQAAAGIN GQKTENLSFS IGINILKSVL AQFGAGAGIE AQYNQARKVR FEFSNVLADS VEPLAVGQFL KMAEVDADNP VLKQYVLGNG RLYVITQVIK SNEFTVAAEK ...文字列: SGLCSDPAIT YLKRLGYNVV RLPREGIQPL HLLGQQRGTV EYLGSLEKLI TQPPSEPPAI TRDQAAAGIN GQKTENLSFS IGINILKSVL AQFGAGAGIE AQYNQARKVR FEFSNVLADS VEPLAVGQFL KMAEVDADNP VLKQYVLGNG RLYVITQVIK SNEFTVAAEK SGGGSIQLDV PEIQKVVGGK LKVEASVSSQ STVTYKGEKQ LVFGFKCFEI GVKNGEITLF ASQLVPR UniProtKB: Gasdermin bGSDM |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
詳細: 150 mM NaCl, 20 mM HEPES-HOH (pH 7.5), 50 mM HECAMEG | ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV | ||||||||||||
| 詳細 | The sample was monodisperse |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy最大 デフォーカス(公称値): 2.8000000000000003 µm 最小 デフォーカス(公称値): 1.0 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 実像数: 33411 / 平均電子線量: 53.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー








 Z
Z Y
Y X
X