+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of photosynthetic LH1-RC super-complex of Rhodobacter capsulatus | |||||||||||||||||||||
 マップデータ マップデータ | negative B-factor applied map | |||||||||||||||||||||
 試料 試料 |
| |||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報organelle inner membrane / plasma membrane-derived chromatophore membrane / plasma membrane light-harvesting complex /  bacteriochlorophyll binding / photosynthetic electron transport in photosystem II / bacteriochlorophyll binding / photosynthetic electron transport in photosystem II /  photosynthesis, light reaction / electron transporter, transferring electrons within the cyclic electron transport pathway of photosynthesis activity / photosynthesis, light reaction / electron transporter, transferring electrons within the cyclic electron transport pathway of photosynthesis activity /  生体膜 / 生体膜 /  metal ion binding / metal ion binding /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||||||||||||||
| 生物種 |   Rhodobacter capsulatus (バクテリア) Rhodobacter capsulatus (バクテリア) | |||||||||||||||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.6 Å クライオ電子顕微鏡法 / 解像度: 2.6 Å | |||||||||||||||||||||
 データ登録者 データ登録者 | Tani K / Kanno R / Ji X-C / Satoh I / Kobayashi Y / Nagashima KVP / Hall M / Yu L-J / Kimura Y / Mizoguchi A ...Tani K / Kanno R / Ji X-C / Satoh I / Kobayashi Y / Nagashima KVP / Hall M / Yu L-J / Kimura Y / Mizoguchi A / Humbel BM / Madigan MT / Wang-Otomo Z-Y | |||||||||||||||||||||
| 資金援助 |  日本, 6件 日本, 6件
| |||||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Rhodobacter capsulatus forms a compact crescent-shaped LH1-RC photocomplex. 著者: Kazutoshi Tani / Ryo Kanno / Xuan-Cheng Ji / Itsusei Satoh / Yuki Kobayashi / Malgorzata Hall / Long-Jiang Yu / Yukihiro Kimura / Akira Mizoguchi / Bruno M Humbel / Michael T Madigan / Zheng-Yu Wang-Otomo /    要旨: Rhodobacter (Rba.) capsulatus has been a favored model for studies of all aspects of bacterial photosynthesis. This purple phototroph contains PufX, a polypeptide crucial for dimerization of the ...Rhodobacter (Rba.) capsulatus has been a favored model for studies of all aspects of bacterial photosynthesis. This purple phototroph contains PufX, a polypeptide crucial for dimerization of the light-harvesting 1-reaction center (LH1-RC) complex, but lacks protein-U, a U-shaped polypeptide in the LH1-RC of its close relative Rba. sphaeroides. Here we present a cryo-EM structure of the Rba. capsulatus LH1-RC purified by DEAE chromatography. The crescent-shaped LH1-RC exhibits a compact structure containing only 10 LH1 αβ-subunits. Four αβ-subunits corresponding to those adjacent to protein-U in Rba. sphaeroides were absent. PufX in Rba. capsulatus exhibits a unique conformation in its N-terminus that self-associates with amino acids in its own transmembrane domain and interacts with nearby polypeptides, preventing it from interacting with proteins in other complexes and forming dimeric structures. These features are discussed in relation to the minimal requirements for the formation of LH1-RC monomers and dimers, the spectroscopic behavior of both the LH1 and RC, and the bioenergetics of energy transfer from LH1 to the RC. | |||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_33931.map.gz emd_33931.map.gz | 228.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-33931-v30.xml emd-33931-v30.xml emd-33931.xml emd-33931.xml | 25.5 KB 25.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_33931_fsc.xml emd_33931_fsc.xml | 14.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_33931.png emd_33931.png | 149.3 KB | ||
| その他 |  emd_33931_half_map_1.map.gz emd_33931_half_map_1.map.gz emd_33931_half_map_2.map.gz emd_33931_half_map_2.map.gz | 194.6 MB 194.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-33931 http://ftp.pdbj.org/pub/emdb/structures/EMD-33931 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33931 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33931 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7ymlMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_33931.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_33931.map.gz / 形式: CCP4 / 大きさ: 244.1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | negative B-factor applied map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.82 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half odd map
| ファイル | emd_33931_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half odd map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
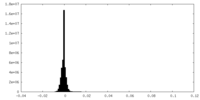
| 密度ヒストグラム |
-ハーフマップ: Half even map
| ファイル | emd_33931_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half even map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
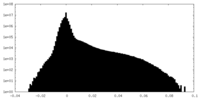
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
+全体 : Photosynthetic LH1-RC complex from the purple phototrophic bacter...
+超分子 #1: Photosynthetic LH1-RC complex from the purple phototrophic bacter...
+分子 #1: Reaction center protein L chain
+分子 #2: Reaction center protein M chain
+分子 #3: Photosynthetic reaction center H subunit
+分子 #4: Light-harvesting protein B-870 alpha chain
+分子 #5: Light-harvesting protein B-870 beta chain
+分子 #6: Photosynthetic reaction center PufX protein
+分子 #7: BACTERIOCHLOROPHYLL A
+分子 #8: BACTERIOPHEOPHYTIN A
+分子 #9: UBIQUINONE-10
+分子 #10: (1R)-2-{[{[(2S)-2,3-DIHYDROXYPROPYL]OXY}(HYDROXY)PHOSPHORYL]OXY}-...
+分子 #11: 1,2-dioleoyl-sn-glycero-3-phosphoethanolamine
+分子 #12: LAURYL DIMETHYLAMINE-N-OXIDE
+分子 #13: DODECYL-BETA-D-MALTOSIDE
+分子 #14: FE (III) ION
+分子 #15: SPHEROIDENE
+分子 #16: MYRISTIC ACID
+分子 #17: water
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5.0 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 80 % / チャンバー内温度: 277 K / 装置: LEICA EM GP |
| 詳細 | This sample was monodisperse. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy Bright-field microscopy最大 デフォーカス(公称値): 2.8000000000000003 µm 最小 デフォーカス(公称値): 0.7000000000000001 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / 平均露光時間: 31.8 sec. / 平均電子線量: 40.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー







 Z
Z Y
Y X
X