+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 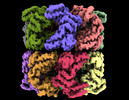 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
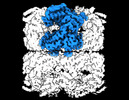
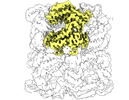
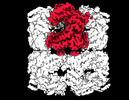
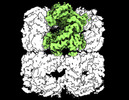
| タイトル | Cryo-EM structure of single empty ring 2 (SER2) of GroEL-UGT1A complex at 3.2 Ang. resolution | ||||||||||||||||||||||||
 マップデータ マップデータ | 3.2 Ang. Resolution Single Empty Ring2 (SER2) GroEL-UGT1A Structure Complex | ||||||||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報GroEL-GroES complex /  chaperonin ATPase / chaperonin ATPase /  virion assembly / chaperone cofactor-dependent protein refolding / virion assembly / chaperone cofactor-dependent protein refolding /  isomerase activity / ATP-dependent protein folding chaperone / response to radiation / unfolded protein binding / isomerase activity / ATP-dependent protein folding chaperone / response to radiation / unfolded protein binding /  フォールディング / response to heat ...GroEL-GroES complex / フォールディング / response to heat ...GroEL-GroES complex /  chaperonin ATPase / chaperonin ATPase /  virion assembly / chaperone cofactor-dependent protein refolding / virion assembly / chaperone cofactor-dependent protein refolding /  isomerase activity / ATP-dependent protein folding chaperone / response to radiation / unfolded protein binding / isomerase activity / ATP-dependent protein folding chaperone / response to radiation / unfolded protein binding /  フォールディング / response to heat / protein refolding / magnesium ion binding / フォールディング / response to heat / protein refolding / magnesium ion binding /  ATP hydrolysis activity / ATP hydrolysis activity /  ATP binding / ATP binding /  生体膜 / identical protein binding / 生体膜 / identical protein binding /  細胞質基質 細胞質基質類似検索 - 分子機能 | ||||||||||||||||||||||||
| 生物種 |   Escherichia coli (大腸菌) / Escherichia coli (大腸菌) /   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||||||||||||||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.26 Å クライオ電子顕微鏡法 / 解像度: 3.26 Å | ||||||||||||||||||||||||
 データ登録者 データ登録者 | Stapleton K / Takagi J / Mizohata E | ||||||||||||||||||||||||
| 資金援助 |  日本, 7件 日本, 7件
| ||||||||||||||||||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Unmasking GroEL: Structure, dynamics, and substrate binding revealed by single-particle cryo-EM 著者: Stapleton KM / Mizobata T / Miyazaki N / Takatsuji T / Kato T / Iwasaki K / Standley DM / Kawamura T / Nakane T / Takagi J / Mizohata E | ||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_33351.map.gz emd_33351.map.gz | 80 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-33351-v30.xml emd-33351-v30.xml emd-33351.xml emd-33351.xml | 21.2 KB 21.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_33351_fsc.xml emd_33351_fsc.xml | 10.7 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_33351.png emd_33351.png | 130.8 KB | ||
| その他 |  emd_33351_half_map_1.map.gz emd_33351_half_map_1.map.gz emd_33351_half_map_2.map.gz emd_33351_half_map_2.map.gz | 30 MB 30 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-33351 http://ftp.pdbj.org/pub/emdb/structures/EMD-33351 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33351 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-33351 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  7xolMC  7xojC  7xokC  7xomC  7xonC  7xooC  7xopC  7xoqC  7xorC  7xosC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_33351.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_33351.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | 3.2 Ang. Resolution Single Empty Ring2 (SER2) GroEL-UGT1A Structure Complex | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.87 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: half-map-1 3.2 Ang. Resolution Single Empty Ring2 (SER2) GroEL-UGT1A...
| ファイル | emd_33351_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half-map-1__3.2 Ang. Resolution Single Empty Ring2 (SER2) GroEL-UGT1A Structure Complex | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: half-map-2 3.2 Ang. Resolution Single Empty Ring2 (SER2) GroEL-UGT1A...
| ファイル | emd_33351_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half-map-2__3.2 Ang. Resolution Single Empty Ring2 (SER2) GroEL-UGT1A Structure Complex | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Single empty ring 2 (SER2) of GroEL-UGT1A Complex
| 全体 | 名称: Single empty ring 2 (SER2) of GroEL-UGT1A Complex |
|---|---|
| 要素 |
|
-超分子 #1: Single empty ring 2 (SER2) of GroEL-UGT1A Complex
| 超分子 | 名称: Single empty ring 2 (SER2) of GroEL-UGT1A Complex / タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: all 詳細: GroEL complexed with unfolded UGT1A present in one ring |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-超分子 #2: GroEL
| 超分子 | 名称: GroEL / タイプ: complex / ID: 2 / キメラ: Yes / 親要素: 1 / 含まれる分子: all 詳細: The model was built in a map at 3.26 Ang. global resolution by FSC 0.143 |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
-超分子 #3: UDP-glucuronosyltransferase 1A (UGT1A)
| 超分子 | 名称: UDP-glucuronosyltransferase 1A (UGT1A) / タイプ: complex / ID: 3 / キメラ: Yes / 親要素: 1 / 詳細: Model was not built because of weak density map |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Chaperonin GroEL
| 分子 | 名称: Chaperonin GroEL / タイプ: protein_or_peptide / ID: 1 / コピー数: 14 / 光学異性体: LEVO / EC番号:  chaperonin ATPase chaperonin ATPase |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) / 株: K12 Escherichia coli (大腸菌) / 株: K12 |
| 分子量 | 理論値: 57.260504 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: AAKDVKFGND ARVKMLRGVN VLADAVKVTL GPKGRNVVLD KSFGAPTITK DGVSVAREIE LEDKFENMGA QMVKEVASKA NDAAGDGTT TATVLAQAII TEGLKAVAAG MNPMDLKRGI DKAVTAAVEE LKALSVPCSD SKAIAQVGTI SANSDETVGK L IAEAMDKV ...文字列: AAKDVKFGND ARVKMLRGVN VLADAVKVTL GPKGRNVVLD KSFGAPTITK DGVSVAREIE LEDKFENMGA QMVKEVASKA NDAAGDGTT TATVLAQAII TEGLKAVAAG MNPMDLKRGI DKAVTAAVEE LKALSVPCSD SKAIAQVGTI SANSDETVGK L IAEAMDKV GKEGVITVED GTGLQDELDV VEGMQFDRGY LSPYFINKPE TGAVELESPF ILLADKKISN IREMLPVLEA VA KAGKPLL IIAEDVEGEA LATLVVNTMR GIVKVAAVKA PGFGDRRKAM LQDIATLTGG TVISEEIGME LEKATLEDLG QAK RVVINK DTTTIIDGVG EEAAIQGRVA QIRQQIEEAT SDYDREKLQE RVAKLAGGVA VIKVGAATEV EMKEKKARVE DALH ATRAA VEEGVVAGGG VALIRVASKL ADLRGQNEDQ NVGIKVALRA MEAPLRQIVL NCGEEPSVVA NTVKGGDGNY GYNAA TEEY GNMIDMGILD PTKVTRSALQ YAASVAGLMI TTECMVTDLP KNDAADLGAA GGMGGMGGMG GMM |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 33 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
詳細: Sample containing GorEL-UGT1A was made fresh and used without undergoing any freeze-thaw cycles to avoid degradation in the solution. The sample was in a buffer solution of 150mM NaCl, 20mM Tris-HCl at pH 7.5 | |||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 90 sec. / 前処理 - 雰囲気: AIR | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV 詳細: 3 ul of 33 mg/ml GroEL-UGT1A was placed on Holey carbon Quanitifoil copper grids (300 mesh size R1.2/1.3) and blotted for 3 seconds (blot force =1). |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.2 µm Bright-field microscopy / 最大 デフォーカス(公称値): 2.0 µm / 最小 デフォーカス(公称値): 1.2 µm |
| 特殊光学系 | エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 2951 / 平均電子線量: 40.0 e/Å2 詳細: Using a Titan KRIOS TEM operated at 300 kV, 2,951 movies were collected using the Falcon III DED in Counting mode at a magnification of 75000x corresponding to a pixel size of 0.87 Pixel/A at ...詳細: Using a Titan KRIOS TEM operated at 300 kV, 2,951 movies were collected using the Falcon III DED in Counting mode at a magnification of 75000x corresponding to a pixel size of 0.87 Pixel/A at the specimen level. All 2,951 movies were imported into the RELION pipeline and prepared for single-particle analysis |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 精密化 | 空間: REAL / プロトコル: RIGID BODY FIT / 温度因子: 65.94 |
|---|---|
| 得られたモデル |  PDB-7xol: |
 ムービー
ムービー コントローラー
コントローラー




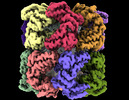
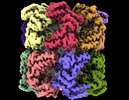
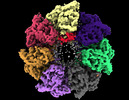



 Z
Z Y
Y X
X


















