+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-20918 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of the human TRPV3 K169A mutant determined in lipid nanodisc | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | ion channel TRPV3 /  MEMBRANE PROTEIN (膜タンパク質) MEMBRANE PROTEIN (膜タンパク質) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報negative regulation of hair cycle /  TRPチャネル / response to temperature stimulus / positive regulation of calcium ion import / calcium ion transmembrane transport / TRPチャネル / response to temperature stimulus / positive regulation of calcium ion import / calcium ion transmembrane transport /  calcium channel activity / calcium channel activity /  receptor complex / identical protein binding / receptor complex / identical protein binding /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.66 Å クライオ電子顕微鏡法 / 解像度: 3.66 Å | |||||||||
 データ登録者 データ登録者 | Deng Z / Yuan P | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2020 ジャーナル: Nat Struct Mol Biol / 年: 2020タイトル: Gating of human TRPV3 in a lipid bilayer. 著者: Zengqin Deng / Grigory Maksaev / Michael Rau / Zili Xie / Hongzhen Hu / James A J Fitzpatrick / Peng Yuan /  要旨: The transient receptor potential cation channel subfamily V member 3 (TRPV3) channel plays a critical role in skin physiology, and mutations in TRPV3 result in the development of a congenital skin ...The transient receptor potential cation channel subfamily V member 3 (TRPV3) channel plays a critical role in skin physiology, and mutations in TRPV3 result in the development of a congenital skin disorder, Olmsted syndrome. Here we describe multiple cryo-electron microscopy structures of human TRPV3 reconstituted into lipid nanodiscs, representing distinct functional states during the gating cycle. The ligand-free, closed conformation reveals well-ordered lipids interacting with the channel and two physical constrictions along the ion-conduction pore involving both the extracellular selectivity filter and intracellular helix bundle crossing. Both the selectivity filter and bundle crossing expand upon activation, accompanied by substantial structural rearrangements at the cytoplasmic intersubunit interface. Transition to the inactivated state involves a secondary structure change of the pore-lining helix, which contains a π-helical segment in the closed and open conformations, but becomes entirely α-helical upon inactivation. Together with electrophysiological characterization, structures of TRPV3 in a lipid membrane environment provide unique insights into channel activation and inactivation mechanisms. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_20918.map.gz emd_20918.map.gz | 59.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-20918-v30.xml emd-20918-v30.xml emd-20918.xml emd-20918.xml | 10.1 KB 10.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_20918.png emd_20918.png | 115.4 KB | ||
| Filedesc metadata |  emd-20918.cif.gz emd-20918.cif.gz | 5.4 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-20918 http://ftp.pdbj.org/pub/emdb/structures/EMD-20918 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20918 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-20918 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_20918.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_20918.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : human TRPV3
| 全体 | 名称: human TRPV3 |
|---|---|
| 要素 |
|
-超分子 #1: human TRPV3
| 超分子 | 名称: human TRPV3 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: Transient receptor potential cation channel subfamily V member 3
| 分子 | 名称: Transient receptor potential cation channel subfamily V member 3 タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 90.670688 KDa |
| 組換発現 | 生物種:  Pichia kluyveri (酵母) Pichia kluyveri (酵母) |
| 配列 | 文字列: MKAHPKEMVP LMGKRVAAPS GNPAVLPEKR PAEITPTKKS AHFFLEIEGF EPNPTVAKTS PPVFSKPMDS NIRQCISGNC DDMDSPQSP QDDVTETPSN PNSPSAQLAK EEQRRKKRRL KKRIFAAVSE GCVEELVELL VELQELCRRR HDEDVPDFLM H KLTASDTG ...文字列: MKAHPKEMVP LMGKRVAAPS GNPAVLPEKR PAEITPTKKS AHFFLEIEGF EPNPTVAKTS PPVFSKPMDS NIRQCISGNC DDMDSPQSP QDDVTETPSN PNSPSAQLAK EEQRRKKRRL KKRIFAAVSE GCVEELVELL VELQELCRRR HDEDVPDFLM H KLTASDTG ATCLMKALLN INPNTKEIVR ILLAFAEEND ILGRFINAEY TEEAYEGQTA LNIAIERRQG DIAALLIAAG AD VNAHAKG AFFNPKYQHE GFYFGETPLA LAACTNQPEI VQLLMEHEQT DITSRDSRGN NILHALVTVA EDFKTQNDFV KRM YDMILL RSGNWELETT RNNDGLTPLQ LAAKMGKAEI LKYILSREIK EKRLRSLSRK FTDWAYGPVS SSLYDLTNVD TTTD NSVLE ITVYNTNIDN RHEMLTLEPL HTLLHMKWKK FAKHMFFLSF CFYFFYNITL TLVSYYRPRE EEAIPHPLAL THKMG WLQL LGRMFVLIWA MCISVKEGIA IFLLRPSDLQ SILSDAWFHF VFFIQAVLVI LSVFLYLFAY KEYLACLVLA MALGWA NML YYTRGFQSMG MYSVMIQKVI LHDVLKFLFV YIVFLLGFGV ALASLIEKCP KDNKDCSSYG SFSDAVLELF KLTIGLG DL NIQQNSKYPI LFLFLLITYV ILTFVLLLNM LIALMGETVE NVSKESERIW RLQRARTILE FEKMLPEWLR SRFRMGEL C KVAEDDFRLC LRINEVKWTE WKTHVSFLNE DPGPVRRTDF NKIQDSSRNN SKTTLNAFEE VEEFPETSV UniProtKB: Transient receptor potential cation channel subfamily V member 3 |
-分子 #2: [(2~{R})-1-[2-azanylethoxy(oxidanyl)phosphoryl]oxy-3-hexadecanoyl...
| 分子 | 名称: [(2~{R})-1-[2-azanylethoxy(oxidanyl)phosphoryl]oxy-3-hexadecanoyloxy-propan-2-yl] (~{Z})-octadec-9-enoate タイプ: ligand / ID: 2 / コピー数: 20 / 式: 6OU |
|---|---|
| 分子量 | 理論値: 717.996 Da |
| Chemical component information | 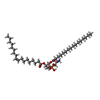 ChemComp-6OU: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: OTHER |
| 撮影 | #0 - Image recording ID: 1 #0 - フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) #0 - 平均電子線量: 62.0 e/Å2 / #1 - Image recording ID: 2 #1 - フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) #1 - 平均電子線量: 62.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: OTHER |
|---|---|
| 初期 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 角度割当 | タイプ: ANGULAR RECONSTITUTION |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.66 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 85510 |
| Image recording ID | 1 |
 ムービー
ムービー コントローラー
コントローラー



















