-検索条件
-検索結果
検索 (著者・登録者: pei & h)の結果1,330件中、1から50件目までを表示しています

EMDB-38215: 
Human GPR34 -Gi complex bound to S3E-LysoPS

EMDB-38217: 
Human GPR34 -Gi complex bound to S3E-LysoPS, receptor focused

PDB-8xbe: 
Human GPR34 -Gi complex bound to S3E-LysoPS

PDB-8xbg: 
Human GPR34 -Gi complex bound to S3E-LysoPS, receptor focused
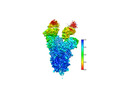
EMDB-37240: 
SARS-CoV-2 Omicron spike in complex with 5817 Fab

EMDB-37241: 
The interface structure of Omicron RBD binding to 5817 Fab

PDB-8khc: 
SARS-CoV-2 Omicron spike in complex with 5817 Fab

PDB-8khd: 
The interface structure of Omicron RBD binding to 5817 Fab

EMDB-35832: 
Cryo-EM structure of the GPR34 receptor in complex with the antagonist YL-365

PDB-8iyx: 
Cryo-EM structure of the GPR34 receptor in complex with the antagonist YL-365

EMDB-36634: 
5-HT1A-Gi in complex with compound (R)-IHCH-7179

PDB-8jt6: 
5-HT1A-Gi in complex with compound (R)-IHCH-7179
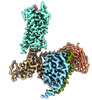
EMDB-36144: 
Senktide bound to active human neurokinin 3 receptor in complex with Gq

PDB-8jbf: 
Senktide bound to active human neurokinin 3 receptor in complex with Gq
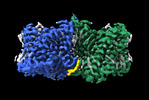
EMDB-36448: 
Structure of AE2 in complex with PIP2
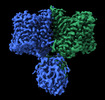
EMDB-36449: 
Structure of R932A/K1147A/H1148A mutant AE2

PDB-8jni: 
Structure of AE2 in complex with PIP2

PDB-8jnj: 
Structure of R932A/K1147A/H1148A mutant AE2

EMDB-36145: 
Neurokinin B bound to active human neurokinin 3 receptor in complex with Gq

EMDB-36146: 
Substance P bound to active human neurokinin 3 receptor in complex with Gq

PDB-8jbg: 
Neurokinin B bound to active human neurokinin 3 receptor in complex with Gq

PDB-8jbh: 
Substance P bound to active human neurokinin 3 receptor in complex with Gq

EMDB-34928: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.2 S-trimer in complex with fab L4.65 and L5.34

EMDB-34940: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.4 S-trimer in complex with fab L4.65 and L5.34

EMDB-34945: 
Cryo-EM structure of SARS-CoV-2 Omicron Prototype S-trimer in complex with fab L4.65 and L5.34

PDB-8hp9: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.2 S-trimer in complex with fab L4.65 and L5.34

PDB-8hpq: 
Cryo-EM structure of SARS-CoV-2 Omicron BA.4 S-trimer in complex with fab L4.65 and L5.34

PDB-8hpv: 
Cryo-EM structure of SARS-CoV-2 Omicron Prototype S-trimer in complex with fab L4.65 and L5.34

EMDB-35758: 
Cryo-EM structure of mouse BIRC6, N-terminal section optimized

EMDB-35759: 
Cryo-EM structure of mouse BIRC6, Global map

EMDB-35760: 
Cryo-EM structure of mouse BIRC6, with endogenous Smac binding

EMDB-38461: 
Cryo-EM structure of mouse BIRC6, Core region

EMDB-38462: 
Cryo-EM structure of mouse BIRC6, Half map of the core region

EMDB-38464: 
Cryo-EM structure of mouse BIRC6, Composite map

PDB-8ivq: 
Cryo-EM structure of mouse BIRC6, Global map

EMDB-41623: 
Cryo-EM structure of CPD-stalled Pol II in complex with Rad26 (engaged state)

EMDB-41647: 
Cryo-EM structure of CPD-stalled Pol II in complex with Rad26 (open state)

EMDB-41648: 
Cryo-EM structure of CPD stalled 10-subunit Pol II in complex with Rad26

EMDB-41650: 
Cryo-EM structure of backtracked Pol II in complex with Rad26

EMDB-41652: 
Cryo-EM structure of backtracked Pol II

EMDB-41653: 
Cryo-EM structure of CPD-stalled Pol II (conformation 1)

EMDB-41654: 
Cryo-EM structure of CPD-stalled Pol II (Conformation 2)

EMDB-41655: 
Cryo-EM structure of CPD lesion containing RNA Polymerase II elongation complex with Rad26 and Elf1 (closed state)

PDB-8tug: 
Cryo-EM structure of CPD-stalled Pol II in complex with Rad26 (engaged state)

PDB-8tvp: 
Cryo-EM structure of CPD-stalled Pol II in complex with Rad26 (open state)

PDB-8tvq: 
Cryo-EM structure of CPD stalled 10-subunit Pol II in complex with Rad26

PDB-8tvs: 
Cryo-EM structure of backtracked Pol II in complex with Rad26

PDB-8tvv: 
Cryo-EM structure of backtracked Pol II

PDB-8tvw: 
Cryo-EM structure of CPD-stalled Pol II (conformation 1)

PDB-8tvx: 
Cryo-EM structure of CPD-stalled Pol II (Conformation 2)
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
