-検索条件
-検索結果
検索 (著者・登録者: ho & my)の結果1,016件中、1から50件目までを表示しています

EMDB-18170: 
YPEL5-bound WDR26-CTLH E3 ligase - assembly I

EMDB-18171: 
YPEL5-bound WDR26-CTLH E3 ligase - assembly II

EMDB-18172: 
NMNAT1 core-bound RANBP9-TWA1-WDR26 module of WDR26-CTLH E3 ligase

EMDB-18173: 
NMNAT1 loop-bound RANBP9-TWA1-WDR26 module of WDR26-CTLH E3 ligase

EMDB-18174: 
NMNAT1-bound WDR26-CTLH E3 ligase assembly I - class 1

EMDB-18175: 
NMNAT1-bound WDR26-CTLH E3 ligase assembly I - class 2

EMDB-18176: 
NMNAT1-bound WDR26-CTLH E3 ligase assembly II - class 1

EMDB-18177: 
NMNAT1-bound WDR26-CTLH E3 ligase assembly II - class 2

EMDB-18178: 
NMNAT1-bound WDR26-CTLH E3 ligase assembly II - class 3

EMDB-18316: 
Structure of the non-canonical CTLH E3 substrate receptor WDR26 bound to YPEL5

EMDB-18345: 
Structure of the non-canonical CTLH E3 substrate receptor WDR26 bound to NMNAT1 substrate

PDB-8qbn: 
Structure of the non-canonical CTLH E3 substrate receptor WDR26 bound to YPEL5

PDB-8qe8: 
Structure of the non-canonical CTLH E3 substrate receptor WDR26 bound to NMNAT1 substrate

EMDB-44496: 
Cryo-EM co-structure of AcrB with the CU032 efflux pump inhibitor

EMDB-44500: 
Cryo-EM co-structure of AcrB with the EPM35 efflux pump inhibitor

EMDB-44501: 
Cryo-EM co-structure of AcrB with the CU232 efflux pump inhibitor

EMDB-44506: 
Cryo-EM co-structure of AcrB with CU244

PDB-9bfh: 
Cryo-EM co-structure of AcrB with the CU032 efflux pump inhibitor

PDB-9bfm: 
Cryo-EM co-structure of AcrB with the EPM35 efflux pump inhibitor

PDB-9bfn: 
Cryo-EM co-structure of AcrB with the CU232 efflux pump inhibitor

PDB-9bft: 
Cryo-EM co-structure of AcrB with CU244

EMDB-34548: 
Spike 3-up RBD with THSC20.HVTR04 (Fab4): State - III

EMDB-34563: 
Spike 2-up RBD with THSC20.HVTR26 (Fab26): State - I

EMDB-34602: 
State - I: Spike 2-up RBD with THSC20.HVTR26 (Fab26)

EMDB-34603: 
State - II: Spike 3-up RBD with THSC20.HVTR26 (Fab26)

EMDB-34546: 
State - I: Spike 2-up RBD with THSC20.HVTR04 (Fab4)

EMDB-34547: 
Spike 3-up RBD with THSC20.HVTR04 (Fab4): State - II

EMDB-19767: 
Structure of a 2873 Scaffold Base DNA Origami V1

EMDB-19769: 
Structure of a 2873 Scaffold Base DNA Origami V2

EMDB-19770: 
Structure of a 2873 Scaffold Base DNA Origami V3

EMDB-19775: 
Structure of a 1033 Scaffold Base DNA Origami Nanostructure V4 with Desalted Purified Staples

EMDB-19776: 
Structure of a 1033 Scaffold Base DNA Origami Nanostructure V4 with HPLC Purified Staples

EMDB-19867: 
Cryo-EM Structure of a 1033 Scaffold Base DNA Origami Nanostructure V4 and TBA

EMDB-19874: 
Refinement Focused on the 1st Body of a 1033 Scaffold-Based DNA Origami Nanostructure V4 with TBA

EMDB-19875: 
Refinement Focused on the 2nd Body of a 1033 Scaffold-Based DNA Origami Nanostructure V4 with TBA

EMDB-19876: 
Refinement Focused on the 3rd Body of a 1033 Scaffold-Based DNA Origami Nanostructure V4 with TBA

PDB-9eoq: 
Cryo-EM Structure of a 1033 Scaffold Base DNA Origami Nanostructure V4 and TBA

EMDB-41363: 
Cryo-EM structure of DDB1deltaB-DDA1-DCAF5

PDB-8tl6: 
Cryo-EM structure of DDB1deltaB-DDA1-DCAF5

EMDB-40589: 
hPAD4 bound to Activating Fab hA362

EMDB-40590: 
hPAD4 bound to inhibitory Fab hI365

PDB-8smk: 
hPAD4 bound to Activating Fab hA362

PDB-8sml: 
hPAD4 bound to inhibitory Fab hI365
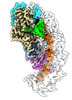
EMDB-29877: 
Exploiting Activation and Inactivation Mechanisms in Type I-C CRISPR-Cas3 for Genome Editing Applications

EMDB-29878: 
Exploiting Activation and Inactivation Mechanisms in Type I-C CRISPR-Cas3 for Genome Editing Applications

EMDB-29879: 
Exploiting Activation and Inactivation Mechanisms in Type I-C CRISPR-Cas3 for Genome Editing Applications

EMDB-29896: 
Exploiting Activation and Inactivation Mechanisms in Type I-C CRISPR-Cas3 for Genome Editing Applications
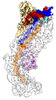
EMDB-29900: 
Exploiting Activation and Inactivation Mechanisms in Type I-C CRISPR-Cas3 for Genome Editing Applications

EMDB-29901: 
Exploiting Activation and Inactivation Mechanisms in Type I-C CRISPR-Cas3 for Genome Editing Applications

PDB-8g9s: 
Exploiting Activation and Inactivation Mechanisms in Type I-C CRISPR-Cas3 for Genome Editing Applications
ページ:
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア EMN検索について
EMN検索について



 wwPDBはEMDBデータモデルのバージョン3へ移行します
wwPDBはEMDBデータモデルのバージョン3へ移行します
