+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human DNA polymerase theta helicase domain dimer bound to DNA in the microhomology annealed conformation | |||||||||
 マップデータ マップデータ | Main sharp map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  DNA repair (DNA修復) / TMEJ / MMEJ / DNA repair (DNA修復) / TMEJ / MMEJ /  DNA binding protein (DNA結合タンパク質) DNA binding protein (DNA結合タンパク質) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報single-stranded DNA endodeoxyribonuclease activity / HDR through MMEJ (alt-NHEJ) / double-strand break repair via alternative nonhomologous end joining / single-stranded DNA helicase activity / replication fork processing / site of DNA damage / 5'-deoxyribose-5-phosphate lyase activity / negative regulation of double-strand break repair via homologous recombination / somatic hypermutation of immunoglobulin genes / error-prone translesion synthesis ...single-stranded DNA endodeoxyribonuclease activity / HDR through MMEJ (alt-NHEJ) / double-strand break repair via alternative nonhomologous end joining / single-stranded DNA helicase activity / replication fork processing / site of DNA damage / 5'-deoxyribose-5-phosphate lyase activity / negative regulation of double-strand break repair via homologous recombination / somatic hypermutation of immunoglobulin genes / error-prone translesion synthesis /  DNA helicase activity / DNA helicase activity /  塩基除去修復 / protein homooligomerization / 塩基除去修復 / protein homooligomerization /  逆転写酵素 / 逆転写酵素 /  RNA-directed DNA polymerase activity / double-strand break repair / site of double-strand break / RNA-directed DNA polymerase activity / double-strand break repair / site of double-strand break /  ヘリカーゼ / damaged DNA binding / ヘリカーゼ / damaged DNA binding /  DNAポリメラーゼ / DNAポリメラーゼ /  DNA-directed DNA polymerase activity / DNA-directed DNA polymerase activity /  DNA修復 / DNA damage response / DNA修復 / DNA damage response /  chromatin binding / chromatin binding /  ゴルジ体 / magnesium ion binding / ゴルジ体 / magnesium ion binding /  ATP hydrolysis activity / ATP hydrolysis activity /  核質 / 核質 /  ATP binding / identical protein binding / ATP binding / identical protein binding /  細胞質基質 細胞質基質類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) / synthetic construct (人工物) Homo sapiens (ヒト) / synthetic construct (人工物) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.5 Å クライオ電子顕微鏡法 / 解像度: 3.5 Å | |||||||||
 データ登録者 データ登録者 | Zerio CJ / Lander GC | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Human polymerase theta helicase positions DNA microhomologies for double-strand break repair 著者: Zerio CJ / Bai Y / Sosa-Alvarado BA / Guzi T / Lander GC | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_44538.map.gz emd_44538.map.gz | 122.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-44538-v30.xml emd-44538-v30.xml emd-44538.xml emd-44538.xml | 25.4 KB 25.4 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_44538_fsc.xml emd_44538_fsc.xml | 12.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_44538.png emd_44538.png | 97.9 KB | ||
| マスクデータ |  emd_44538_msk_1.map emd_44538_msk_1.map | 129.7 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-44538.cif.gz emd-44538.cif.gz | 7.7 KB | ||
| その他 |  emd_44538_additional_1.map.gz emd_44538_additional_1.map.gz emd_44538_half_map_1.map.gz emd_44538_half_map_1.map.gz emd_44538_half_map_2.map.gz emd_44538_half_map_2.map.gz | 5.5 MB 120.6 MB 120.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-44538 http://ftp.pdbj.org/pub/emdb/structures/EMD-44538 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-44538 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-44538 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_44538.map.gz / 形式: CCP4 / 大きさ: 129.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_44538.map.gz / 形式: CCP4 / 大きさ: 129.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Main sharp map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.833 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_44538_msk_1.map emd_44538_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
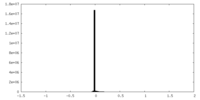
| 密度ヒストグラム |
-追加マップ: Locally filtered map
| ファイル | emd_44538_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Locally filtered map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
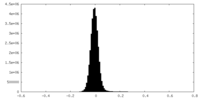
| 密度ヒストグラム |
-ハーフマップ: Half map A
| ファイル | emd_44538_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
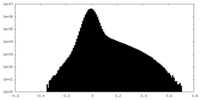
| 密度ヒストグラム |
-ハーフマップ: Half map B
| ファイル | emd_44538_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
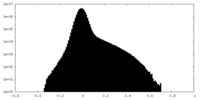
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Human DNA polymerase theta helicase domain bound to stem-loop DNA...
| 全体 | 名称: Human DNA polymerase theta helicase domain bound to stem-loop DNA with microhomology in the 3' overhang |
|---|---|
| 要素 |
|
-超分子 #1: Human DNA polymerase theta helicase domain bound to stem-loop DNA...
| 超分子 | 名称: Human DNA polymerase theta helicase domain bound to stem-loop DNA with microhomology in the 3' overhang タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 20 KDa |
-超分子 #2: Human DNA polymerase theta helicase domain
| 超分子 | 名称: Human DNA polymerase theta helicase domain / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #3: Stem-loop DNA with microhomology in the 3' overhang
| 超分子 | 名称: Stem-loop DNA with microhomology in the 3' overhang / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #2 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-分子 #1: DNA polymerase theta
| 分子 | 名称: DNA polymerase theta / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO / EC番号:  ヘリカーゼ ヘリカーゼ |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 99.671344 KDa |
| 組換発現 | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) |
| 配列 | 文字列: NLLRRSGKRR RSESGSDSFS GSGGDSSASP QFLSGSVLSP PPGLGRCLKA AAAGECKPTV PDYERDKLLL ANWGLPKAVL EKYHSFGVK KMFEWQAECL LLGQVLEGKN LVYSAPTSAG KTLVAELLIL KRVLEMRKKA LFILPFVSVA KEKKYYLQSL F QEVGIKVD ...文字列: NLLRRSGKRR RSESGSDSFS GSGGDSSASP QFLSGSVLSP PPGLGRCLKA AAAGECKPTV PDYERDKLLL ANWGLPKAVL EKYHSFGVK KMFEWQAECL LLGQVLEGKN LVYSAPTSAG KTLVAELLIL KRVLEMRKKA LFILPFVSVA KEKKYYLQSL F QEVGIKVD GYMGSTSPSR HFSSLDIAVC TIERANGLIN RLIEENKMDL LGMVVVDELH MLGDSHRGYL LELLLTKICY IT RKSASCQ ADLASSLSNA VQIVGMSATL PNLELVASWL NAELYHTDFR PVPLLESVKV GNSIYDSSMK LVREFEPMLQ VKG DEDHVV SLCYETICDN HSVLLFCPSK KWCEKLADII AREFYNLHHQ AEGLVKPSEC PPVILEQKEL LEVMDQLRRL PSGL DSVLQ KTVPWGVAFH HAGLTFEERD IIEGAFRQGL IRVLAATSTL SSGVNLPARR VIIRTPIFGG RPLDILTYKQ MVGRA GRKG VDTVGESILI CKNSEKSKGI ALLQGSLKPV RSCLQRREGE EVTGSMIRAI LEIIVGGVAS TSQDMHTYAA CTFLAA SMK EGKQGIQRNQ ESVQLGAIEA CVMWLLENEF IQSTEASDGT EGKVYHPTHL GSATLSSSLS PADTLDIFAD LQRAMKG FV LENDLHILYL VTPMFEDWTT IDWYRFFCLW EKLPTSMKRV AELVGVEEGF LARCVKGKVV ARTERQHRQM AIHKRFFT S LVLLDLISEV PLREINQKYG CNRGQIQSLQ QSAAVYAGMI TVFSNRLGWH NMELLLSQFQ KRLTFGIQRE LCDLVRVSL LNAQRARVLY ASGFHTVADL ARANIVEVEV ILKNAVPFKS ARKAVDEEEE AVEERRNMRT IWVTGRKGLT EREAAALIVE EARMILQQD LVEM UniProtKB:  DNA polymerase theta DNA polymerase theta |
-分子 #2: Stem-loop DNA with microhomology in the 3' overhang
| 分子 | 名称: Stem-loop DNA with microhomology in the 3' overhang / タイプ: dna / ID: 2 / コピー数: 2 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 17.264012 KDa |
| 配列 | 文字列: (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DT)(DT)(DT)(DT)(DC)(DA)(DC)(DT) (DG)(DT)(DG)(DA)(DG)(DC)(DT)(DT)(DA) (DG)(DC)(DG)(DT)(DT)(DA)(DG)(DA)(DG)(DT) (DA) (DG)(DG)(DT)(DT)(DT) ...文字列: (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DT)(DT)(DT)(DT)(DC)(DA)(DC)(DT) (DG)(DT)(DG)(DA)(DG)(DC)(DT)(DT)(DA) (DG)(DC)(DG)(DT)(DT)(DA)(DG)(DA)(DG)(DT) (DA) (DG)(DG)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DG)(DC)(DC)(DC)(DG)(DG)(DG) |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1.25 mg/mL | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.5 構成要素:
| ||||||||||||
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 30 sec. 詳細: Grids were glow discharged under vacuum for 30 s at 15 mA in a Pelco easiGlow 91000 Glow Discharge Cleaning System. | ||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 95 % / チャンバー内温度: 277 K / 装置: HOMEMADE PLUNGER 詳細: 3 microliters of sample was applied to the surface of the grid, blotted with Whatman 1 filter paper until 2 seconds after the liquid spot on the filter paper stopped spreading, and the grid ...詳細: 3 microliters of sample was applied to the surface of the grid, blotted with Whatman 1 filter paper until 2 seconds after the liquid spot on the filter paper stopped spreading, and the grid was plunged into a liquid ethane bath cooled by liquid nitrogen.. | ||||||||||||
| 詳細 | We added 2X pre-annealed DNA to the protein, lowered the NaCl concentration to 100 mM, and purified the DNA-bound complex by SEC. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 150.0 µm / 最大 デフォーカス(補正後): 2.5 µm / 最小 デフォーカス(補正後): 0.3 µm / 倍率(補正後): 60024 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 105000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.0 µm / 最小 デフォーカス(公称値): 1.0 µm / 倍率(公称値): 105000 |
| 特殊光学系 | エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 温度 | 最低: 70.0 K / 最高: 77.0 K |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) デジタル化 - サイズ - 横: 5760 pixel / デジタル化 - サイズ - 縦: 4092 pixel / 撮影したグリッド数: 1 / 実像数: 12440 / 平均露光時間: 1.4 sec. / 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル |
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 詳細 | We modeled DNA bases into sharpened density when possible. We modeled the DNA backbone into locally filtered density in low resolution areas. | |||||||||
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT / 温度因子: 53.26 当てはまり具合の基準: Cross-correlation coefficient | |||||||||
| 得られたモデル |  PDB-9bha: |
 ムービー
ムービー コントローラー
コントローラー















 Z
Z Y
Y X
X