+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Umb1 umbrella toxin particle | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  Streptomyces (ストレプトマイセス属) / Umbrella toxin particles / Alanine leucine phenylalanine-rich (ALF) repeat proteins / Seattle structural genomics center for infectious disease / SSGCID / Streptomyces (ストレプトマイセス属) / Umbrella toxin particles / Alanine leucine phenylalanine-rich (ALF) repeat proteins / Seattle structural genomics center for infectious disease / SSGCID /  TOXIN (毒素) TOXIN (毒素) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Streptomyces coelicolor A3(2) (バクテリア) Streptomyces coelicolor A3(2) (バクテリア) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.3 Å クライオ電子顕微鏡法 / 解像度: 4.3 Å | |||||||||
 データ登録者 データ登録者 | Park YJ / Zhao Q / Seattle Structural Genomics Center for Infectious Disease (SSGCID) / DiMaio F / Mougous JD / Veesler D | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nature / 年: 2024 ジャーナル: Nature / 年: 2024タイトル: Streptomyces umbrella toxin particles block hyphal growth of competing species. 著者: Qinqin Zhao / Savannah Bertolli / Young-Jun Park / Yongjun Tan / Kevin J Cutler / Pooja Srinivas / Kyle L Asfahl / Citlali Fonesca-García / Larry A Gallagher / Yaqiao Li / Yaxi Wang / Devin ...著者: Qinqin Zhao / Savannah Bertolli / Young-Jun Park / Yongjun Tan / Kevin J Cutler / Pooja Srinivas / Kyle L Asfahl / Citlali Fonesca-García / Larry A Gallagher / Yaqiao Li / Yaxi Wang / Devin Coleman-Derr / Frank DiMaio / Dapeng Zhang / S Brook Peterson / David Veesler / Joseph D Mougous /  要旨: Streptomyces are a genus of ubiquitous soil bacteria from which the majority of clinically utilized antibiotics derive. The production of these antibacterial molecules reflects the relentless ...Streptomyces are a genus of ubiquitous soil bacteria from which the majority of clinically utilized antibiotics derive. The production of these antibacterial molecules reflects the relentless competition Streptomyces engage in with other bacteria, including other Streptomyces species. Here we show that in addition to small-molecule antibiotics, Streptomyces produce and secrete antibacterial protein complexes that feature a large, degenerate repeat-containing polymorphic toxin protein. A cryo-electron microscopy structure of these particles reveals an extended stalk topped by a ringed crown comprising the toxin repeats scaffolding five lectin-tipped spokes, which led us to name them umbrella particles. Streptomyces coelicolor encodes three umbrella particles with distinct toxin and lectin composition. Notably, supernatant containing these toxins specifically and potently inhibits the growth of select Streptomyces species from among a diverse collection of bacteria screened. For one target, Streptomyces griseus, inhibition relies on a single toxin and that intoxication manifests as rapid cessation of vegetative hyphal growth. Our data show that Streptomyces umbrella particles mediate competition among vegetative mycelia of related species, a function distinct from small-molecule antibiotics, which are produced at the onset of reproductive growth and act broadly. Sequence analyses suggest that this role of umbrella particles extends beyond Streptomyces, as we identified umbrella loci in nearly 1,000 species across Actinobacteria. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_43736.map.gz emd_43736.map.gz | 101.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-43736-v30.xml emd-43736-v30.xml emd-43736.xml emd-43736.xml | 19.8 KB 19.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_43736.png emd_43736.png | 53.7 KB | ||
| Filedesc metadata |  emd-43736.cif.gz emd-43736.cif.gz | 6.6 KB | ||
| その他 |  emd_43736_additional_1.map.gz emd_43736_additional_1.map.gz emd_43736_half_map_1.map.gz emd_43736_half_map_1.map.gz emd_43736_half_map_2.map.gz emd_43736_half_map_2.map.gz | 54 MB 99.6 MB 99.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-43736 http://ftp.pdbj.org/pub/emdb/structures/EMD-43736 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-43736 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-43736 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8w20MC  8w22C M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_43736.map.gz / 形式: CCP4 / 大きさ: 107.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_43736.map.gz / 形式: CCP4 / 大きさ: 107.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.686 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-追加マップ: #1
| ファイル | emd_43736_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
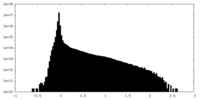
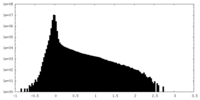
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_43736_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
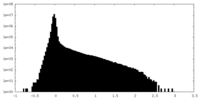
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_43736_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
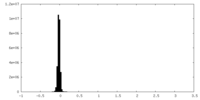
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Umb1 umbrella toxin particle
| 全体 | 名称: Umb1 umbrella toxin particle |
|---|---|
| 要素 |
|
-超分子 #1: Umb1 umbrella toxin particle
| 超分子 | 名称: Umb1 umbrella toxin particle / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Streptomyces coelicolor A3(2) (バクテリア) Streptomyces coelicolor A3(2) (バクテリア) |
-分子 #1: Secreted protein
| 分子 | 名称: Secreted protein / タイプ: protein_or_peptide / ID: 1 / コピー数: 5 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Streptomyces coelicolor A3(2) (バクテリア) Streptomyces coelicolor A3(2) (バクテリア) |
| 分子量 | 理論値: 17.18627 KDa |
| 配列 | 文字列: MANTSRTRQA LMAIAVSVLA AGVTTLGVAH ADNGDAVAAA AEMPQAVEDF SYPGAAKIQA ETGAILKRGN GHMLMTSCDG SEDIQVMSR TGQKDFCFNV MAKPAYLTLE VPQAYGIWTS ADPVKTTIKD TDGTATVINA PANDFTGYGE AGSTGEPTTL I ELRVAG UniProtKB:  Secreted protein Secreted protein |
-分子 #2: Secreted esterase
| 分子 | 名称: Secreted esterase / タイプ: protein_or_peptide / ID: 2 詳細: endogenous tags to purified the complex from natural source コピー数: 5 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Streptomyces coelicolor A3(2) (バクテリア) Streptomyces coelicolor A3(2) (バクテリア) |
| 分子量 | 理論値: 54.437102 KDa |
| 配列 | 文字列: MFRMPRPIRA TALSAAVLAG ALASTPAQAT VGDPTTDAKL DFTARLTIGT DYRSCSGALV DTQWVLTAAS CFADDPNQPD TVAAGKPAQ LTRATVGRAD SNIANGYVRE VVELVPHPER DMVLARLDKA IPDIAPVRLA SDAPTAGTPL TAVGFGRTKD E WVPIQRHQ ...文字列: MFRMPRPIRA TALSAAVLAG ALASTPAQAT VGDPTTDAKL DFTARLTIGT DYRSCSGALV DTQWVLTAAS CFADDPNQPD TVAAGKPAQ LTRATVGRAD SNIANGYVRE VVELVPHPER DMVLARLDKA IPDIAPVRLA SDAPTAGTPL TAVGFGRTKD E WVPIQRHQ GAFTVTSVTA GAVNVTGQGG DAICAGDTGG PLLQDKNGTL HLVGVNNRSM QGGCYGSETT STDAIAAMSD AD FVTQTVN RDLGTGNLSD LVASADFNSD GRTDVAAVLE DGSLHAFYAK PDGTLEYGRE LWNDNTWSPM VQIIGGDFNS DGN GDIAAV RSDGTLNLYT GTATGILNKS KPMWHDTSWK TIKQVTRFKF NGRDGLVAQW GDGNLYGYYT GTDGTLTGTK VKMW PDATW GKTRLTGTAD INADGRDDLT AVRDDGSLNW YAGNTKGGLD AARKLWPDNT WTPMKRIIGG DFNGDNKGDI AAVGG QSTL LLYTGTGTGT LNKGIAMRPA SGSHHHHHHH H UniProtKB: Secreted esterase |
-分子 #3: Intein C-terminal splicing domain-containing protein
| 分子 | 名称: Intein C-terminal splicing domain-containing protein タイプ: protein_or_peptide / ID: 3 詳細: endogenous tags to purified the complex from natural source コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Streptomyces coelicolor A3(2) (バクテリア) Streptomyces coelicolor A3(2) (バクテリア) |
| 分子量 | 理論値: 142.716734 KDa |
| 配列 | 文字列: MRRRIPSRTP GSGAKQKSWF PRRSLQVLLS AGMLSGLLGT PAALAAEPAP LPANVRADIV GYWETGGAGL KAAAEQALLG GDEAIRKFL ADAPSIQHDD NRIDAARMAM TGGSGLRQAA KDAIRLSPAE LEKFLLYGYE EPLDDDHKVE IARLINLGGP G VREAGKAA ...文字列: MRRRIPSRTP GSGAKQKSWF PRRSLQVLLS AGMLSGLLGT PAALAAEPAP LPANVRADIV GYWETGGAGL KAAAEQALLG GDEAIRKFL ADAPSIQHDD NRIDAARMAM TGGSGLRQAA KDAIRLSPAE LEKFLLYGYE EPLDDDHKVE IARLINLGGP G VREAGKAA LQGTAEEREL FLNSGQYTAQ QDDNRVDVAR LATTGGPNVQ AAAKVALRGT PEDMVEFLEV GQFTARNRDQ EH ATIAELI KQAELAGKQA DDARKTAEES SKKAVDASQL AKEAAQKAAE ETEAAKDDSQ KAAVKAKQAA DAARAAADAA QEA IGSANA ANRAARRAAL AAAQTASAAT AAAEAANKAY KAAIAAAGDE GKADEAKEAA KQARAAADAA TKSGLAAENA GLAS AAAAT ASTAAKSASS NARAAAGAAE EANQHADAAG VHSNEAALAA AEARRHADAA DRAADRSSAL AQRAATAAYG ARDAA NSAA EHANKAADYA DESAAHAGDS AAYAATAKRN AQAAQEAAKT ATAAVTKANE IFKLARETET ANLETRTDAA IERARS MKS ASETSITASA TTQVEALALN DTATELAKEA SRPDIDVQAT AAKGRQLAMQ AMKLLGPWHQ EAAARALSGT DQDVLDY LR TRWKEANHND IRQQIVDLST QSPYASVRTA AAEALNGTPE QIEAFHTTGQ YTAGSDDMKV DVARLANTGG PGVSQAAK T ALADGTGKTL ATFLQIGQYG ERLSDEKVVT ARLAETGSPE VQAAAKIALA GPPELLHEFV TTGQYMAKRK DDLADVHVN QVERLLAEGS LIAAEANEDA WRATEAAATA EGAAADAATA AEKAEASAAQ AKQHAADADA SADAATRSAA DAAASAATAR DAADRAAQD ATAAENSAAE AAFSAAYARD SASKADDAAD RARASALAAG KSADEAEAEA KEAWKTTRAL AEKEAEEARR K AAEERKRQ QEQAGEPKRV CIPHPTRETM IPIMPCAASP DDSMIMPGPV DPTIRAVVWE LAGLNDIKAC IDKPLSGNCV MA VVGVTPW GKFKLVSKLG NGLDAVKDAR GARRTVACLT GAAHSFPAGT RVLMADGTRR SIEQIEAGDL VTATDPTTGE TGA RTVTRT IHTPDDRNFT DVALADGSTL TSTTHHPYWS QNDQTWKNAG DLEAGDTLRT PQNTAVVIAA THDWPGLQDA YDLT VDGFH SYYVSTGTTD VLVHNNDNPC PDWVSKAWKK LPKRKSGDPT SGYVFEADGT LVWDSVLTSG RSPLSEDISA FLKGS PDFP NFPGYADVAH HAEAKIAWEM RTKMGKGKKL HIVINTNYVC PKVSSPNSMG CKQAVPAILY EDQTLYVHYP GASDAL ELK GTAKR UniProtKB: Intein C-terminal splicing domain-containing protein |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.2 µm Bright-field microscopy / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.2 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 60.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: INSILICO MODEL |
|---|---|
| 初期 角度割当 | タイプ: PROJECTION MATCHING |
| 最終 角度割当 | タイプ: PROJECTION MATCHING |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 4.3 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC / 使用した粒子像数: 386275 |
 ムービー
ムービー コントローラー
コントローラー








 Z
Z Y
Y X
X