+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 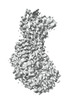 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM Structure of CdnG-E2 complex from Serratia marcescens | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  cGAS / CdnG / E2 / CBASS / cGAS / CdnG / E2 / CBASS /  ANTIVIRAL PROTEIN ANTIVIRAL PROTEIN | |||||||||
| 機能・相同性 | Uncharacterized protein 機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Serratia marcescens (霊菌) Serratia marcescens (霊菌) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.3 Å クライオ電子顕微鏡法 / 解像度: 3.3 Å | |||||||||
 データ登録者 データ登録者 | Xiao J / Wang L | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Microbiol / 年: 2024 ジャーナル: Nat Microbiol / 年: 2024タイトル: Phage defence system CBASS is regulated by a prokaryotic E2 enzyme that imitates the ubiquitin pathway. 著者: Yan Yan / Jun Xiao / Fengtao Huang / Wei Xian / Bingbing Yu / Rui Cheng / Hui Wu / Xueling Lu / Xionglue Wang / Wenjing Huang / Jing Li / Greater Kayode Oyejobi / Carol V Robinson / Hao Wu / ...著者: Yan Yan / Jun Xiao / Fengtao Huang / Wei Xian / Bingbing Yu / Rui Cheng / Hui Wu / Xueling Lu / Xionglue Wang / Wenjing Huang / Jing Li / Greater Kayode Oyejobi / Carol V Robinson / Hao Wu / Di Wu / Xiaoyun Liu / Longfei Wang / Bin Zhu /    要旨: The cyclic-oligonucleotide-based anti-phage signalling system (CBASS) is a type of innate prokaryotic immune system. Composed of a cyclic GMP-AMP synthase (cGAS) and CBASS-associated proteins, CBASS ...The cyclic-oligonucleotide-based anti-phage signalling system (CBASS) is a type of innate prokaryotic immune system. Composed of a cyclic GMP-AMP synthase (cGAS) and CBASS-associated proteins, CBASS uses cyclic oligonucleotides to activate antiviral immunity. One major class of CBASS contains a homologue of eukaryotic ubiquitin-conjugating enzymes, which is either an E1-E2 fusion or a single E2. However, the functions of single E2s in CBASS remain elusive. Here, using biochemical, genetic, cryo-electron microscopy and mass spectrometry investigations, we discover that the E2 enzyme from Serratia marcescens regulates cGAS by imitating the ubiquitination cascade. This includes the processing of the cGAS C terminus, conjugation of cGAS to a cysteine residue, ligation of cGAS to a lysine residue, cleavage of the isopeptide bond and poly-cGASylation. The poly-cGASylation activates cGAS to produce cGAMP, which acts as an antiviral signal and leads to cell death. Thus, our findings reveal a unique regulatory role of E2 in CBASS. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_39353.map.gz emd_39353.map.gz | 167.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-39353-v30.xml emd-39353-v30.xml emd-39353.xml emd-39353.xml | 14.8 KB 14.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_39353.png emd_39353.png | 90.6 KB | ||
| Filedesc metadata |  emd-39353.cif.gz emd-39353.cif.gz | 5.6 KB | ||
| その他 |  emd_39353_half_map_1.map.gz emd_39353_half_map_1.map.gz emd_39353_half_map_2.map.gz emd_39353_half_map_2.map.gz | 165.4 MB 165.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-39353 http://ftp.pdbj.org/pub/emdb/structures/EMD-39353 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-39353 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-39353 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8yjyMC  8hsbC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_39353.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_39353.map.gz / 形式: CCP4 / 大きさ: 178 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.67 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_39353_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_39353_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : CdnG-E2 binary complex
| 全体 | 名称: CdnG-E2 binary complex |
|---|---|
| 要素 |
|
-超分子 #1: CdnG-E2 binary complex
| 超分子 | 名称: CdnG-E2 binary complex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Serratia marcescens (霊菌) Serratia marcescens (霊菌) |
-分子 #1: SmCdnG
| 分子 | 名称: SmCdnG / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Serratia marcescens (霊菌) Serratia marcescens (霊菌) |
| 分子量 | 理論値: 45.481707 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MYGSTTARNL PSGKKQRIAD LLSQIIETLD LTKTQYANIE SAYNGVGTFL SEGDDPLLQD AVIYPQGSVR LNTTVKPKNE EQYDIDLIC YLPHATQADY TGVISAIRQR LESHKTYKTL LSELPRGFRI NYAGDYHLDI TPGRDHTGTA HPGQPLWVVD A QTAWKESN ...文字列: MYGSTTARNL PSGKKQRIAD LLSQIIETLD LTKTQYANIE SAYNGVGTFL SEGDDPLLQD AVIYPQGSVR LNTTVKPKNE EQYDIDLIC YLPHATQADY TGVISAIRQR LESHKTYKTL LSELPRGFRI NYAGDYHLDI TPGRDHTGTA HPGQPLWVVD A QTAWKESN PSGYAEWFES SASVQPLRTI LVMDSASRVG TEALLPLPDS TDKKLLNHIV QILKRHRDEW AAEQDEVRQR CR PISVIIT TLACHAYNHI IADRRAYDND LDILLDVLEL MPDFIVSTQG AIHVNNPHMP EENFAEKWNR SEQDEGPQRS EAF YQWHAA AQATFNTIAA SVGEDNLFLS LEDSFGKTPV DVVRQRLMEH MQSAREQGSL HLDKKTGGLI ATGLAGTAAQ AGVP KNTFY GE |
-分子 #2: Type VI secretion protein
| 分子 | 名称: Type VI secretion protein / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Serratia marcescens (霊菌) Serratia marcescens (霊菌) |
| 分子量 | 理論値: 18.78267 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MNNVVIRHHC KPLTIAQQYR ALKAGGPYER LRIIHHDRTL LWEGWLQPSL FSRRYKVAVR YSLGTPPICV VTEPDLFALA GTRAIPHLY PADKHIPGAR LCLFLPRSQA DDGLSEWRAQ LKISDTLIPW ASLWLFYFEQ WLHTGHWEGG GKHPRPSEVK N ER UniProtKB: Uncharacterized protein |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.5 µm Bright-field microscopy / 最大 デフォーカス(公称値): 3.0 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 再構成 | 解像度のタイプ: BY AUTHOR / 解像度: 3.3 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 使用した粒子像数: 263744 |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X