[wwPDB] PDBアーカイブとEMDBアーカイブのスナップショットを公開しました
2021年1月5日現在のPDBアーカイブ (wwPDB:ftp://ftp.wwpdb.org、 PDBj:ftp://ftp.pdbj.org)のスナップショットを、スナップショットサイト (wwPDB:ftp://snapshots.wwpdb.org、PDBj:ftp://snapshots.pdbj.org)に追加しました。 2005年1月以来、毎年、スナップショットをアーカイブし、PDBアーカイブに関する研究に役立つデータセットを提供し続けています。
ディレクトリ 20210105は、2021年1月5日時点での173,005件の実験的に決定された座標ファイルと、関連する実験データを含んでいます。 座標及び関連データのファイルは、PDBx/mmCIFフォーマット、PDBフォーマット、XMLフォーマットファイルで、それぞれご利用頂けます。 各ファイルの日付とタイムスタンプは、そのファイルが最後に更新された日付を表しています。 上記ディレクトリ内のPDBアーカイブのスナップショットのサイズは、822 GBとなっています。
2021年1月4日現在のEMDBアーカイブ(ftp://ftp.ebi.ac.uk/pub/databases/emdb)のスナップショットは、 ftp://ftp.ebi.ac.uk/pub/databases/emdb_vault/20210104/と、ftp://snapshots.pdbj.org/20210105/からご利用頂けます。 公開済みエントリー(13,731件)と廃止されたエントリー(142件)に対する、マップファイルとメタデータのXMLファイルを含んでおり、スナップショットのサイズは、2.9 TBとなっています。
[ wwPDB News ]
[wwPDB] 区切りとなる10,000件以上のエントリーを処理したバイオキュレータ
 2020年2月、SARS-CoV-2メインプロテアーゼについて語るRCSB PDBのバイオキュレータのBrian P. Hudson 博士
2020年2月、SARS-CoV-2メインプロテアーゼについて語るRCSB PDBのバイオキュレータのBrian P. Hudson 博士
wwPDBのバイオキュレーター Brian Hudson博士が処理したPDBエントリーが10,000件に到達しました。おめでとうございます。 Hudson博士は、カリフォルニア工科大学で化学の博士号を取得し、X線結晶学とクライオ電子顕微鏡を専門としています。 2010年にPDBに加わり、科学データと様々な実験技術への深い理解と、卓越した品質のデータキュレーションへの献身で、高度な専門家としての地位を確立しています。 Hudson博士はOneDepシステムが構築される以前から、1000件以上のEMマップをキュレーションしてきた、EMデータキュレーションのパイオニアの一人です。 wwPDBは、Hudson博士のこの素晴らしい成果を祝福するとともに、今後の活躍を期待しています。
[ wwPDB News ]
[wwPDB] 特定のEMエントリー登録時にハーフマップが必須になります
2022年2月25日より、単粒子、単粒子ベースのヘリカル、およびサブトモグラムの平均化再構成法によるエントリーについて、 ハーフマップをEMデータバンク(EMDB)に登録することが必須になります。 この変更はwwPDB EMDBコアアーカイブに対する長年の要望に応えたものであり、 2020年のwwPDB単粒子クライオ電子顕微鏡データ管理ワークショップ(準備中の論文)からの提言でもあります。 このワークショップで得られたいくつかの提言は、すでにwwPDB OneDepシステムに実装されています。 これには、wwPDB検証レポートの改善や、 登録インターフェイスを介したメタデータの取り込みの強化などが含まれます。
必須のハーフマップは、フィルタなし、マスクなし、シャープニングなしで、一次マップと重なり合うよう同じ座標空間と方向に配置されている必要があります。 ハーフマップの利用は、wwPDBの検証レポートに反映されているように、EM構造の検証の向上に貢献します。
wwPDBは、対象となる様式のクライオ電子顕微鏡処理ソフトウェアの開発者に対して、 (まだサポートされていない場合は)このようなハーフマップの出力をサポートするよう強く求めています。
このポリシーの変更についてご質問などございましたら、info@wwpdb.orgまで英語でお問い合わせください。
[ wwPDB News ]
「PDB アジア地区50周年記念シンポジウム」開催とPDBjポスター賞について
2021年11月24日(水)、PDB創設50周年を記念して「PDBアジア地区50周年記念シンポジウム~アジア地区構造生物学の最先端とProtein Data Bank 50年の歩み~」をオンラインで開催しました。国内外から300名以上の研究者のご参加をいただき盛会のうちに終えることができました。
ポスターセッションでは、アジアを中心に7か国から計78件の発表があり、そのうち、学生ならびに若手研究者による57件がPDBjポスター賞の審査対象となりました。
選考の結果、以下の4名が受賞者と決定しました。ここに発表するとともに、心よりお祝いいたします。受賞者のみなさんには証明書と副賞(書籍、PDBjノベルティグッズ)をお送りします。
- ID
- SP2-9
- タイトル
- Structural Insights into the Clustering and Activation of Tie2 Receptor Mediated by Tie2 Agonistic Antibody
- 発表者名
- Gyunghee Jo
- 所属
- Center for Biomolecular and Cellular Structure, Institute for Basic Science(IBS), Republic of Korea
- 肩書
- Postdoctoral Research Associates
- ID
- SP4-12
- タイトル
- Crystal structure of methylenetetrahydrofolate reductase from Sphingobium sp. SYK-6
- 発表者名
- Hongyang Yu
- 所属
- Structural Biology Research Center(SBRC), Institute of Materials Structure Science, High Energy Accelerator Organization (KEK) , and Department of Materials Structure Science, School of High Energy Accelerator Science, The Graduate University for Advanced Studies (SOKENDAI), Japan
- 肩書
- Post Doctoral Fellow
- ID
- SP4-14
- タイトル
- Exploring the structural aspects of a novel phospholipid-binding protein
- 発表者名
- Angshu Dutta
- 所属
- Department of Biosciences and Bioengineering, Indian Institute of Technology Guwahati, India
- 肩書
- PhD student
- ID
- SP4-40
- タイトル
- A new trend of protein crystallography: Polymorph analysis using hierarchical clustering of diffraction data
- 発表者名
- Hiroaki Matsuura
- 所属
- RIKEN/SPring-8 Center, Japan
- 肩書
- Postdoctoral Researcher
以下に記すポスター賞の審査員の先生方にはご協力に感謝申し上げます。また、すべての発表者のみなさま、すべての参加者のみなさまにお礼申し上げます。
有森貴夫(大阪大学)、大山拓次(山梨大学)、加藤貴之(大阪大学)、川端猛((一財)蛋白質研究奨励会)、川本晃大(大阪大学)、岸川淳一(大阪大学)、栗栖源嗣(大阪大学)、児嶋長次郎(横浜国立大学)、小手石泰康(大阪大学)、杉木俊彦(大阪大学)、杉島正一(久留米大学)、髙木淳一(大阪大学)、髙﨑寛子(大阪大学)、田中秀明(大阪大学)、藤間祥子(奈良先端科学技術大学院大学)、中川敦史(大阪大学)、長尾知生子(大阪大学)、沼本修孝(東京医科歯科大学)、橋本浩介(大阪大学)、橋本博(静岡県立大学)、平田邦生(理化学研究所)、深井周也(京都大学)、藤原敏道(大阪大学)、松木陽(大阪大学)、水口賢司(大阪大学)、宮ノ入洋平(大阪大学)、山下栄樹(大阪大学)(敬称略、五十音順)
末筆となりますが、このシンポジウム開催につきましては、一般財団法人蛋白質研究奨励会より多大なるご支援を賜りました。また、第59回日本生物物理学会サテライトイベントならびに大阪大学蛋白質研究所の国際蛋白研セミナーとして開催しました。関係各位のご配慮・ご協力に改めて感謝申し上げます。
2021年11月24日(水)オンラインにて「PDBアジア地区50周年記念シンポジウム」を開催します

PDB創設50周年を記念し、下記内容で「PDBアジア地区50周年記念シンポジウム~アジア地区構造生物学の最先端とProtein Data Bank 50年の歩み~」を開催します。
- 日程
- 2021年11月24日(水)(以下いずれも日本時間=UTC+9)
- 会場
- オンライン
- 開催趣旨・概要
-
1971年、構造生物学のコミュニティは、生体高分子の三次元構造データを保存する世界で唯一のグローバルアーカイブProtein Data Bank(PDB)を設立しました。 PDBデータ全体の約4分の1はアジアからのものです。 2000年、Protein Data Bank Japan(PDBj)は、PDBのアジア地区のハブとしてここ大阪でデータ処理を開始しました。 2001年以来、PDBjは独自のWebサイト(https://pdbj.org)を通じて、新しく開発したサービスを無料で公開し、現在もなおデータベースのさらなる充実を図っています。 2003年に米国RCSB PDBと欧州PDBeとともにworldwide PDB(wwPDB)を設立して国際連携を確かなものとし、PDBjは以降20年以上にわたりアジア地域のデータ拠点として、国際協調によるデータ登録とデータ提供の活動を推進してきました。
2021年、私たちはPDB設立50周年を祝い、特に今回は、アジア地域の構造生物学のフロンティアについて話し合いたいと思います。
全てオンラインで行われるシンポジウムは、ポスターセッションと記念シンポジウムの2部構成となっており、タイムテーブルは以下の通りとなっています。
シンポジウムは、録音/録画し、後日参加登録者限定で公開することを考えております。
また、学生・ポスドクのみなさんの優れた研究発表にはPDBjポスター賞を授与し、PDBjのホームページで公表します。みなさまのご参加を心よりお待ちしております 。
- 使用言語
- 英語
- 参加費
- 無料

- スケジュール
-
ポスターセッション(蛋白研セミナーとして実施)
10:00-10:10 主旨説明 10:10-10:50 奇数番号発表 10:50-11:00 休憩 11:00-11:40 偶数番号発表 -
記念シンポジウム(日本生物物理学会サテライトシンポジウムとして実施)
セッション1: 座長 中村春木(大阪大学) 13:30-14:15 「アジアにおけるPDB50年の歴史」栗栖源嗣(大阪大学 蛋白質研究所) 「CryoEM structure of small heat shock protein」Kyeong Kyu Kim(Sungkyunkwan University School of Medicine、韓国) セッション2: 座長 中川敦史(大阪大学 蛋白質研究所) 14:25-15:10 「Structure of the sodium-dependent phosphate transporter reveals insights into human solute carrier SLC20」Yuh-Ju Sun(国立清華大学、台湾) 「Molecular movies and beyond」岩田想(京都大学大学院 医学研究科) セッション3: 座長 藤原敏道(大阪大学 蛋白質研究所) 15:20-16:05 「NMRによる膜タンパク質の機能解明」嶋田一夫(理化学研究所 生命機能科学研究センター) 「高速撮影と高分解能を両立したクライオ電子顕微鏡による構造生物学」難波啓一(大阪大学 大学院生命機能研究科、理化学研究所 放射光科学研究センター・生命機能科学研究センター) セッション4: 座長 水口賢司(大阪大学 蛋白質研究所) 16:15-17:00 「Structural and mechanistic dissection of the Wnt signaling pathway」Wenqing Xu(国立蛋白質科学研究センター、上海科学技術大学、中国) 「A new era in structural biology: the impact of structure prediction using AI methods」Sameer Velankar(欧州生物情報学研究所、英国) 17:00-17:05 閉会挨拶 - 主催者
-
- 栗栖源嗣(大阪大学 蛋白質研究所)
- 藤原敏道(大阪大学 蛋白質研究所)
- 後援
- 一般財団法人 蛋白質研究奨励会
- 登録期間
-
- ポスター発表登録期間
- 2021年9月6日(月)~10月17日(日)日本時間23:59までただし予定発表件数(100件)に達した場合は、その時点で締め切ります。
- 参加登録期間
- 2021年9月6日(月)~11月8日(月)日本時間23:59まで
- 発表資料登録期間
- 2021年11月1日(月)~11月8日(月)日本時間23:59 11月12日(金)日本時間17:00まで
- 発表・参加登録
- こちらから登録をお願いします。
- 備考
-
- このシンポジウムは第59回日本生物物理学会年会のサテライトイベントとして実施されます。運営管理上、年会本体に参加登録されている場合も、このシンポジウムへの参加登録を行ってください。
- このイベントに参加登録されても年会本体のプログラムには参加できません。
- 取得した個人情報は、取得目的の範囲内で利用します。
- ポスター発表登録要領
-
- 発表応募条件
- 研究者の方ならどなたでも応募できます。日本生物物理学会会員である必要はありません。発表担当者として応募できるのは当ポスターセッション内で一人1演題のみです。生物物理学会年会での発表はここにカウントしません(当ポスターセッションと年会の両方で発表可能です)。
- ポスターのテーマ
-
- Protein Structure and Supramolecular complexes
- RNA/DNA molecular machines
- Application of Structural Biology
- X-ray/XFEL
- NMR
- 3DEM
- Bioinformatics/Protein Structure Prediction
- 募集件数
- 100件(予定発表件数に達した場合は、その時点で締め切ります。)
- 確認メール
- 登録が完了すると完了通知メールが自動送信されます。ポスター番号(ID)と発表資料登録手順は、別に2021年10月末までにお送りいたします。迷惑メールと認識される場合があるため、フリーのメールアドレスでの登録は避けてください。
- PDBjポスター賞
- 学生・ポスドクによる優れた研究発表に対しPDBj ポスター賞を授与します。
- 発表原稿登録について
- 発表原稿(静止画または動画)は、ご自身で日本生物物理学会のホームページよりconfitというシステムを使用して、2021年11月1日(月)~11月8日(月)日本時間23:59までに登録していただきます。この登録期間中は、登録内容を確認、修正、取消をすることができます。登録期間終了後の修正・登録については一切応じられません。
- 問い合わせ先
-
PDBアジア地区50周年記念シンポジウム事務局大阪大学蛋白質研究所 プロテインデータバンク研究室Email: db_sec@protein.osaka-u.ac.jp電話: 06-6879-4311
[wwPDB] 化学のためのオープンソース・ツールに関するワークショップ
王立化学会の化学情報・計算機応用グループは、 化学のためのオープンソース・ツールに関するワークショップシリーズの一環として、PDB50を記念して2つの無料オンラインイベントを開催します。
50年目の蛋白質構造データバンク(PDB): PDBデータの利用と理解、評価
1日目: 2021年11月16日(火)3-5pm GMT(日本時間:11月17日午前0時~11月17日午前2時)
-
History of the Protein Data Bank (PDB) and the Worldwide PDB(PDBとwwPDBの歴史)
Stephen K. Burley, RCSB Protein Data Bank (RCSB PDB) -
PDBx/mmCIF Data Standard and PDB Data Deposition with OneDep(PDBx/mmCIFデータ規格とOneDepを利用したPDBデータ登録)
Ezra Peisach, Jasmine Young, RCSB PDB -
Small Molecules in the PDB(PDBにおける低分子化合物)
Chenghua Shao, RCSB PDB -
OneDep Tutorial(OneDepチュートリアル)
Jasmine Young, Ezra Peisach, RCSB PDB
2日目: 2021年11月18日(木)3-5pm GMT(日本時間:11月19日午前0時~11月19日午前2時)
-
wwPDB Validation: Assessing the Quality of PDB structures(wwPDBの検証:PDB構造の品質評価)
John Berrisford, Protein Data Bank in Europe (PDBe) -
How to Interpret the Quality of a PDB Structure using the wwPDB validation report(wwPDB検証レポートを使ってPDB構造の品質を評価する方法)
David Armstrong, John Berrisford, Jack Turner, PDBe -
3D Visualization of PDB Data with the Mol* viewer(Mol*ビューアを使ったPDBデータの3次元構造可視化)
James Tolchard, PDBe -
Impact of AI and Future of PDB Data: Next Generation PDB Archive(AIの影響とPDBデータの将来:次世代PDBアーカイブ)
Sameer Velankar (PDBe) -
Round Table Discussion(座談会)
Sameer Velankar (PDBe), Stephen K. Burley (RCSB PDB)
ワークショップへのご参加後、以下のことが可能になります。
- 生体高分子構造を登録する際、適切にwwPDB OneDepシステムにアクセスし、使用することができます。
- PDBでは低分子データがどのように表現されているか、またアクセス方法を理解できます。
- wwPDBの検証レポートから、PDB構造の品質と正確さを評価できます。
- Mol*分子ビューアを使ってPDB構造を可視化する方法を理解できます。
- 次世代PDBアーカイブの構築に向けたwwPDBの計画を評価することができます。
化学のためのオープンソース・ツールワークショップへご参加をご希望の方は、こちらからお申込みください。
[ wwPDB News ]
[wwPDB] John Westbrook氏の訃報

インド・ハイデラバードで開催された2017年国際結晶学連合会議に参加したJohn Westbrook氏
ラトガース(Rutgers)大学特任教授であり、RCSB PDBのデータ&ソフトウェア・アーキテクトのリーダーであったJohn D. Westbrook Jr.氏(1957-2021) が2021年10月18日に逝去されました。
彼はラトガース大学の同僚や世界中の人々から非常に愛され、尊敬されていて、 辛口のウィットと、データとデータ管理のあらゆる側面について考えることへの限りない熱意で知られていました。
Johnは、構造生物学や生命科学の分野で、データ取得、検証、標準化、マイニングにおけるオントロジー、ツール、インフラの開発に携わり、 長年にわたり大きな成功を収めてきました。 彼の功績により、PDBx/mmCIFのデータ辞書とフォーマットが確立され、現在のProtein Data Bank(PDB)アーカイブの基礎となりました (wwPDB.org).
25年以上前、まだ大学院生だったJohnは、高品質で信頼性の高い構造情報をデータの利用者に確実に提供するためには、 明確に定義されたデータモデルが重要であることを認識していました。 そして、生体高分子データのためのmmCIFデータ表現の主要な設計者となりました。 シンプルな文脈自由文法(列幅の制約なし)に基づいて、データはキー・バリュー形式または表形式で表示されます。 共通のデータ項目(原子や残基の識別子など)の関係は、PDBx辞書(mmcif.wwpdb.org)に明示的に記載されています。 PDBx/mmCIFフォーマットを使用することで、ソフトウェアアプリケーションは、あらゆるPDBエントリー内の参照整合性を評価・検証することができます。 mmCIF技術の主な強みは、ソフトウェアからアクセス可能なメタデータ群が豊富で拡張性が高いことです。
現在のPDBx/mmCIF辞書には、高分子の構造決定に関わる実験や構造そのものの記述など、6,200以上の定義が含まれています。 このスキーマの最初の実装は、核酸を含むX線結晶構造のデータリソースであるNucleic Acid Databaseに使用されました。 今日では、この辞書がPDBのすべてのデータ管理を支えています。また、2014年からはPDBアーカイブのマスターフォーマットとして使用されています。 この辞書はまた、PDB内の低分子化学物質参照データの維持・配布に使用されている 化合物辞書(Chemical Component Dictionary:wwpdb.org/data/ccd)の基礎にもなっています。
2011年、国際蛋白質構造データバンク(Worldwide Protein Data Bank:wwPDB)の PDBx/mmCIF Working Groupが設立され、 主要な高分子結晶構造解析ソフトウェアツールでPDBx/mmCIFフォーマットファイルを直接使用できるようにし、 より大きな高分子構造をPDBに登録するために必要なフォーマット拡張に関する提言を提供することになりました。 これにより、リボソームのような高分子機械を単一のPDB構造として(原子座標が異なるx,y,zファイルに分散している分割エントリーではなく) 解析できるようになり、PDBアーカイブの進化の重要な一歩となりました。 2019年には、登録の際にPDBx/mmCIFフォーマットファイルの提出を必須とすることが発表されました (Adams et al. Acta Crystallographica D75, 451-454)。
Johnは、PDBx/mmCIF辞書とフォーマットの成功を確かなものにするために、コミュニティの幅広い専門家と協力して、 高分子のX線結晶構造解析実験、3次元低温電子顕微鏡実験、NMR分光実験、蛋白質や核酸の構造的特徴、 回折画像データ、タンパク質の製造・結晶化プロトコルなどの記述を包含するようフレームワークを拡張してきました。 最近では、X線自由電子(XFEL)法や、統合的またはハイブリッド法(I/HM)に対応したデータ表現の開発に力を入れています。 現在、プロトタイプのPDB-Devアーカイブ(pdb-dev.wwpdb.org)に格納されているI/HM構造は、 急速に進化している異種の実験リポジトリ間でのデータ交換に新たな課題をもたらしました。 また、PDB-DevでI/HM構造を適切に管理するためには、PDBx/mmCIFのデータ辞書を拡張して、粗視化モデルやマルチスケールモデルを含める必要がありました。 これは、クライオ電子トモグラフィーやその他のバイオイメージング手法を用いて、高分子構造を研究するために不可欠なものです。
Johnは、生物学や構造生物学の分野で相互運用やデータ統合を可能にするコミュニティのデータ標準化に幅広く貢献してきました。 彼の取り組みには、(i)複雑化する高分子構造データの記述、(ii)I/M技術を含む新しい実験手法の表現、 (iii)様々な生物医学リソースとの統合を促進するために必要な生物学的コンテキストの拡大、などが含まれます。 Johnの研究は、生体高分子の結晶学的および関連する構造データを、科学分野の主要なリソースに結びつけるための中心的な役割を果たしています。 彼の努力は120以上の査読付き出版物に記載されており、そのうちの1つは、Web of Scienceによれば21,000回以上引用されています (Berman et al. Nucleic Acids Research 28, 235-242)。 また、最も影響力のある8つの論文がInternational Tables of Crystallographyに掲載されています。
また、Johnは長年にわたり結晶学界に貢献しており、2016年には国際バイオキュレーション学会から第一回バイオキュレーションキャリア賞を受賞しました。
国際結晶学連合(IUCr)では、CIF規格維持委員会(COMCIFS)、データ回折登録委員会(DDDWG)、データ委員会(CommDat)のメンバーを務めました。 また、Acta Crystallographica Section Fの共同編集者を務めました。
米国結晶学会の長年の会員であり、データ、標準化及び計算委員会(Standards & Computing Committee)に所属していました。 また、研究データ同盟(Research Data Alliance)のメタデータ研究会(Metadata Interest Group)にも所属していました。
Johnには、妻のBonnie J. Wagner-Westbrook(Ed.D.)、 義母のJoan N. Wagner(ニュージャージー州クリントン郡区)、 Chandler Turner(バージニア州ポーツマス)、 Ann (Turner) Heyes(オーストラリア・タスマニア)、 Louise (Turner) Brown(カリフォルニア州オークランド) など、多くの親戚がいます。
2021年11月6日(土)午後2時から4時(EST)まで面会が行われ、午後4時から追悼式が行われます。 場所は、Scarponi-Bright Funeral Home, 26 Main Street, Lebanon, NJで、埋葬は非公開で行われます。
追悼の意を込めて、Capicatsまたは任意の団体にご寄付いただけます。
詳細情報は、Scarponi-Brightでご覧いただけます。
[ wwPDB News ]
[wwPDB] PDBが50周年を迎えました
PDBは1971年10月20日に学術誌Nature New BiologyのCrystallographyの欄で設立が発表されました。( Protein Data Bank Nature New Biology 233: 223 (1971) doi: 10.1038/newbio233223b0)。
現在、PDBアーカイブには、18万件以上の蛋白質、核酸、および複合体の構造が含まれており、 学生や研究者が蛋白質合成から健康や病気まで、生物医学や農業のあらゆる側面を理解するのに役立っています。 また、PDBは世界的な組織であるWorldwide PDB (wwPDB)によって運営されており、 世界中の人がPDBを自由に利用できるようになっています。
wwPDBはこの50周年を記念して、2021年を通してシンポジウムやイベントを開催しています。
wwPDB財団へのご寄付により、PDBの50年にわたる開放性、協調性、教育の精神を支援することをご検討ください。 wwPDB財団は、wwPDBのアウトリーチ活動を支援するための資金調達を目的として2010年に設立されました。 PDB50のイベント、ワークショップ、教育的出版物を支援するための資金も集めてきました。
財団は、科学、文学、慈善、教育を目的とした501(c)(3)の団体として設立されています。
[ wwPDB News ]
[wwPDB] PDBアジア地区50周年記念シンポジウムの開催
11月24日にオンラインにて「PDBアジア地区50周年記念シンポジウム~アジア地区構造生物学の最先端とProtein Data Bank 50年の歩み~」を開催します。

[ wwPDB News ]
CBI学会2021年大会にてCBI学会スポンサーセッションを開催します
2021年10月26日(火)、CBI学会2021年大会にて下記の通りCBI学会スポンサーセッションを開催します。
- 日時
- 2021年10月26日(火) 15:00~16:30
- 開催方法
- オンライン
- プログラム
- スポンサーセッション(SS-12)
- 参加登録
- 学会の事前参加登録(参加費がかかります)が必要。( 申し込み )
- 講演言語
- 日本語/英語
- 演題・演者
-
-
「コロナ禍におけるPDBの活動と構造データの品質向上およびデータ検証の取り組みについて」
栗栖 源嗣(大阪大学蛋白質研究所) -
「wwPDBがデータ検証で用いるCSDを活用したBUSTERによる構造精密化事例について」
中川 敦史(大阪大学蛋白質研究所) -
「Data integration to facilitate drug discovery」
Francis L. Atkinson, Cheminformatics Data Scientist, Cambridge Crystallographic Data Centre (CCDC)
-
「コロナ禍におけるPDBの活動と構造データの品質向上およびデータ検証の取り組みについて」
- その他資料
- 備考
-
- 今回のセミナーは化学情報協会様との協同出展となります。
- アンケート回答者のうち抽選で30名様に、PDBj設立20周年を記念して作製したトートバッグをプレゼントします。

第59回日本生物物理学会年会にてバイオフィジックスセミナーを開催します
2021年11月26日(金)、第59回日本生物物理学会年会にて下記の通りバイオフィジックスセミナーを開催します。
- 日時
- 2021年11月26日(金) 12:00~12:50
- 開催方法
- オンライン
- プログラム名
- バイオフィジックスセミナー 部屋番号:ch2
- 参加登録
- 学会の事前参加登録(参加費がかかります)が必要。( 申し込み )
- 講演言語
- 英語
- 演題・演者
-
-
PDB activities under ongoing Covid-19 situation, and up-to-date information on our quality improvement and data validation
Genji Kurisu,(Institute for Protein Research, Osaka University) -
PDBj tools and services for analyzing and visualizing structural data.
Gert-Jan Bekker,(Institute for Protein Research, Osaka University) -
Services and databases for 3D electron microscopy data by PDBj and wwPDB
Hirofumi Suzuki, Waseda University
-
PDB activities under ongoing Covid-19 situation, and up-to-date information on our quality improvement and data validation
- その他資料
- 備考
-
- アンケート回答者のうち抽選で30名様に、PDBj設立20周年を記念して作製したトートバッグ(写真別添)をプレゼントします。

[wwPDB] 10,000件の区切りのPDBデータを処理した研究員
wwPDBのバイオキュレーター Dr. Sutapa Ghosh博士とMonica Sekharan博士が処理したPDBエントリーが10,000件に到達しました。おめでとうございます。 二人は、このマイルストーンに到達した2番目・3番目のバイオキュレーターとなります。 2021年4月に見学有美子さんが初めてこのマイルストーンに到達しました( wwPDBニュース / PDBjニュース )。
Ghosh博士はカルカッタ大学で構造生物学の博士号を取得し、企業で構造に基づいた薬剤設計に携わった後、PDBメンバーに加わりました。 Sekharan博士は、ワシントン大学で生物化学の博士号を取得し、核磁気共鳴分光法を専門としています。 15年間のPDBでのキャリアの中で、多くの登録者が、彼らの正確で包括的なデータ分析と表現における専門的なスキルを信頼しています。 彼らの深い科学的知識、深いデータキュレーションの専門知識、そして卓越性への取り組みは、科学界のための高品質なデータアーカイブの構築に貢献しました。 私たちは、Ghosh博士とSekharan博士のこの素晴らしい成果を祝福するとともに、彼らの今後の活躍を期待しています。

RCSB PDB Biocurators Dr. Sutapa Ghosh and Dr. Monica Sekharan
[ wwPDB News ]
BINDS-PDBj講習会「PDBから見てわかるタンパク質の最新研究」(開催:2021-09-30)

- 講習会名
- BINDS-PDBj講習会「PDBから見てわかるタンパク質の最新研究」
- 日時
- 2021年9月30日(木) 13:00 ~ 17:30
- 開催趣旨
- 生体高分子の立体構造を決定する構造生物学は、現代の生物学にとって不可欠なアプローチです。蛋白質構造データバンクPDBには、多くの立体構造データが登録されており、そこから多くの知見を引き出すことができます。しかしながら、立体構造は大変複雑であり、分子ビューアの操作も初心者にはなかなか厄介です。そこで、本講習会では、3人の研究者の方をお招きして、最新の研究成果を発表していただくと同時に、その研究で使われた立体構造データを、受講者の皆さんに、分子ビューアソフトを使って、よく観察していただき、より深く研究内容を理解していただくことを目指します。分子ビューアは、PyMOL、UCSF Chimera、Molmilを用いる予定です。設定や使い方は、PDBjのスタッフがサポートいたします。構造生物学に関心のある方の積極的なご参加をお待ちしております。
- 主催
- 創薬等先端技術支援基盤プラットフォーム(BINDS), 日本蛋白質構造データバンク(PDBj)
- 開催方法
- Zoomのミーティングによるリモート開催
- 定員
- 100名程度
- 参加費
- 無料
- 参加申込
- 参加申込は、9月23日(木)で締め切りました。 参加登録者には、9月24日(木)に、Zoomの接続アドレスをメールで送信いたしました。登録済みにも関わらず、Zoomのアドレスを未受信の方は、
- [PDBjのお問い合わせページ]からご連絡をお願いいたします。
- 事前インストールが必要なソフトウエア
- 分子ビューアとして、PyMOLとUCSF Chimeraのインストールを事前に行っておいてください。詳細な手続きは別項に示します。また、できるだけマウスを用意してください。
- 講演言語
- 日本語
- お問い合わせ
- [PDBjのお問い合わせページ]をご利用ください。
- プログラム
- 必要なソフトウエアのインストール方法
- PyMOLのインストール方法
Windows, macOS, Linux版があります。インストール作業以外にライセンスファイルの取得が必要ですが、今回の講習では、ライセンス無し版か、Student/Teacher無償ライセンス版で十分です。インストールはWindows版のインストール手続きを参考にしてください。- UCSF Chimeraのインストール方法
- https://www.cgl.ucsf.edu/chimera/download.htmlのサイトから、ダウンロードしインストールを行ってください。 Windows, MacOS X, Linux版があります。最新版は1.15です。非商用目的での使用のみ無料で用いることができます。 Windows版のインストール手続き、および Mac版のインストール手続きを参考にしてください。 ※企業の方でUCSF Chimeraのインストールが難しい場合は、代わりにPyMOLをインストールしておいてください。
- Molmilは標準的なブラウザであれば動作するはずなので、特別なインストールは不要です。ただし、できるだけブラウザを最新版に更新しておくようにしてください。
- 講演概要
- 「新型コロナウイルスを標的とした高親和性ACE2の開発とその構造解析」
有森貴夫(大阪大学蛋白質研究所 准教授)
2019年末から流行が始まった新型コロナウイルス感染症(COVID-19)は,変異株の出現の影響もあり未だに終息の兆しが見えない.新型コロナウイルスは,その表面にあるスパイクタンパク質のレセプター結合ドメイン(RBD)が,ヒトの細胞表面にあるアンジオテンシン変換酵素II(ACE2)に結合することで感染が開始される.そこで我々は,感染阻害薬としての応用を目指し,ACE2に変異を導入することでRBDに対する親和性を約100倍まで向上させた改変ACE2を開発した.この改変ACE2は強力なウイルス中和活性を示し,COVID-19感染モデルハムスターにおいても治療効果が認められた.さらに,改変ACE2-RBD複合体の結晶構造解析の結果,改変ACE2は野生型と同じインターフェースを介してRBDに結合することが確認できた.すなわち,改変ACE2に結合しない変異ウイルスは細胞表面のACE2にも結合できず感染力を失う可能性が高いため,この改変ACE2に対する耐性株は出現しにくいと考えられる.さらに結晶構造からは,変異導入部位の側鎖が親和性向上に直接的に貢献していることが示された.本講習会では,PyMOLを用いて改変ACE2と野生型ACE2の結晶構造を比較する. Higuchi, Y., Suzuki, T., Arimori, T. et al. Engineered ACE2 receptor therapy overcomes mutational escape of SARS-CoV-2. Nat Commun 12, 3802 (2021). - 「効率的なデータ収集を目指した新たなクライオ電子顕微鏡用グリッドの開発」
藤田 純三 (大阪大学大学院生命機能研究科 日本電子YOKOGUSHI協働研究所 特任助教)
クライオ電子顕微鏡による単粒子解析ではタンパク質等の試料を非晶質氷中に包埋する手法が一般的であるが、気液界面における試料の変性や粒子の局在、配向の偏り、ブロッティングによる粒子数の減少といった現象が問題となる。改善策の一つとしてグラフェン等のごく薄い支持膜をグリッド表面に張り付けるという方法が知られているが、グラフェンは疎水性でありそのままではタンパク質が吸着しにくいため、グラフェンに酸素官能基を導入した酸化グラフェンが支持膜として多用されている。我々はグラフェングリッドを二酸化塩素を用いた光酸化法により酸化し、さらに化学修飾を施すことでタンパク質を強く吸着する新たなグリッド(EG-grid)を開発した。本発表ではEG-gridによる構造解析例およびそこから見えてきたデータ収集の効率化や粒子の配向性の変化といったメリットを紹介するとともに、得られた密度マップについて受講者の方々に実際に触れて頂き、クライオ電子顕微鏡による構造解析に少しでも慣れ親しんで頂けるような時間を設けたい。
- 「Gating-modifier toxin APETx1による電位依存性カリウムイオンチャネルhERG阻害機構の解析」
大澤匡範(慶應義塾大学 薬学部, 教授)
ヒト心臓に発現する電位依存性K+チャネルhERGは膜電位に応じて構造変化し、K+の膜透過を制御することで、心臓の正常な機能に関わる。Gating modifier toxinであるAPETx1は、hERGの静止状態における電位センサードメイン(VSD)に結合し、hERGを阻害することが知られている。しかしながら、これまでに、膜電位存在下で生じるhERGの静止状態の立体構造は未解明であり、APETx1のhERG結合様式は不明である。そこで本研究ではAPETx1およびhERGの変異体を用いた電気生理学的解析により、APETx1によるhERG阻害機構の原子レベルでの解明を目的とした。 我々は、これまで報告の無かったリコンビナントでのAPETx1の調製法を確立し、NMRによる立体構造解析および電気生理解析を行い、リコンビナントAPETx1が天然体と同等の構造とhERG阻害活性を有することを確認した。そこで、APETx1の1残基変異体15種を調製し、パッチクランプ法により、APETx1の変異体添加時のhERG電流を測定した。その結果、hERG阻害にAPETx1の4残基(F15, Y32, F33, L34)が重要な役割を果たすことが分かった。これらの残基は、このAPETx1の分子表面上で局在しており、hERGとの直接の結合に関わることが示唆された。 さらに、hERG変異体に対するAPETx1の阻害活性を電気生理学的に解析した。アフリカツメガエルの卵母細胞にhERG野生型および変異体21種を発現させ、二本刺し膜電位固定法を行い、APETx1の添加前後でhERG電流を測定した。その結果、hERG変異体21種のうち、5種でAPETx1の阻害活性が有意に減少した。hERG阻害活性が減少した変異部位は、既知のhERG活性化状態の構造上では互いに離れた位置に存在した。 そこで、hERG-VSDの電位センサーであるS4ヘリックスの位置をシフトさせた複数の立体構造をモデリングし、APETx1とのdocking simulationを行ったところ、ヘリックス1~2ターン分S4を細胞内側にシフトしたモデルで、APETx1によるhERG阻害が変調を受けたhERG残基が近接しAPETx1との相補的表面を形成した。このdocking modelは、今回の変異体を用いた一連の電気生理学実験結果を矛盾なく説明するものであることから、APETx1のhERG阻害様式を反映していることが示唆された。 Matsumura, K., Shimomura, T., Kubo, Y. et al. Mechanism of hERG inhibition by gating-modifier toxin, APETx1, deduced by functional characterization. BMC Mol and Cell Biol 22, 3 (2021).
| 時間 | 題目・演者 | 講義資料・講義データなど | |
| 13:00-13:15 | 「PDBの最近の動向」栗栖 源嗣(大阪大・蛋白研) | 講義資料「PDBの最近の動向」 | [講演動画(YouTube)] |
| 13:15-14:15 | 「PDBデータの使い方・分子ビューワの使い方の基礎」川端 猛(蛋白質研究奨励会・大阪大) | 講義資料「PDBデータの使い方・分子ビューワの使い方の基礎」 | [講演動画(YouTube)] |
| 14:15-15:15 | 「新型コロナウイルスを標的とした高親和性ACE2の開発とその構造解析」有森 貴夫(大阪大・蛋白研)[PyMOLを使用] |
講義資料「PyMOLの使い方」 PyMOLのセッションファイル"7dmu_6m0j.pse" |
|
| 15:15-15:30 | 休憩 | ||
| 15:30-16:30 | 「効率的なデータ収集を目指した新たなクライオ電子顕微鏡用グリッドの開発」藤田 純三 (大阪大・生命機能) [UCSF Chimeraを使用] |
講義資料「UCSF Chimeraを使って電顕マップを見てみよう」 |
|
| 16:30-17:30 | 「Gating-modifier toxin APETx1による電位依存性カリウムイオンチャネルhERG阻害機構の解析」大澤 匡範(慶應大・薬)[Molmilを使用] |
Molmilを用いた分子表示のページ (https://numon.pdbj.org/binds_workshop_202109/osawa/)
講義資料「Molmilで立体構造を見る」 |
[wwPDB] PDBx/mmCIFデータファイルに主任研究者(PI)の連絡先情報が追加されます

PI name, email, and ORCiD ID will be publicly available in PDBx/mmCIF data files starting September 24, 2021
wwPDBは、世界中の研究、教育、創薬の支援を続けています。 PDBデータを誰もが無料で利用できることは、抗コロナウイルス薬、ワクチン、中和抗体などの構造誘導型の発見・開発において、研究者の助けとなっています。 研究者がPDBに存在するよく似た構造など、既存の構造を解析する場合、一次引用文献が公開されていなければPDBエントリーからだけでは得られないような追加情報が必要になることがよくあります。 特に、あるエントリーに関連する一次引用文献がない場合、連絡先情報を得ることはできません。
生体高分子に関する国際結晶学会委員会(IUCr Commission on Biological Macromolecules)と データに関する国際結晶学会委員会(IUCr Committee on Data)の勧告に従い、2021年9月24日から初回の登録または再登録の際に、 エントリーPIの名前、メールアドレス、ORCiD IDを公開することになりました。 これにより、その日に公開されたすべてのPDBエントリーの著者と、簡単に連絡が取れるようになります。 今回の公開は、科学雑誌が対応する著者情報を提供する標準的な方法にPDBを合わせるものです。
上記の「PDB利用規約」に同意した日付は、OneDepシステム内に記録されます。 登録を行った責任者は、公開されているPDBx/mmCIFファイルにPIの名前、メールアドレス、 ORCiDを含めるようにポリシーが変更されたことをエントリーPIに知らせる必要があります。
[ wwPDB News ]
[wwPDB] 化合物の定義ファイルとアーカイブの一覧ファイルへのアクセスを改善しました
 化合物辞書のエントリー別の定義ファイルとアーカイブ内のファイル一覧をダウンロードできるようになりました
化合物辞書のエントリー別の定義ファイルとアーカイブ内のファイル一覧をダウンロードできるようになりました
低分子の定義ファイルへのアクセスを改善 (新しいディレクトリ:/pdb/refdata/)
化合物辞書(Chemical Component Dictionary, CCD)とBIRD辞書 (Biologically Interest molecule Reference Dictionary)の 個々の定義ファイルが、PDBアーカイブ内の新しいFTPツリーからアクセスできるようになりました。 ユーザーからの要望に応えて、これら個別のCCDとBIRDのエントリーファイルは、 それぞれ /pdb/refdata/chem_comp/ と /pdb/refdata/bird/ に、最後の文字のハッシュをサブディレクトリとして配置されました。
例えば、化合物コードD8CとBIRD PRD_001068の定義ファイルは、以下の場所に保管されます。
- /pdb/refdata/chem_comp/C/D8C/D8C.cif
- /pdb/refdata/bird/prd/8/PRD_001068.cif
PDBアーカイブが保有する情報へのアクセスを改善 (新しいディレクトリ:/pdb/holdings/)
新しい一覧データファイルを参照すると、アーカイブ内のデータの概要を簡単に確認できます。 これらのファイルは、拡張が容易なJSON形式で記述され、新しいFTP内のディレクトリ/pdb/holdings/の下に配置されています。
一覧ファイルには以下の種類があります。
- all_removed_entries.json.gz ・・・削除されたPDBエントリー(廃止、モデル)、登録者、エントリーのタイトル、公開日、廃止された日、そして(もしあれば)置換後のPDB IDが記載されたリスト
- current_file_holdings.json.gz ・・・公開済みPDBエントリーと、PDBコアアーカイブ内の各エントリーに存在するファイル種別 (例:座標データ、実験データ、検証レポート等)のリスト
- obsolete_structures_last_modified_dates.json.gz ・・・廃止されたPDBエントリーとPDBx/mmCIFファイルの最終更新日時のリスト
- refdata_id_list.json.gz ・・・公開済みの化学物質参照エントリー、内容種別(例えば、化合物、BIRD)、参照ファイルの最終更新日時のリスト
- released_structures_last_modified_dates.json.gz ・・・公開済みのPDBエントリーとPDBx/mmCIFファイルの最終更新日時のリスト
- unreleased_entries.json.gz ・・・未公開のPDBエントリーとステータス、登録日、事前公開配列情報のリスト
これまで、/pdb/derived_data/配下に提供されてきた一覧(インデックス)ファイルは、当面は更新されますが、 最終的にはPDBアーカイブから削除されます。ユーザーの皆様は、新しい一覧ファイルをご利用ください。
[ wwPDB News ]
[wwPDB] PDBが標準的なクリエイティブ・コモンズ・オープンソース・ライセンスを採用
wwPDBは、PDBコア・アーカイブをFAIR原則(検索、アクセス、相互運用、再利用可能)に則った公共財として管理しています。 FAIRの目的に賛同し、wwPDBは過去のデータアクセスライセンスを、クリエイティブ・コモンズの 標準的なオープンソースライセンスである CC0 1.0 全世界 (CC0 1.0)パブリック・ドメイン提供に変更しました。
新しいCC0ライセンスは、従来のライセンスと同様にオープンアクセスを提供します。 PDBデータは、商用ユーザーを含むすべてのPDBユーザーが自由にご利用頂けます。
2021年のwwPDBの定款(Charter)と 利用規約は、新しいライセンスを反映して更新されました。
PDBデータの利用者は、可能な限りPDB構造データの原著者を表示するようにしてください。 (wwPDBサイト / PDBjサイト)
[ wwPDB News ]
「Webサイエンスフェスタ2021」に「新型コロナウィルスの立体構造」の動画とWEBページを出展しています
2021年8月21日から1年間開催されている「第30回青少年のための科学の祭典 大阪大会 2021 Webサイエンスフェスタ2021」に、PDBjから動画を出展しています。
今回は、「生命を支えるタンパク質のかたち -新型コロナウィルスの形-」と題して、新型コロナウィルス関連の立体構造をMolmilで表示するWEBページを作成し、それを説明する動画を一般公開しています。
コロナのウィルス粒子の形、スパイクタンパク質、受容体や抗体との結合構造、デルタ株などの変異部位、抗体カクテル療法、レムデシビルなどの治療薬、などに関する立体構造をまとめ、一般の方向けに、できるだけわかりやすく説明しています。
興味がある方は是非ご覧ください。
WEBページや動画のアドレスは、以下の通りです。
WEBページ:https://numon.pdbj.org/covid19/
動画:サイエンスフェスタ用(5分)
スパイク中心(8分)
全ページの説明(14分)
|

|
PDBjウェブサーバの移行に伴いPDBjウェブサービスがリニューアルされます
- PDB詳細検索とMineトップページのクイック検索の統合
- mmCIFデータや我々独自のpdbjplusデータを抽出するのにこれまであまり馴染みのなかったSQL検索をもっと簡単にご利用していただけるようにように、グラフィックインターフェースのSQL検索である新しいPDBj RDB検索サービスがご利用いただけるようになります。
- CIF-QueryとXML-Queryサービスは廃止されますが、その機能は私たちの検索サービスの中の"結果をダウンロード"機能に統合されます。"結果をダウンロード"機能では、RDBの中から検索HITしたどんなカテゴリ/アイテムもmmCIFやmmJSON, JSONファイルなどお好みのフォーマットを指定して抽出することができます。
- 新しく整備したPDBjデータアーカイブを下記のサイトからもご利用いただけます。https://data.pdbjbk1.pdbj.org/ (PDBj FTPアーカイブサイトもこれまで通りご利用いただけます)
- PDBMLplusフォーマットの提供は廃止予定です。代わりにmmJSONフォーマットが下記データアーカイブサイトからご利用頂けます。https://data.pdbjbk1.pdbj.org/pdbjplus/data/cc/mmjson-plus/ このmmJSONフォーマットファイルは、新PDBj Mineの各PDBエントリーページからもダウンロード頂けます。
- Sequence Navigator/Sequence Neighborインターフェースもリニューアルされ、superposition機能は従来のPDBフォーマットファイルを使ったサーバサイドからではなく、mmJSONファイルを使ったMolmilによりクライアントサイドで提供されるようになります。
- PDBj MineのPDBエントリーページのMolmilインターフェース (AU/BU) もニューアルされます。
- 電子密度マップサービスのMolmilインターフェースもリニューアルされます。
- eF-siteサービスのMolmilインターフェースもリニューアルされます。
- Promode Elasticサービスのインターフェースもリニューアルされます。
- PDBj RESTサービスもリニューアルされます。
- PDBjによる付加データがさらにpdbjplusデータ(アーカイブやファイル)に追加されます。
- これまでのMine RDBの週次差分ファイルの提供は廃止され、代わりに PDBjの更新サービスに置き換わります。
PDBjウェブサーバ移行によるサービスリニューアルについて、何かご質問等ございましたら、PDBjお問い合わせページからお問い合わせください。
[wwPDB] wwPDBはEMDBデータモデルのバージョン3へ移行します
2022年2月9日より、wwPDB EMDBコアアーカイブは、データモデルのバージョン3のみをサポートし、バージョン1.9.6のヘッダファイルは廃止します。 この変更に伴い、アーカイブでのファイル提供に関していくつかの変更がありますので、その概要を説明します。
2015年にOneDepが開始されて以来、EMDBコアアーカイブ (EMBL-EBI: ftp.ebi.ac.uk/pub/databases/emdb/, PDBj: ftp.pdbj.org/pub/emdb/ 又は data.pdbjbk1.pdbj.org/pub/emdb/, wwPDB mirror site: ftp.wwpdb.org/pub/emdb/) では、2つのバージョンのデータモデルを並行して管理し、また、各エントリーのヘッダファイルも2つのバージョンを提供してきました。 現在の公式EMDBデータモデルはバージョン1.9.6ですが、2015年には、EMDBエントリーに関するメタデータのより豊かな表現を容易にするバージョン3が導入されました。
このたびバージョン3が最終決定されたため、EMDBは公式データモデルのバージョンをv1.9.6からv3に変更します。これに伴い、2022年2月9日付でEMDBコア・アーカイブに3つの変更が加えられます。
- 現在のEMDBデータモデルは、<EMDBコアアーカイブ>/doc/XML-schemas/emdb-schemas/current/ に置かれたemdb.xsdというファイルの中のXSDスキーマで定義されていますが、これが最新のv3データモデル(現在はv3.0.2.6)に変更されます。
- EMDBコアアーカイブの各エントリの公式ヘッダファイルとなっているXMLファイルemd-xxxxx.xmlは、最新版のv3データモデルの構造に沿ったv3 XMLファイルになります。
- EMDBコアアーカイブでは、すべてのエントリに対してv1.9.6のヘッダファイルの提供を終了します。
現在、EMDBコアアーカイブの場所 <EMDBコアアーカイブ>/structures/EMD-xxxxx/header/にあるEMDBエントリーに対して、 以下のヘッダファイルが提供されています。
- 公式ヘッダファイル: emd-xxxxx.xml
- データモデル v3のヘッダファイル: emd-xxxxx-v30.xml
- データモデル v1.9.6のヘッダファイル: emd-xxxxx-v19.xml
ここで、現在のemd-xxxxx.xmlは、EMDBデータモデルのv1.9.6に準拠したヘッダファイルemd-xxxxx-v19.xmlの複製です。
2022年2月9日以降は、どのエントリーに対しても、提供されるヘッダファイルは以下の種類のみになります。
- 公式ヘッダファイル: emd-xxxxx.xml
- データモデル v3のヘッダファイル: emd-xxxxx-v30.xml
ここで、emd-xxxxx.xmlは、EMDBデータモデルの v3に準拠したヘッダファイルemd-xxxxx-v30.xmlの複製です。
ご質問などございましたら、info@wwpdb.org.まで英語でおたずねください。
[ wwPDB News ]
PDBj FTP Archiveの整備について
現在、PDBjでは、FTPアーカイブサービスとして、ftp.pdbj.org および ftp-versioned.pdbj.org に加え、5/27にリニューアルされたPDBj Mineサービスとともに公開されました、data.pdbjbk1.pdbj.org を合わせた3つのアーカイブサイトを運営しております。
PDBjでは、これらのアーカイブサイトを以下のように整理し、9/1から整備したものに切り替えます。
| (1) ftp.pdbj.org | wwPDB共通のアーカイブ pub (pdb/emdb) のみを保持 (wwPDB, rcsbのFTPサイトと同様) |
|---|---|
| (2) ftp-versioned.pdbj.org | これまで通り、versioning化されたPDBアーカイブのみを保持 |
| (3) data.pdbjbk1.pdbj.org | (1) (2) を含み、かつ PDBjで生成している付加データ群、PDBjで開発した2次データベース群の各データを保持 |
現在、(1)の ftp.pdbj.org から "pub (pdb/emdb) " 以外のデータをdownloadしてご利用されている方々は、data.pdbjbk1.pdbj.org のご利用への移行対応をお願いいたします。
"ftp.pdbj.org/pub(pdb/emdb)" をご利用の方には変更ありませんので影響はございません。
PDBjの新しいアーカイブサイトの詳細につきましては、PDBjデータサイト・FTPサイトの詳細 のヘルプページもご覧ください。
また、9/1の切り替えに伴い、ftp.pdbj.orgから公開しておりました、PDBMLadd (PDBjによる付加情報XMLファイル群) の提供は終了します。
今後は、既に"data.pdbjbk1.pdbj.org"から公開しております、より使いやすいフォーマット群 (JSONファイルなど)をぜひお試し頂き、PDBj付加データ等のご利用は、PDBjの新データアーカイブサイトに切り替えをお願いいたします。
PDBj FTPサイトのデータ整備についてのご質問等ございましたら、PDBjお問い合わせページからお願いします。
[wwPDB] 拡張した8桁のPDB IDとPDB DOIがPDBx/mmCIFファイルに含まれるようになりました

学術雑誌、PDBユーザー、関連ソフトウェア開発者の皆様は、ご自身の開発されたソフトウェアのコードを見直し、 PDBx/mmCIFファイル内のPDB IDのフォーマット変更とPDB DOIの組み込みに向けて準備を開始してください。
wwPDBは、PDBx/mmCIFワーキンググループと協力して、 PDBおよびCCD(化合物辞書、Chemical Component Dictionary)のIDについて、 コードの文字数を将来的に拡張する計画を立てました(過去のニュース)。 これらの拡張IDを含むエントリーは、従来のPDBファイルフォーマットではサポートされません。
以前お知らせしたように、wwPDBではPDB IDの長さを8文字に拡張し、先頭に「PDB」を付けています(例:pdb_00001abc)。
各PDB IDには対応するデジタルオブジェクト識別子(DOI)があり、これは多くの学術誌で原稿を投稿する際に必要とされ、 論文に記載されることがあります。
登録者が学術誌に情報を提供し易いように、OneDepは登録確認ページにPDB IDとDOIを表示するようにしました。
既存の4文字のPDB IDに加えて、拡張PDB IDとそれに対応するPDB DOIがPDBx/mmCIFフォーマットのファイルに含まれるようになりました。 当初は、新規公開されたPDBエントリーと更新されたPDBエントリーにのみ含まれ、後にアーカイブ全体で全てのエントリーに対して更新を行う予定です。
新しいアクセション情報はPDBx/mmCIFの _database_2 カテゴリに追加されます。
例えば、PDBエントリー「1ABC」は、拡張PDB ID(pdb_00001abc)と 対応するPDB DOI(10.2210/pdb1abc/pdb)を持ちます。
loop_ _database_2.database_id _database_2.database_code _database_2.pdbx_database_accession _database_2.pdbx_DOI PDB 1abc pdb_00001abc 10.2210/pdb1abc/pdb WWPDB D_1xxxxxxxxx ? ?
学術誌、ユーザー、ソフトウェア開発者の皆様は、コードを見直し、関連する制限を取り除くようお願いいたします。
[ wwPDB News ]
[wwPDB] PDBx/mmCIFデータファイルに主任研究者(PI)の連絡先情報が追加されます
wwPDBは、世界中の研究、教育、創薬の支援を続けています。 PDBデータを誰もが無料で利用できることは、抗コロナウイルス薬、ワクチン、中和抗体などの構造誘導型の発見・開発において、研究者の助けとなっています。 研究者がPDBに存在するよく似た構造など、既存の構造を解析する場合、一次引用文献が公開されていなければPDBエントリーからだけでは得られないような追加情報が必要になることがよくあります。 特に、あるエントリーに関連する一次引用文献がない場合、連絡先情報を得ることはできません。
生体高分子に関する国際結晶学会委員会(IUCr Commission on Biological Macromolecules)と データに関する国際結晶学会委員会(IUCr Committee on Data)の勧告に従い、2021年9月24日から初回の登録または再登録の際に、 エントリーPIの名前、メールアドレス、ORCiD IDを公開することになりました。 これにより、その日に公開されたすべてのPDBエントリーの著者と、簡単に連絡が取れるようになります。 今回の公開は、科学雑誌が対応する著者情報を提供する標準的な方法にPDBを合わせるものです。
上記の「PDB利用規約」に同意した日付は、OneDepシステム内に記録されます。 登録を行った責任者は、公開されているPDBx/mmCIFファイルにPIの名前、メールアドレス、 ORCiDを含めるようにポリシーが変更されたことをエントリーPIに知らせる必要があります。

[ wwPDB News ]
[wwPDB] PDB50会議のポスター賞受賞者
 ポスター賞受賞者
ポスター賞受賞者
PDB50開会のミーティングでは、275件のポスターが発表されました (各ポスターの要旨:1日目、 2日目)。 このうち209件の発表がポスター賞の対象となりました。
- 高校生最優秀賞: Nicholas Mamisashvili, Shelter Island High School, Molecular Dynamics Simulation of 6PEY.pdb a Novel Mutation in the Enzyme Methylenetetrahydrofolate Reductase
- 大学生最優秀賞: Ijeoma Okoye, Vassar College, X-ray and Antioxidant Determination of Butein and 2’,4’-dihydroxy-3,4-dimethoxychalcone to Examine their Antimalarial Activity by Binding to Falcipain-2
- 大学院生最優秀賞: Daniel Sultanov, New York University, Mining for functional ribosomal variants in Saccharomyces cerevisiae
- 博士研究員最優秀賞: Seda Kocaman, National Institute of Environmental Health Sciences, Different ATP binding states of the essential AAA (ATPases Associated with various Activities)-ATPase Rix7 facilitate substrate translocation in ribosome biogenesis
ポスター賞の審査員に感謝します。
- BMRB: Hamid Eghbalnia
- PDBe: Genevieve Evans, John Berrisford
- 日本蛋白質構造データバンク(PDBj): 栗栖 源嗣 (Genji Kurisu)
- UConn: Bing Hao, Irina Bezsonova, Melissa Caimano
- ナポリ大学: Luigi Di Costanzo
- RCSB: Brian Hudson, Brinda Vallat, Cathy Lawson, Chenghua Shao,
- David Goodsell, Dennis Piehl, Ezra Peisach, Helen Berman, Irina Persikova, Joan Segura, Justin Flatt, Rachel Kramer Green, Stephen Burley, Yuhe Liang, Zukang Feng
- ロチェスター工科大学(RIT): Paul Craig
wwPDBは、2021年を通して、シンポジウムや資料等を用意してPDB50周年を記念行事を進めていく予定です。
[ wwPDB News ]
第21回日本蛋白質科学会年会にてランチタイムセミナーを開催します
2021年6月16日(水)、第21回日本蛋白質科学会年会にて下記の通りランチタイムセミナーを開催します。
- 日時
- 2021年6月16日(水) 12:40 ~ 13:30
- 開催方法
- オンライン
- 参加登録
- 学会の事前参加登録(参加費がかかります)が必要。前期登録は5月11日まで。会期直前及び会期中の後期登録の予定あり。
- 講演言語
- 日本語
- 演題・演者
-
-
「PDBjの最近の活動とwwPDBの今後の活動方針について」(Activity report of PDBj and scheduled activity plan of wwPDB)
栗栖 源嗣(大阪大学蛋白質研究所) -
「PDBjが提供する、構造データを分析、編集・登録するためのツールやサービス」(PDBj tools and services for analyzing, editing and registering
structural data)
Gert-Jan Bekker(大阪大学蛋白質研究所)
-
「PDBjの最近の活動とwwPDBの今後の活動方針について」(Activity report of PDBj and scheduled activity plan of wwPDB)
- その他資料
- 備考
- セミナー参加者向けの講演アンケート回答者、先着30名にPDBj設立20周年を記念して作製したトートバッグをプレゼントします。

6月24日(木)・25日(金)、CiCLE単粒子解析リモート講習会を開催します
- 開催趣旨
- 近年のクライオ電子顕微鏡の単粒子解析の進展は著しく、X線結晶解析に迫る分解能の生体高分子像が得られるようになってきています。 本リモート講習会では、電顕の初心者の方を対象に、電子顕微鏡の原理と単粒子解析の原理の講義、画像処理、三次元再構成、 原子モデリングについての計算機実習を行います。遠隔による講習会のため、本格的な実習はできませんが、動画や画像データの配布を行うことで、 できるだけ体感的な講習を行います。今回はcryoSPARCを用いて、画像処理・3次元再構成を行います。
- 主催
- 医療研究開発革新基盤創成事業(CiCLE)「タンパク質構造解析のハイスループット化へ向けた装置開発」
- 日程
- 2021年 6月24日(木) 13:00-18:00、25日(金) 13:00-18:00
- 開催方法
- ZOOMによるミーティング形式での開催
※画像処理・3次元再構成の計算にはGPU付き計算機が必要ですが、リモートの参加者全員にその環境を準備するのは大変です。 そこで、今回は数名のアシスタントの方だけにGPUマシンでリアルタイムにcryoSPARCを操作してもらい、リモートの参加者の方はその様子をリモートでご覧になりながら、 別途配布する計算途中の画像ファイルをご自分のPCで検討していただく形式にします。原子モデリングについては、一般的なパソコンで十分可能です。 事前に必要なソフト(UCSF ChimeraとCoot)をインストールしていただき、リモート参加者の方全員に実習を行っていただきます。 - 定員
- 100名程度 (先着順)
- 参加費
- 無料 (事前登録必要)
- 参加申込
-
参加定員に達しましたので、参加申込を締め切らせていただきます。
6/18(金)の夕方に、参加登録者全員宛に、Zoomの接続方法をメールで送付いたしました。 登録者の方でZoomの接続方法が届いていない方はPDBjのお問い合わせページからご連絡ください。 - ソフトウエアの事前インストールのお願い
-
事前に以下のソフトをお使いのPCにインストールしておいてください。
- Comment Screen
- UCSF Chimera
- Coot
※UCSFChimeraは非商用(non-commercial)目的の場合のみ無料で使えるソフトです。商用目的の場合は有料となります。企業の方でUCSF Chimeraのインストールが難しい場合は、代わりに PyMOLをインストールしておいてください。 - 講義・講習の担当
- 加藤 貴之(大阪大学 蛋白質研究所)、田中 秀明(大阪大学 蛋白質研究所)、牧野 文信(日本電子・大阪大学招へい准教授)、岸川淳一(大阪大学 蛋白質研究所), 川端 猛(蛋白質研究奨励会・大阪大学招へい准教授)
- お問い合わせ
- PDBjのお問い合わせページ をご利用ください。

<スケジュール>
2021年6月24日(木)| 13:00 - 15:00 | 講義「単粒子解析の原理」加藤 貴之 |
[講義資料:分解能の話] |
|
| 15:10 - 16:00 | 講義「日本電子の低温電子顕微鏡の紹介」牧野 文信 | ||
| 16:10 - 18:00 | 実習「cryoSPARCによる電顕画像処理」川端 猛、加藤 貴之 |
[講習資料:CryoSPARC演習] [講義資料:Blob pickerを使った場合] [パソコン用の講習会データ (cryoSPARC_INX-6_results_202106.zip) (67MB)] |
[講習会の様子の動画] |
| 13:00 - 14:05 | 実習「cryoSPARCによる三次元再構成」川端 猛、加藤 貴之 | [講習会の様子の動画] | |
| 14:15 - 15:00 | 実習「Chimera 便利な機能」加藤 貴之 | [講義資料:Chimera便利な機能] | [講習会の様子の動画] |
| 15:30 - 18:00 | 実習「Cootによる原子モデリング」田中 秀明 |
[講義資料] [モデリング用データ(EM_kousuukai.zip)(53MB)] [講習ビデオ:Cootによる原子モデリング(youtube)] |
[講習会の様子の動画] |
<事前インストールが必要なソフトウエア>
- Windowsの場合
- WinCootのページ(https://bernhardcl.github.io/coot/wincoot-download.html) からダウンロードして、インストールできます。 [Download WinCoot 0.8.9]をクリックすると、0.9.2の版がインストールできます。
- https://www2.mrc-lmb.cam.ac.uk/personal/pemsley/cootのページ からCoot 0.8.9のWindows版をダウンロードできます。
- CCP4のページ(http://www.ccp4.ac.uk/download/#os=windows)から、 CCP4をインストールすると、Coot 0.8.9もいっしょにインストールされます。
- MacOS Xの場合 http://www.ccp4.ac.uk/download/#os=macのサイトからCCP4をインストールすると、 Coot 0.8.9も同時にインストールされます。
[wwPDB] wwPDBの定款を更新し,RDFを公式フォーマットの一つと定義しました

国際蛋白質データバンク(wwPDB)は、国際分業でPDBデータの登録、データ処理、配布を行う組織です。 2003年にRCSB PDB(米国)、PDBe(欧州)、PDBj(日本)の3団体が設立し、NMRデータのアーカイブキーパーとしてBMRB(米国)が2006年に加わりました。 これまでのwwPDBの定款(Charter)は2013年に合意したもので、幾つか現状にそぐわない点がでていました。そこでこの度、新しい定款(Charter)を作成し、 2021年1月に合意しました。この定款では古い物に比べて幾つもの点が具体に記載されていますが、特に以下の点が注目されます。
・電子顕微鏡構造解析(3DEM)によるマップやトモグラムのアーカイブキーパーとしてElectron Microscopy Data Bank (EMDB)をwwPDBの正メンバーに迎え入れました。
・wwPDBに准メンバーというカテゴリーを作り、新しい国や地域がwwPDBに参加するルールを明文化しました。
・PDBアーカイブのフォーマット規定を作り、PDBx/mmCIFフォーマットをPrimary format、PDBML(XML)とRDFフォーマットを正式なSecondary formatと定めました。
今回の正式合意は、EMDBとwwPDBの長年の関係を正式なものとするものです。 EMDBは2002年に欧州バイオインフォマティクス研究所(EBI)のPDBeによって設立されました。 それ以来、EMDBとwwPDBはデータの登録、アノテーション、検証に関する問題で協力してきました。 2012年以降、EMDBのマップデータは、wwPDBアーカイブ(ftp://ftp.wwpdb.org または ftp://ftp.pdbj.org/pub/ (PDBjミラーサイト))の一部として配布されています。
今回の正式合意で准メンバーというカテゴリーが新設されたので、今後、中国とインドを念頭にwwPDBの新しい地域拠点を設置することが可能となりました。 近年特に、中国からのデータ登録数は増加の一途とたどり、PDBjでのデータ処理数の半数程度を占めるようになっています。 wwPDBの枠組みの中で中国に新しいwwPDBの拠点を設置するべく、PDBjの主導的な役割が期待されています。
今回の正式合意では、wwPDBが認める公式データフォーマットについて記載しました。 PDBx/mmCIFフォーマットがPrimary formatであることを正式に記載し、PDBML(XML)とRDFの2つのフォーマットを公式なSecondary formatに指定しました。 データベースの統合利用に優れたRDFフォーマットの作成と利用を推進してきたPDBjとしては大変重要な変更点です。 代わりに、古くて限界の多いPDBフォーマットは正式なフォーマットとして記載されていません。
[ wwPDB News ]
[wwPDB] 検証レポートと座標データに対して一貫したPDBx/mmCIFフォーマットを提供します
OneDepの全ての新規登録エントリーに対して、wwPDB検証レポートをPDBjがPDBx/mmCIFフォーマットで先行して提供していましたが、wwPDB版の提供を開始します。 これによって検証データのPDBアーカイブとの相互運用性が国際的に高まることが期待されます。 検証レポートでは、PDBx/mmCIFフォーマットの方がデータは論理的に整理されているため、XMLフォーマットと比べて、 「データベースに適した」形となっています。 新しく公開又は更新されたエントリーのPDBx/mmCIFフォーマットの検証レポートは、 PDBアーカイブ(PDBj)と EMDBアーカイブ(PDBj)から配布されます
新しいPDBx/mmCIFレポートは、より解釈しやすくなっています。 大まかな要約情報を含み、また、残基レベルの情報へのアクセスが容易になっています。 データは、エンティティ・鎖別・そして個々の残基等、複数のレベルで提供されます。 例えば、クラッシュの総数を得ることも容易にできます。 検証レポートに対応するディクショナリは、wwPDBサイト mmcif.wwpdb.org/dictionaries/mmcif_pdbx_vrpt.dic/Index からご利用頂けます。 X線、電子顕微鏡、NMR構造に対応するPDBx/mmCIFフォーマットの検証レポート例は、GitHubから公開されています。
PDBおよびEMDBアーカイブの全エントリーに対してのPDBx/mmCIF検証レポートは、アーカイブ全体の検証レポートの再計算が行われた際に提供されます。
wwPDBは、PDBユーザーやソフトウェア開発者に対して、今後のアプリケーションには、PDBx/mmCIFフォーマットを採用することを強く推奨します。
[ wwPDB News ]
[wwPDB] 新しい4文字のCCD IDを含むエントリーや8文字のPDB IDのエントリーは、PDB形式では提供できなくなります
wwPDBは、PDBx/mmCIFワーキンググループと協力して、PDBおよびCCD(化合物辞書、Chemical Component Dictionary)のIDについて、コードの文字数を将来的に拡張します。これらの拡張IDを含むエントリーは、従来のPDBファイルフォーマットではサポートされません。
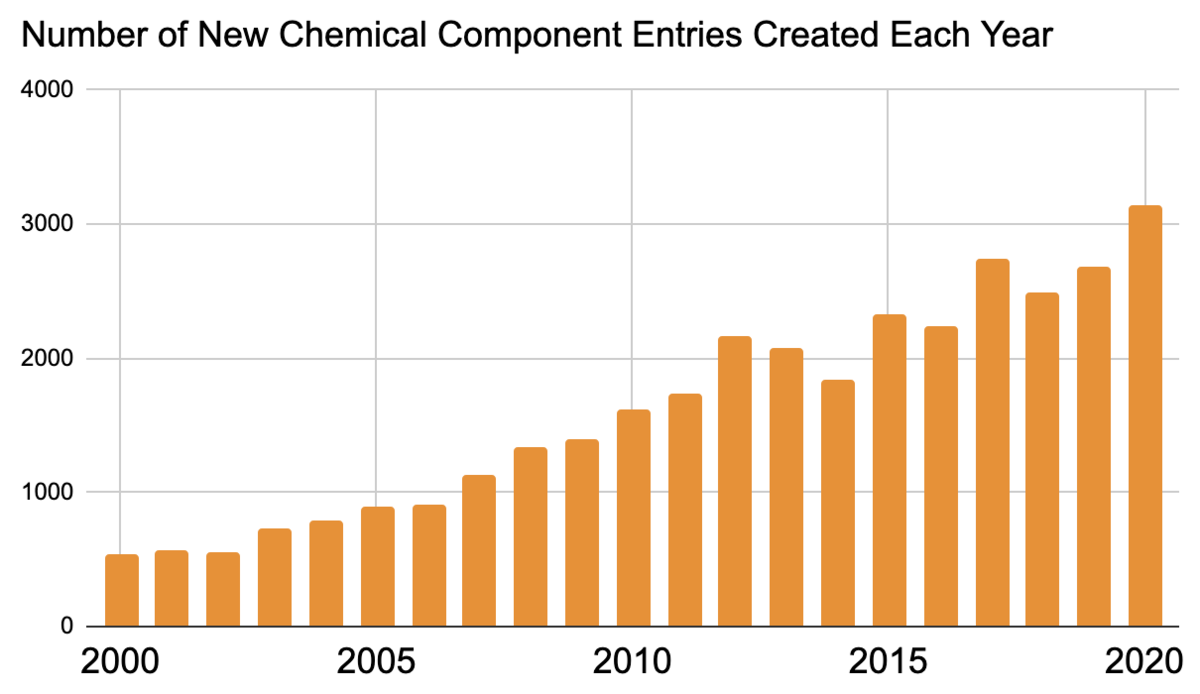
現在、CCDエントリーは3文字の英数字で識別されています。しかし現在の増加率で想定すると、3年から4年の間に3文字利用可能な新しいコードがなくなってしまうことが予想されます。この時点で、wwPDBはOneDepシステムにおいて、CCD IDとして4文字の英数字コードを発行するようになります。従来のPDBフォーマットは制約により新しい4文字のIDコードを表記できませんので、PDBx/mmCIFフォーマットでのみ提供されるようになります。 wwPDBでは、2022年に拡張CCD IDコードの実装を開始する予定です。
さらに、wwPDBでは、PDB_00001abcのように、PDB IDの長さを8文字に拡張し、PDBの前に「PDB」を付けることも計画しています。各PDB IDには対応するデジタルオブジェクト識別子(DOI)があり、これは多くの学術誌で原稿を投稿する際に必要とされ、論文に記載されます。拡張PDB IDとそれに対応するPDB DOIは、既存の4文字のPDB IDと合わせて、2021年秋までにすべての新規エントリーのPDBx/mmCIFフォーマットファイルに含まれる予定です。
例えば、PDBエントリー「1ABC」は、拡張PDB ID(pdb_00001abc)と対応するPDB DOI(10.2210/pdb1abc/pdb)もPDBx/mmCIFの _database_2 カテゴリに登録されます。
loop_ _database_2.database_id _database_2.database_code _database_2.pdbx_database_accession _database_2.pdbx_DOI PDB 1abc pdb_00001abc 10.2210/pdb1abc/pdb WWPDB D_1xxxxxxxxx ? ?
4文字のPDB IDがすべて消費されると、新規に登録されたPDBエントリーには拡張PDB IDコードのみが発行され、エントリーはPDBx/mmCIFフォーマットでのみ配布されます。
PDBユーザーと関連ソフトウェア開発者の皆様は、ご自身の開発されたソフトウェアのコードを見直し、将来確実に発生する制限を事前に取り除く作業を開始するようお願いします。
[ wwPDB News ]
[wwPDB] pdb-l 掲示板が新しくなりました
pdb-l@lists.wwpdb.org は、 蛋白質の構造、解析、および関連するトピックに関する質問やPDBユーザーコミュニティとの議論のためのオープンなディスカッションフォーラムです。 pdb-l@lists.wwpdb.org に送信されたメッセージは、すべての購読者に送信されます。 HTMLフォーマットと画像に対応したこの掲示板は、以前のフォーラム(pdb-l@sdsc.edu)に代わるものです。 前の掲示板から移行したメッセージも含めてアーカイブされています。
既存の購読者は、再登録の必要はありません。 新規ユーザーの方は、pdb-l@lists.wwpdb.org 情報ページにアクセスして購読してください。
移行に関する質問は、info@wwpdb.orgまでお願いします。
PDB構造に関する質問は、deposit-help@mail.wwpdb.orgまでお願いします。

[ wwPDB News ]
[wwPDB] 10,000件の区切りのPDBデータを処理した研究員
wwPDBの見学有美子さんが処理したPDBエントリーが10,000件に到達しました。おめでとうございます。 見学さんはPDBjの設立メンバーとして,母校である大阪大学にて2000年に業務を開始されました。 今年4月に彼女は10,000件以上の構造を処理した最初のwwPDB研究員となりました。 多くのアジア地区の構造生物学者は,彼女の名前をよくご存知と思います。 見学さんはPDBの編集作業を通じて,20年以上にわたり多くのPDB登録者とともに,高精度エントリーのタイムリーなリリースに貢献しています。 本年4月に彼女のPDBj, wPDBそして構造生物学コミュニティーへの貢献に対して,記念品を贈呈しました(写真)。 我々wwPDBでは,次の10,000件エントリー処理到達者がでることを心待ちにしています。

PDBj統括責任者・栗栖 源嗣教授と見学有美子さん

[ wwPDB News ]
[wwPDB] 拡張されたPDBx/mmCIF構造因子ファイルに対してOneDepのサポートを改善しました
PDBx/mmCIF辞書が,回折強度データの異方性を考慮した処理や,フレーム毎にマージしないデータ,異常分散データの品質記述を拡張した事を受けて,OneDepのサポートを改善しました。 2020年10月にwwPDBPDBx/mmCIF専門委員会のサブグループが招集され、実験データのより詳細な記述と関連するデータの品質評価基準の開発を開始しました。データ収集・処理に関するサブグループメンバーは,回折データ処理ソフトウェアの開発とサポートに積極的に取り組みました。 具体的には、数ヶ月にわたってオンライン会議を行い、新しい辞書コンテンツの拡張について議論、検討、最終決定を行い、2021年2月16日にPDBx/mmCIF辞書に組み込みました。 この新しいコンテンツ拡張の試験的な実装は、Global Phasing Ltd.により確認済みです。
これらの機能拡張により、より広範な回折データの保存とアーカイブが可能になり、データに関連する新しい品質評価基準も提供されます。拡張には主に次の3つの内容が含まれます。
- 回折異方性を考慮して処理されたスケール済み,マージ済み反射データに対し,(1)パラメータに依存せず反射ごとの「信号」とその信号のしきい値によりカットオフ分解能を定義し、(2)結果として得られた異方的な分解能を最もよく表現する分解能の異方性表す楕円記述子を提供します。
- 関連するモデルから独立してその品質が評価されることを目的とし追加項目を定義することによって,スケール済み,マージ済み反射データに対して,アーカイブから取り出した後にデータが有意義に再分析可能になります。
- 多くの関連する記述子を追加することにより,これまで欠けていた回折データの異常分散に関する統計的記述項目を追加しました。
回折分解能の異方性を記述する新しいmmCIFデータ拡張により、Global Phasing社が提供するSTARANISOプログラムによる解析結果をアーカイブすることが可能になりました。他のソフトウェアの開発者は、これらを利用したり、用途に合わせて現在の定義を拡張したりすることができます。新しい辞書拡張に準拠したautoPROCおよびBUSTER(version 20210224) で作成されたファイルの例は、GitHubリポジトリで提供されています。
これらのサンプルファイルや、他のデータ処理プログラムや精密化プログラムで作成された同様の準拠ファイルは、wwPDB OneDepシステムに直接アップロードするのに適しています。新しいpdbx_audit_conformレコードを使用した明示的な辞書のバージョニングの自動認識により、登録時の不要な前処理を回避することができます。今回のOneDepの改良により、研究室でのデータ処理/精密化とPDBへの登録との間でデータを失う事なく登録することが保証されます。
wwPDBは、構造生物学者に対して、PDB登録用のデータファイルを作成する際に、常に最新バージョンの構造決定ソフトウェアパッケージを使用することを強く推奨します。また、新しい構造を登録したいと考えている結晶学者に対しても、関連する回折データと一緒にこれらの新しいmmCIF辞書の拡張性を利用することを推奨しています。なぜなら、拡張を組み込んだソフトウェアは、データと最終モデルの間の一貫性を保証できるからです。 wwPDBはまた、オリジナルの回折画像データに直接アクセスできるように、登録者へ、回折像自体をいずれかの公開アーカイブから利用できるようにするよう推奨しています。
[ wwPDB News ]
[wwPDB] OneDep上のMol*ビューアでキュレーション後の生物学的単位を確認できます
キュレーション処理における生物学的単位の定義をより明確にするために、 wwPDBは、処理後のPDB生物学的単位を、登録者がOneDep上でMol*ビューアを使って確認できるように改善しました。
PDBエントリーのキュレーションにおける重要なプロセスの1つに、各構造の生物学的単位の定義があります。 これにより、PDBデータの利用者は、実験条件で複雑に形成された構造を理解するのに役立ちます。
生物学的単位が正しくキュレーションされていることを確認するために、 処理の過程でキュレータが確認した後、処理後に登録者へ報告します。
今回、OneDepの登録システムが強化され、キュレーション後の生物学的単位がMol* 3次元構造ビューアで表示され、 登録者が視覚的に確認できるようになりました。 このビューアは、登録インターフェイス上の新しい項目、Reviewセクションからご利用頂けます。Reviewセクションはキュレーション後に表示されます。 Mol*ビューアは、PDB構造データを最小限のメモリでブラウザ内に表示できるため、生物学的単位情報を視覚的に素早く簡単に表示することができます。
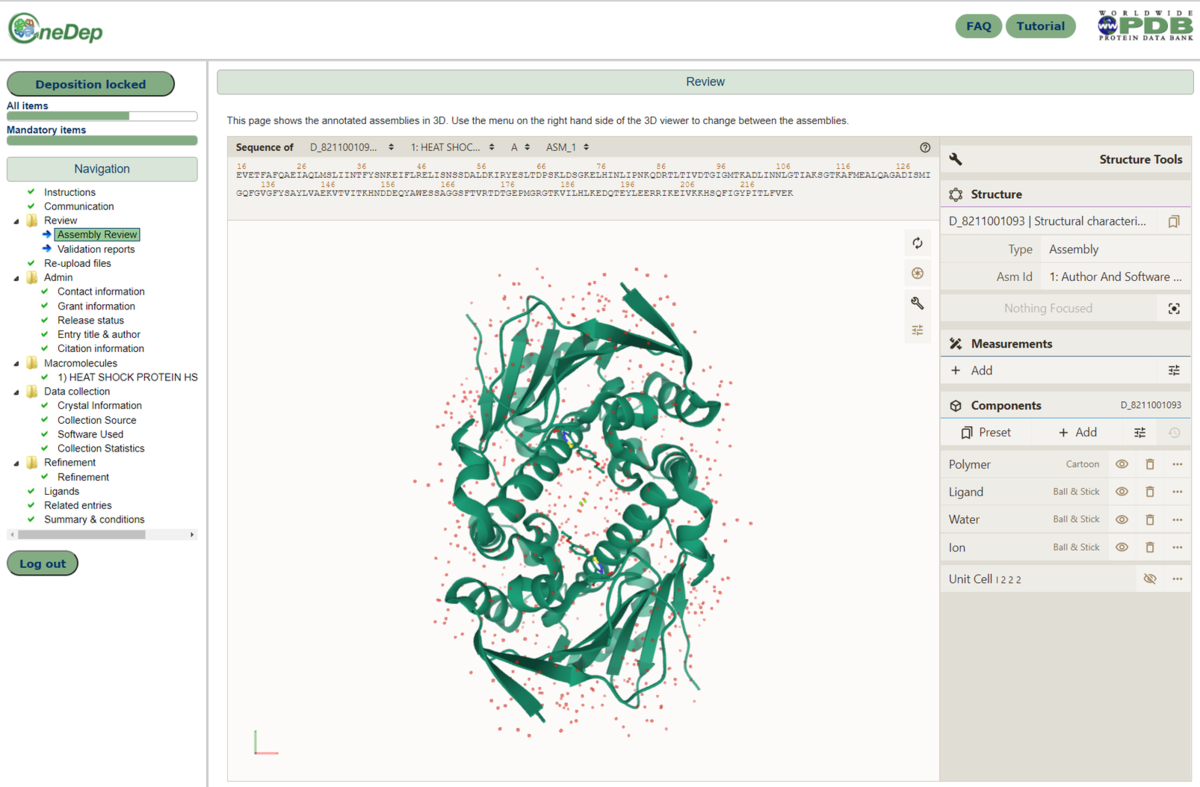 キュレーション後に登録者に表示される生物学的単位の確認ページ。キュレーションされた生物学的単位は、ブラウザ内のMol* 3Dビューアで表示されます。
キュレーション後に登録者に表示される生物学的単位の確認ページ。キュレーションされた生物学的単位は、ブラウザ内のMol* 3Dビューアで表示されます。
この変更により、PDBエントリーのための生物学的単位のキュレーションの検証と、登録過程における登録者への報告がより明確に分かり易くなります。
[ wwPDB News ]
[wwPDB] 2021年6月にSHEETとリガンドのSITEレコードへの対応を変更します
2014年にPDBx/mmCIFがPDBの標準フォーマットとなり、レガシーPDBフォーマットは凍結されました。 wwPDBでは、すべてのエントリーのPDBx/mmCIFファイルに加えて、このレガシーファイルフォーマットで表現できるエントリーについては、PDBフォーマットのファイルを作成しています (例えば、99,999個を超える原子を持つエントリーや、複数文字の鎖IDを持つエントリーはPDBx/mmCIFでしか利用できません)。
PDB構造のサイズと複雑さが増すにつれ、従来のPDBフォーマットの限界が明らかになってきており、対処する必要が出てきました。
複雑なシート(SHEET)レコードの定義
従来のPDBファイルフォーマットでは、SHEETレコードの列制限により、複雑なβシートトポロジーを生成することができませんでした。 複雑なβシートトポロジーとは、βストランドが複数のβシートに分かれている場合や、βシート内のストランドの定義が直線的な記述では表現できない場合などを指します。 例えば、PDBエントリー5wlnでは、1つのタンパク質の複数のコピーから大きなベータバレル構造が作成されています。 バレルを形成するベータシート内では、1本のベータ鎖が片側で複数の他の鎖と接触している例があり、それは異なる鎖のものであっても同様である。
しかし、この制限は、PDBx/mmCIFフォーマットのファイルでは問題にならず、これらの複雑なβシートトポロジーは、 _struct_sheet, _struct_sheet_order, _struct_sheet_range, and _struct_sheet_hbondで捉えることができます。
2021年6月8日より、SHEETトポロジーが生成できないPDBエントリーについては、レガシーPDBフォーマットのファイルは生成されなくなります。 これらの構造については、wwPDBは引き続きPDBx/mmCIFフォーマットのファイルで、ヘリックスとシートの情報を含む二次構造情報を提供します。
_struct_siteカテゴリ (SITEレコード)の廃止
wwPDBでは、OneDepのバイオキュレーションに使用したソフトウェアを定期的に検討・見直しています。 PDBx/mmCIFの_struct_siteと_struct_site_genカテゴリ(従来のPDBファイルフォーマットのSITEレコード)は、 組織内の昔からのソフトウェアによって生成され、純粋に距離計算に基づいているため、生物学的機能部位を反映していない可能性があります。
2021年6月より、_struct_siteおよび_struct_site_genレコードを生成するレガシーソフトウェアが廃止され、 wwPDBは新規に登録されたPDBエントリーに対してこれらのカテゴリを生成しなくなります。既存のエントリーは影響を受けません。
[ wwPDB News ]
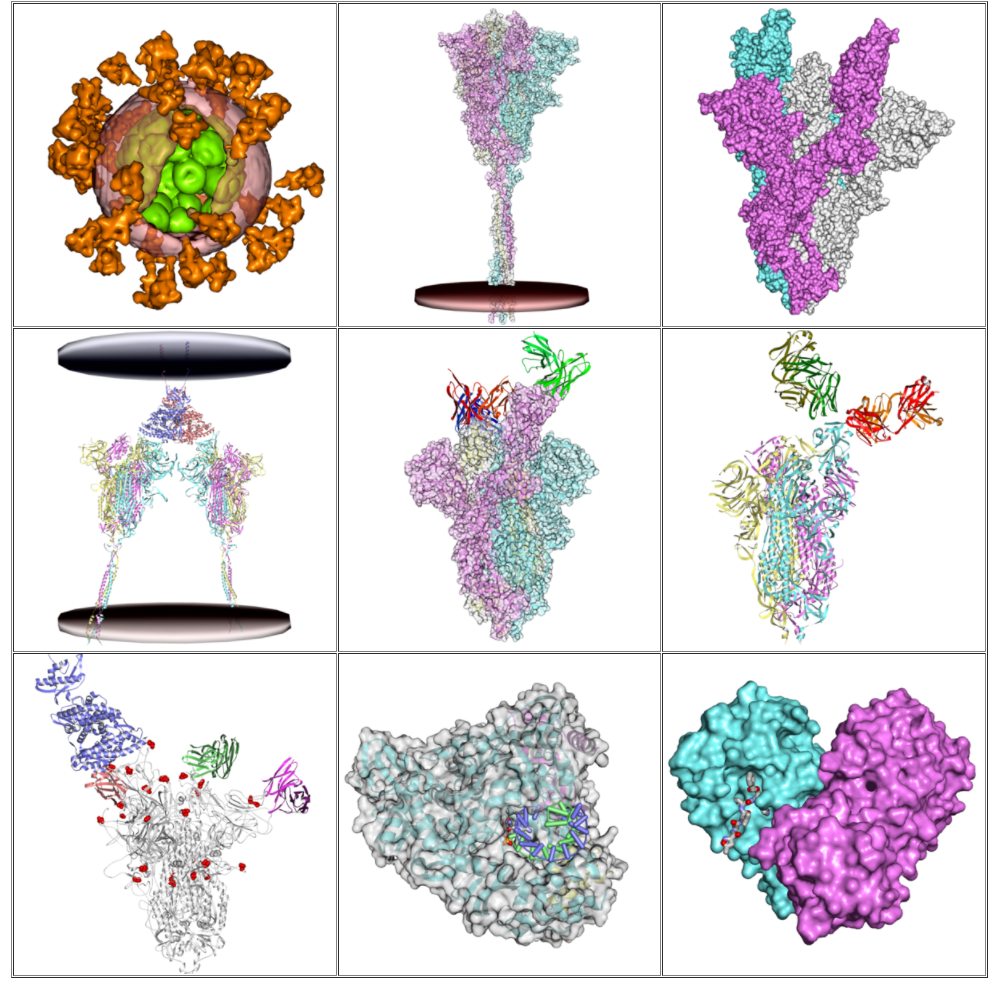
[wwPDB] 低分子リガンドと糖鎖の検証の強化について
Structure誌に掲載された新しい記事では、 糖鎖の分岐表現や2次元SNFG画像、関心のあるリガンドの識別、電子密度重ね合わせの3次元ビュー、低分子配置の2次元画像などの新機能が紹介されています。
3次元低分子構造の検証のためのこれらの機能強化とプロセスは、wwPDB/CCDC/D3R Ligand Validation Workshopからの提言と、 コミュニティの共同作業によるソフトウェアの採用を反映しています。
この原稿では、Validation of the Structures in the Protein Data Bank (2017) Structure 25: 1916-1927 doi: 10.1016/j.str.2017.10.009 に記載されているように、Validation Reportsの最初の実装以降に行われた強化点も紹介しています。
Enhanced Validation of Small-Molecule Ligands and Carbohydrates in the Protein Data Bank
Zukang Feng, John D. Westbrook, Raul Sala, Oliver S. Smart, Gérard Bricogne, Masaaki Matsubara,
Issaku Yamada, Shinichiro Tsuchiya, Kiyoko F. Aoki-Kinoshita, Jeffrey C. Hoch, Genji Kurisu, Sameer Velankar,
Stephen K. Burley, and Jasmine Y. Young
(2021) Structure doi: 10.1016/j.str.2021.02.004
[ wwPDB News ]
[wwPDB] 3月15日までにPDB50のアブストラクトを投稿してください
wwPDBは2021年を通してPDBアーカイブの50周年の記念行事を行います (wwpdb.org/pdb50).
まず最初に2021年5月4日、5日(米国東部時間, EST)には、発足シンポジウムがオンラインで開催される予定です。
オンラインのセッションは、米国東部時間 5月4日、5日の午前11時~午後4時30分(日本時間 5月5日、6日の午前0時~5時30分)に開催されます。 イベントの様子を録画し、会議終了後に、登録済みの参加者へ公開する予定です。
学生や若手研究者はポスター賞の対象となりますので,積極的な参加を期待しております。
アブストラクトの投稿と参加費の割引は3月15日までです。https://www.asbmb.org/meetings-events/pdb50からご登録ください。
講演者
[ wwPDB News ]
[wwPDB] 1000件以上の新型コロナウイルス(SARS-CoV-2)関連構造が公開されています
今週の更新で、新型コロナウイルス(SARS-CoV-2)関連構造が1000件を超え、1018件の関連エントリーをPDBからご利用頂けるようになりました。
最初のSARS-CoV-2由来蛋白質の構造はメインプロテアーゼ(PDB 6lu7, wwPDB DOI landing page)の高分解能結晶構造で、流行初期の2020年2月5日に公開されました。
それ以来構造生物学者は、SARS-CoV-2のスパイク蛋白質、そのヒトACE2受容体および中和抗体との複合体など、SARS-CoV-2蛋白質の大部分を可視化し、メインプロテアーゼ、パパイン様プロテアーゼ、その他の有望な創薬ターゲットの構造を明らかにしてきました。
全ての構造と関連データは、国際蛋白質構造データバンク(wwPDB)のパートナーサイト( RCSB PDB, PDBe, PDBj, and BMRB )からご利用頂けます。
また、PDBjサイトの NMRデータ, EMマップ, in Silicoモデル もご利用頂けます。
SARS-CoV-2の構造データの迅速な公開により、Covid-19についての理解が大きく深まり、変異ウイルスの新たな変異導入箇所を直接可視化できるようにもなりました。また、感染症を阻止するための構造情報に基づいた創薬と、既存薬の転用利用が容易になりました。PDB構造データへ自由にアクセスできるおかげで、すでにSARS-CoV-2に対するワクチンの効率的な分子設計も可能となっています。
今回のパンデミックに対する研究者の真摯な対応は、科学データへ即時に自由にアクセスできることの重要性を浮き彫りにしました。wwPDBは、可能な限り包括的で正確なアーカイブを維持しながら、誰もが3次元構造データへ自由にアクセスできるように努力を続けています。
今回の1018件の構造と今後更に解析されるであろう新型コロナウイルスの蛋白質構造がもたらすインパクトは、構造生物学研究データへのオープンアクセス(誰もが無料で自由にアクセスできること)の重要性を証明しています。
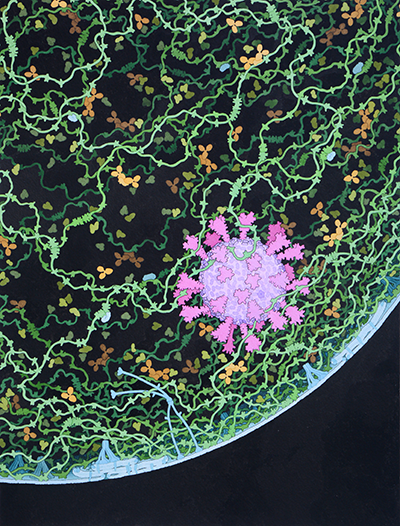
Illustration by David S. Goodsell, RCSB Protein Data Bank; doi: 10.2210/rcsb_pdb/goodsell-gallery-024
[ wwPDB News ]
[wwPDB] PDB50: オンラインイベントの登録受付を開始しました
2021年を通して、wwPDBはPDBアーカイブの50周年を記念しています。
2021年5月4日、5日(米国東部時間, EST)には、 米国生化学分子生物学会(American Society for Biochemistry and Molecular Biology)が主催し、 wwPDB財団が企画するイベントにおいて、発足シンポジウムが開催される予定です。
生物学界初のオープンアクセス・デジタルデータリソースとしてのPDBの設立50周年を記念して、構造生物学とバイオインフォマティクスの分野で多大な進歩を遂げた世界中の講演者による講演が予定されています。
参加者はオンラインのポスターセッションや展示会に参加することをお勧めします。学生や研究者はポスター賞の対象となります。
2021年3月15日までに登録しアブストラクトを投稿すれば、参加費の割引が適用されます。
予定されている講演者は以下の通りです
- Edward Arnold - Rutgers, The State University of New Jersey
- Helen M. Berman - Rutgers, The State University of New Jersey and University of Southern California
- Thomas L. Blundell - University of Cambridge
- Alexandre M. J. J. Bonvin - Utrecht University
- Stephen K. Burley - Rutgers, The State University of New Jersey and University of California San Diego
- Wah Chiu - Stanford University
- Johann Deisenhofer - University of Texas Southwestern Medical Center
- Juli Feigon - University of California Los Angeles
- Angela M. Gronenborn - University of Pittsburgh
- Jennifer L. Martin - University of Wollongong
- Stephen L. Mayo - California Institute of Technology
- Zihe Rao - ShanghaiTech University and Tsinghua University
- Hao Wu - Boston Children's Hospital and Harvard Medical School
オンラインのセッションは、米国東部時間 5月4日、5日の午前11時~午後4時30分(日本時間 5月5日、6日の午前0時~5時30分)に開催されます。 イベントの様子を録画し、会議終了後に、登録済みの参加者へ公開する予定です。
スポンサーシップの機会もご利用ください。詳細については、wwPDB財団にご連絡ください。
[ wwPDB News ]
[wwPDB] EMエントリーのwwPDB検証レポートを公開しました
wwPDBアーカイブ(PDB, EMDB)を更新し、PDBアーカイブ内の全てのEMモデル座標エントリーと全てのEMDBマップエントリーについて、検証レポートを追加・更新しました。検証レポートは、構造の品質について定量的および視覚的な評価を提供し、アーカイブ全体で比較できるようになります (https://www.wwpdb.org/validation/validation-reports).
wwPDBのEM検証レポートは、2019年に初めて、OneDepの登録者に向けて提供されました。 (wwPDBニュース). 現在のレポートは、2010年 (Structure 20: 205-214, wwpdb.org/task/em) と2020年(準備中の報告書)に行われたEM検証専門委員会(VTF, Validation Task Force)の推奨事項とEM検証チャレンジイベント (https://www.ncbi.nlm.nih.gov/pubmed/32002441, https://www.biorxiv.org/content/10.1101/2020.06.12.147033v1)に基づいたものです。 最近の改善例には、登録されたマスクの画像、改良されたマップとモデルの重ね合わせ画像、2つのハーフマップからの(おおよその)生マップの視覚化、および回転平均したパワースペクトルのグラフ図が含まれます。基盤となる方法論は、EMコミュニティの要件や要求、フィードバックに基づいて継続的に改善を行っています。
PDBコアアーカイブには、各PDBエントリーと、それに関連するEMDBの実験マップ/トモグラムを評価する検証レポートが配置されます。EMマップ+モデル座標の検証レポートは、以下のwwPDBミラーサイトからダウンロード頂けます。
- wwPDB: ftp://ftp.wwpdb.org/pub/pdb/validation_reports/
- RCSB PDB: ftp://ftp.rcsb.org/pub/pdb/validation_reports/
- PDBe: ftp://ftp.ebi.ac.uk/pub/databases/pdb/validation_reports/
- PDBj: ftp://ftp.pdbj.org/pub/pdb/validation_reports/
EMDBコアアーカイブには、EMDBのマップ/トモグラムエントリーを評価する検証レポートが配置されます。EMマップに関する検証レポートは、以下のURLからダウンロード頂けます。
- EMDB: ftp://ftp.ebi.ac.uk/pub/databases/emdb/validation_reports/
- wwPDB: ftp://ftp.wwpdb.org/pub/emdb/validation_reports/
- RCSB PDB: ftp://ftp.rcsb.org/pub/emdb/validation_reports/
- PDBj: ftp://ftp.pdbj.org/pub/emdb/validation_reports/
EM マップ+モデル、EM マップのみ、 EM トモグラムに対する検証レポートについての更に詳細な情報は、各リンク先のwwPDBの検証レポートユーザガイドをご覧ください。
wwPDBの検証レポートについて、ご質問やコメントがございましたら、validation@mail.wwpdb.orgまで、英語でご連絡ください。
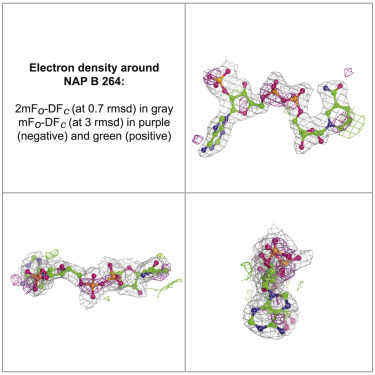
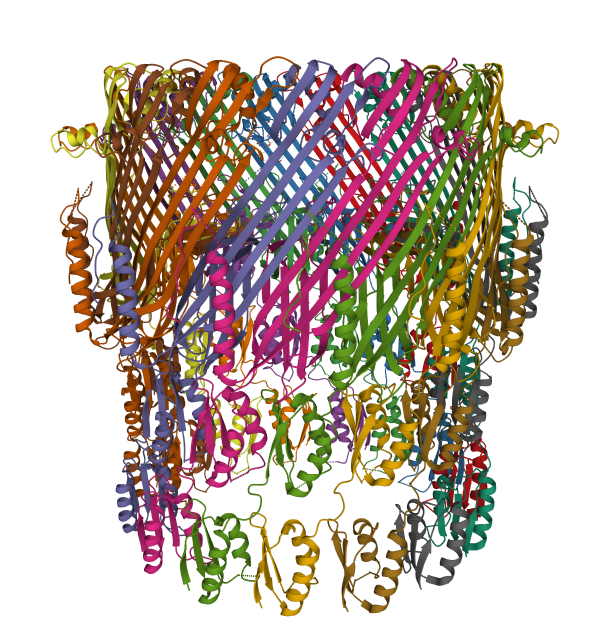 マップとモデルの重ねあわせ画像の例:EMD-30388/7CWU 「SARS-CoV-2 spike proteins trimer in complex with P17 and FC05 Fabs cocktail」
マップとモデルの重ねあわせ画像の例:EMD-30388/7CWU 「SARS-CoV-2 spike proteins trimer in complex with P17 and FC05 Fabs cocktail」
[ wwPDB News ]