+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Medicago truncatula HISN5 (IGPD) in complex with MN and IG2 | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Medicago truncatula HISN5 IGPD histidine /  LYASE (リアーゼ) LYASE (リアーゼ) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 imidazoleglycerol-phosphate dehydratase / imidazoleglycerol-phosphate dehydratase /  imidazoleglycerol-phosphate dehydratase activity / L-histidine biosynthetic process imidazoleglycerol-phosphate dehydratase activity / L-histidine biosynthetic process類似検索 - 分子機能 | |||||||||
| 生物種 |   Medicago truncatula (タルウマゴヤシ) Medicago truncatula (タルウマゴヤシ) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.23 Å クライオ電子顕微鏡法 / 解像度: 2.23 Å | |||||||||
 データ登録者 データ登録者 | Witek W / Ruszkowski M | |||||||||
| 資金援助 |  ポーランド, 1件 ポーランド, 1件
| |||||||||
 引用 引用 |  ジャーナル: Front Plant Sci / 年: 2024 ジャーナル: Front Plant Sci / 年: 2024タイトル: Targeting imidazole-glycerol phosphate dehydratase in plants: novel approach for structural and functional studies, and inhibitor blueprinting. 著者: Wojciech Witek / Joanna Sliwiak / Michal Rawski / Milosz Ruszkowski /  要旨: The histidine biosynthetic pathway (HBP) is targeted for herbicide design with preliminary success only regarding imidazole-glycerol phosphate dehydratase (IGPD, EC 4.2.1.19), or HISN5, as referred ...The histidine biosynthetic pathway (HBP) is targeted for herbicide design with preliminary success only regarding imidazole-glycerol phosphate dehydratase (IGPD, EC 4.2.1.19), or HISN5, as referred to in plants. HISN5 catalyzes the sixth step of the HBP, in which imidazole-glycerol phosphate (IGP) is dehydrated to imidazole-acetol phosphate. In this work, we present high-resolution cryoEM and crystal structures of HISN5 (HISN5) in complexes with an inactive IGP diastereoisomer and with various other ligands. HISN5 can serve as a new model for plant HISN5 structural studies, as it enables resolving protein-ligand interactions at high (2.2 Å) resolution using cryoEM. We identified ligand-binding hotspots and characterized the features of plant HISN5 enzymes in the context of the HISN5-targeted inhibitor design. Virtual screening performed against millions of small molecules not only revealed candidate molecules but also identified linkers for fragments that were experimentally confirmed to bind. Based on experimental and computational approaches, this study provides guidelines for designing symmetric HISN5 inhibitors that can reach two neighboring active sites. Finally, we conducted analyses of sequence similarity networks revealing that plant HISN5 enzymes derive from cyanobacteria. We also adopted a new approach to measure HISN5 enzymatic activity using isothermal titration calorimetry and enzymatically synthesized IGP. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18305.map.gz emd_18305.map.gz | 96.9 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18305-v30.xml emd-18305-v30.xml emd-18305.xml emd-18305.xml | 16.9 KB 16.9 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_18305.png emd_18305.png | 57.3 KB | ||
| Filedesc metadata |  emd-18305.cif.gz emd-18305.cif.gz | 5.9 KB | ||
| その他 |  emd_18305_half_map_1.map.gz emd_18305_half_map_1.map.gz emd_18305_half_map_2.map.gz emd_18305_half_map_2.map.gz | 95.2 MB 95.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18305 http://ftp.pdbj.org/pub/emdb/structures/EMD-18305 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18305 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18305 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18305.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18305.map.gz / 形式: CCP4 / 大きさ: 103 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.86 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_18305_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_18305_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Complex of imidazole glycerol phosphate dehydratase (HISN5) with ...
| 全体 | 名称: Complex of imidazole glycerol phosphate dehydratase (HISN5) with inhibitor 2S,3S-IGP (IG2) |
|---|---|
| 要素 |
|
-超分子 #1: Complex of imidazole glycerol phosphate dehydratase (HISN5) with ...
| 超分子 | 名称: Complex of imidazole glycerol phosphate dehydratase (HISN5) with inhibitor 2S,3S-IGP (IG2) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Medicago truncatula (タルウマゴヤシ) Medicago truncatula (タルウマゴヤシ) |
| 分子量 | 理論値: 22.5 kDa/nm |
-分子 #1: Imidazoleglycerol-phosphate dehydratase
| 分子 | 名称: Imidazoleglycerol-phosphate dehydratase / タイプ: protein_or_peptide / ID: 1 / コピー数: 24 / 光学異性体: LEVO / EC番号:  imidazoleglycerol-phosphate dehydratase imidazoleglycerol-phosphate dehydratase |
|---|---|
| 由来(天然) | 生物種:   Medicago truncatula (タルウマゴヤシ) Medicago truncatula (タルウマゴヤシ) |
| 分子量 | 理論値: 22.571293 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: PTFPIDSGAR IGEMKRVTKE TNVSVKINLD GTGVADNSSG IPFLDHMLDQ LASHGLFDVH VKATGDTHID DHHTNEDVAL AIGTALLQA LGDRKGINRF GNFSAPLDEA LVHVSLDLSG RPHLGYDLNI PTQRVGKYDT QLVEHFFQSL VNTSGMTLHI R QFSGTNSH ...文字列: PTFPIDSGAR IGEMKRVTKE TNVSVKINLD GTGVADNSSG IPFLDHMLDQ LASHGLFDVH VKATGDTHID DHHTNEDVAL AIGTALLQA LGDRKGINRF GNFSAPLDEA LVHVSLDLSG RPHLGYDLNI PTQRVGKYDT QLVEHFFQSL VNTSGMTLHI R QFSGTNSH HIIEATFKAF ARALRQATEY DTRRRGTIPS SKGVLSRS UniProtKB:  Imidazoleglycerol-phosphate dehydratase Imidazoleglycerol-phosphate dehydratase |
-分子 #2: MANGANESE (II) ION
| 分子 | 名称: MANGANESE (II) ION / タイプ: ligand / ID: 2 / コピー数: 48 / 式: MN |
|---|---|
| 分子量 | 理論値: 54.938 Da |
-分子 #3: (2S,3S)-2,3-dihydroxy-3-(1H-imidazol-5-yl)propyl dihydrogen phosphate
| 分子 | 名称: (2S,3S)-2,3-dihydroxy-3-(1H-imidazol-5-yl)propyl dihydrogen phosphate タイプ: ligand / ID: 3 / コピー数: 24 / 式: IG2 |
|---|---|
| 分子量 | 理論値: 238.135 Da |
| Chemical component information | 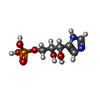 ChemComp-IG2: |
-分子 #4: water
| 分子 | 名称: water / タイプ: ligand / ID: 4 / コピー数: 1131 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 8 |
| グリッド | モデル: Quantifoil / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: SPOT SCAN / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.0 µm Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 3.5 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 1 / 実像数: 3330 / 平均電子線量: 40.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 初期モデル | モデルのタイプ: NONE |
|---|---|
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 3次元分類 | クラス数: 50 / ソフトウェア - 名称: cryoSPARC |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC |
| 最終 再構成 | 使用したクラス数: 14 / 想定した対称性 - 点群: O (正8面体型対称 ) / 解像度のタイプ: BY AUTHOR / 解像度: 2.23 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC (ver. 3.3.2) / 使用した粒子像数: 605614 ) / 解像度のタイプ: BY AUTHOR / 解像度: 2.23 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC (ver. 3.3.2) / 使用した粒子像数: 605614 |
 ムービー
ムービー コントローラー
コントローラー








 Z
Z Y
Y X
X