+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-1511 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | COPII coat | |||||||||
 マップデータ マップデータ | This is a reconstruction of a COPII coat comprised of Sec13-31 and Sec23-24. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  vesicle trafficking (小胞) / vesicle trafficking (小胞) /  COPII (COPII) / icosidodecahedron / COPII (COPII) / icosidodecahedron /  secretory pathway (分泌) / secretory pathway (分泌) /  endoplasmic reticulum (小胞体) / endoplasmic reticulum (小胞体) /  automation (自動) automation (自動) | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 43.0 Å クライオ電子顕微鏡法 / 解像度: 43.0 Å | |||||||||
 データ登録者 データ登録者 | Stagg SM / LaPointe P / Razvi A / Gurkan C / Potter CS / Carragher B / Balch WE | |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2008 ジャーナル: Cell / 年: 2008タイトル: Structural basis for cargo regulation of COPII coat assembly. 著者: Scott M Stagg / Paul LaPointe / Abbas Razvi / Cemal Gürkan / Clinton S Potter / Bridget Carragher / William E Balch /  要旨: Using cryo-electron microscopy, we have solved the structure of an icosidodecahedral COPII coat involved in cargo export from the endoplasmic reticulum (ER) coassembled from purified cargo adaptor ...Using cryo-electron microscopy, we have solved the structure of an icosidodecahedral COPII coat involved in cargo export from the endoplasmic reticulum (ER) coassembled from purified cargo adaptor Sec23-24 and Sec13-31 lattice-forming complexes. The coat structure shows a tetrameric assembly of the Sec23-24 adaptor layer that is well positioned beneath the vertices and edges of the Sec13-31 lattice. Fitting the known crystal structures of the COPII proteins into the density map reveals a flexible hinge region stemming from interactions between WD40 beta-propeller domains present in Sec13 and Sec31 at the vertices. The structure shows that the hinge region can direct geometric cage expansion to accommodate a wide range of bulky cargo, including procollagen and chylomicrons, that is sensitive to adaptor function in inherited disease. The COPII coat structure leads us to propose a mechanism by which cargo drives cage assembly and membrane curvature for budding from the ER. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_1511.map.gz emd_1511.map.gz | 13.7 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-1511-v30.xml emd-1511-v30.xml emd-1511.xml emd-1511.xml | 11.3 KB 11.3 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  1511.gif 1511.gif | 48.2 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-1511 http://ftp.pdbj.org/pub/emdb/structures/EMD-1511 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1511 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-1511 | HTTPS FTP |
-関連構造データ
| 類似構造データ |
|---|
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_1511.map.gz / 形式: CCP4 / 大きさ: 26.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_1511.map.gz / 形式: CCP4 / 大きさ: 26.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | This is a reconstruction of a COPII coat comprised of Sec13-31 and Sec23-24. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 7.7 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
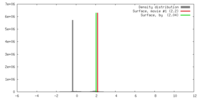
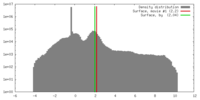
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : Sec13/31 bound to Sec23/24
| 全体 | 名称: Sec13/31 bound to Sec23/24 |
|---|---|
| 要素 |
|
-超分子 #1000: Sec13/31 bound to Sec23/24
| 超分子 | 名称: Sec13/31 bound to Sec23/24 / タイプ: sample / ID: 1000 集合状態: 60 Sec13-31 heterotetramers bound to 120 Sec23-24 heterodimers Number unique components: 4 |
|---|---|
| 分子量 | 理論値: 44.8 MDa |
-超分子 #1: COPII coat
| 超分子 | 名称: COPII coat / タイプ: organelle_or_cellular_component / ID: 1 / Name.synonym: Sec13-31 and Sec23-24 / 組換発現: Yes |
|---|---|
| Ref GO | divclassse qspanoncli ckpopupspa nclassgree n(this)spandata popltspanc lassquotlo adingbarqu otgtltimgs rcquotimgl oadinggifq uotdecodin gquotasync quotgtltsp angtdataur lajaxphp?m odetaxoamp ... divclassse qspanoncli ckpopupspa nclassgree n(this)spandata popltspanc lassquotlo adingbarqu otgtltimgs rcquotimgl oadinggifq uotdecodin gquotasync quotgtltsp angtdataur lajaxphp?m odetaxoamp kGO3A00068 88ampajax1 classpoptr giGO000688 8ispandiv |
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / 別称: Human / 細胞中の位置: cytosol Homo sapiens (ヒト) / 別称: Human / 細胞中の位置: cytosol |
| 分子量 | 実験値: 44.8 MDa / 理論値: 44.8 MDa |
| 組換発現 | 生物種: Insect cells / 組換プラスミド: bacmid |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 6.8 / 詳細: 50 mM MES pH 6.8, 700 mM KOAc, 1 mM MgOAc, 1 mM DTT |
|---|---|
| グリッド | 詳細: 400 mesh grid |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 98 K / 装置: OTHER / 詳細: Vitrification instrument: vitrobot / 手法: blot for 3 seconds before plunging |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI F20 |
|---|---|
| 電子線 | 加速電圧: 200 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 29000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2 mm / 最小 デフォーカス(公称値): 10.0 µm / 倍率(公称値): 29000 Bright-field microscopy / Cs: 2 mm / 最小 デフォーカス(公称値): 10.0 µm / 倍率(公称値): 29000 |
| 試料ステージ | 試料ホルダー: Side entry liquid nitrogen-cooled cryo specimen holder 試料ホルダーモデル: GATAN LIQUID NITROGEN |
| 温度 | 平均: 88 K |
| アライメント法 | Legacy - 非点収差: astigmatism corrected automatically with Leginon. |
| 撮影 | カテゴリ: CCD フィルム・検出器のモデル: GATAN ULTRASCAN 4000 (4k x 4k) 平均電子線量: 20 e/Å2 |
| 実験機器 |  モデル: Tecnai F20 / 画像提供: FEI Company |
- 画像解析
画像解析
| CTF補正 | 詳細: each particle |
|---|---|
| 最終 2次元分類 | クラス数: 212 |
| 最終 再構成 | 想定した対称性 - 点群: I (正20面体型対称 ) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 43.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: EMAN / 詳細: Final map was lowpass filtered to 43 angstroms / 使用した粒子像数: 12120 ) / アルゴリズム: OTHER / 解像度のタイプ: BY AUTHOR / 解像度: 43.0 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: EMAN / 詳細: Final map was lowpass filtered to 43 angstroms / 使用した粒子像数: 12120 |
| 詳細 | Data was automatically collected using Leginon. Data was processed automatically with Appion. Particles were picked manually. |
-原子モデル構築 1
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称: Normal mode-based flexible fitting (nmff) and chimera |
| 詳細 | Protocol: normal modes flexible fitting and rigid body. A bend was modeled into Sec13-31 using the program nmff. Sec13-31 and Sec23-24 were separately fitted into the cryoEM density using the program Chimera. |
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
-原子モデル構築 2
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称: nmff and chimera |
| 詳細 | Protocol: normal modes flexible fitting and rigid body. A bend was modeled into Sec13-31 using the program nmff. Sec13-31 and Sec23-24 were separately fitted into the cryoEM density using the program Chimera. |
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
-原子モデル構築 3
| 初期モデル | PDB ID: |
|---|---|
| ソフトウェア | 名称: nmff and chimera |
| 詳細 | Protocol: normal modes flexible fitting and rigid body. A bend was modeled into Sec13-31 using the program nmff. Sec13-31 and Sec23-24 were separately fitted into the cryoEM density using the program Chimera. |
| 精密化 | 空間: REAL / プロトコル: FLEXIBLE FIT |
 ムービー
ムービー コントローラー
コントローラー








 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)