+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8u4b | ||||||
|---|---|---|---|---|---|---|---|
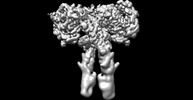
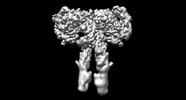
| タイトル | Cryo-EM structure of long form insulin receptor (IR-B) in the apo state | ||||||
 要素 要素 | Insulin receptor インスリン受容体 インスリン受容体 | ||||||
 キーワード キーワード |  SIGNALING PROTEIN / SIGNALING PROTEIN /  Insulin receptor (インスリン受容体) / IGF2 / RTK Insulin receptor (インスリン受容体) / IGF2 / RTK | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of female gonad development / positive regulation of meiotic cell cycle / positive regulation of developmental growth /  insulin-like growth factor II binding / male sex determination / exocrine pancreas development / insulin-like growth factor II binding / male sex determination / exocrine pancreas development /  insulin receptor complex / insulin receptor complex /  insulin-like growth factor I binding / insulin-like growth factor I binding /  insulin receptor activity / positive regulation of protein-containing complex disassembly ...regulation of female gonad development / positive regulation of meiotic cell cycle / positive regulation of developmental growth / insulin receptor activity / positive regulation of protein-containing complex disassembly ...regulation of female gonad development / positive regulation of meiotic cell cycle / positive regulation of developmental growth /  insulin-like growth factor II binding / male sex determination / exocrine pancreas development / insulin-like growth factor II binding / male sex determination / exocrine pancreas development /  insulin receptor complex / insulin receptor complex /  insulin-like growth factor I binding / insulin-like growth factor I binding /  insulin receptor activity / positive regulation of protein-containing complex disassembly / cargo receptor activity / insulin receptor activity / positive regulation of protein-containing complex disassembly / cargo receptor activity /  dendritic spine maintenance / dendritic spine maintenance /  insulin binding / insulin binding /  PTB domain binding / adrenal gland development / neuronal cell body membrane / Signaling by Insulin receptor / IRS activation / amyloid-beta clearance / activation of protein kinase activity / positive regulation of respiratory burst / PTB domain binding / adrenal gland development / neuronal cell body membrane / Signaling by Insulin receptor / IRS activation / amyloid-beta clearance / activation of protein kinase activity / positive regulation of respiratory burst /  regulation of embryonic development / positive regulation of receptor internalization / transport across blood-brain barrier / regulation of embryonic development / positive regulation of receptor internalization / transport across blood-brain barrier /  insulin receptor substrate binding / epidermis development / positive regulation of glycogen biosynthetic process / Signal attenuation / insulin receptor substrate binding / epidermis development / positive regulation of glycogen biosynthetic process / Signal attenuation /  phosphatidylinositol 3-kinase binding / heart morphogenesis / dendrite membrane / Insulin receptor recycling / phosphatidylinositol 3-kinase binding / heart morphogenesis / dendrite membrane / Insulin receptor recycling /  insulin-like growth factor receptor binding / neuron projection maintenance / activation of protein kinase B activity / positive regulation of glycolytic process / Insulin receptor signalling cascade / insulin-like growth factor receptor binding / neuron projection maintenance / activation of protein kinase B activity / positive regulation of glycolytic process / Insulin receptor signalling cascade /  receptor-mediated endocytosis / positive regulation of mitotic nuclear division / receptor-mediated endocytosis / positive regulation of mitotic nuclear division /  学習 / 学習 /  カベオラ / positive regulation of glucose import / positive regulation of MAP kinase activity / カベオラ / positive regulation of glucose import / positive regulation of MAP kinase activity /  receptor internalization / receptor internalization /  受容体型チロシンキナーゼ / 受容体型チロシンキナーゼ /  記憶 / cellular response to growth factor stimulus / 記憶 / cellular response to growth factor stimulus /  cell surface receptor protein tyrosine kinase signaling pathway / cellular response to insulin stimulus / peptidyl-tyrosine phosphorylation / male gonad development / positive regulation of nitric oxide biosynthetic process / late endosome / cell surface receptor protein tyrosine kinase signaling pathway / cellular response to insulin stimulus / peptidyl-tyrosine phosphorylation / male gonad development / positive regulation of nitric oxide biosynthetic process / late endosome /  glucose homeostasis / insulin receptor signaling pathway / glucose homeostasis / insulin receptor signaling pathway /  amyloid-beta binding / PI5P, PP2A and IER3 Regulate PI3K/AKT Signaling / amyloid-beta binding / PI5P, PP2A and IER3 Regulate PI3K/AKT Signaling /  protein tyrosine kinase activity / positive regulation of MAPK cascade / protein autophosphorylation / protein tyrosine kinase activity / positive regulation of MAPK cascade / protein autophosphorylation /  リソソーム / positive regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction / リソソーム / positive regulation of phosphatidylinositol 3-kinase/protein kinase B signal transduction /  receptor complex / carbohydrate metabolic process / endosome membrane / positive regulation of cell migration / symbiont entry into host cell / positive regulation of protein phosphorylation / G protein-coupled receptor signaling pathway / protein domain specific binding / receptor complex / carbohydrate metabolic process / endosome membrane / positive regulation of cell migration / symbiont entry into host cell / positive regulation of protein phosphorylation / G protein-coupled receptor signaling pathway / protein domain specific binding /  神経繊維 / external side of plasma membrane / 神経繊維 / external side of plasma membrane /  protein phosphorylation / positive regulation of cell population proliferation / protein-containing complex binding / regulation of DNA-templated transcription / GTP binding / positive regulation of DNA-templated transcription / extracellular exosome / protein phosphorylation / positive regulation of cell population proliferation / protein-containing complex binding / regulation of DNA-templated transcription / GTP binding / positive regulation of DNA-templated transcription / extracellular exosome /  ATP binding / ATP binding /  生体膜 / identical protein binding / 生体膜 / identical protein binding /  細胞膜 細胞膜類似検索 - 分子機能 | ||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.9 Å クライオ電子顕微鏡法 / 解像度: 3.9 Å | ||||||
 データ登録者 データ登録者 | An, W. / Hall, C. / Li, J. / Huang, A. / Wu, J. / Park, J. / Bai, X.C. / Choi, E. | ||||||
| 資金援助 |  米国, 1件 米国, 1件
| ||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2024 ジャーナル: Nat Commun / 年: 2024タイトル: Activation of the insulin receptor by insulin-like growth factor 2. 著者: Weidong An / Catherine Hall / Jie Li / Albert Hung / Jiayi Wu / Junhee Park / Liwei Wang / Xiao-Chen Bai / Eunhee Choi /  要旨: Insulin receptor (IR) controls growth and metabolism. Insulin-like growth factor 2 (IGF2) has different binding properties on two IR isoforms, mimicking insulin's function. However, the molecular ...Insulin receptor (IR) controls growth and metabolism. Insulin-like growth factor 2 (IGF2) has different binding properties on two IR isoforms, mimicking insulin's function. However, the molecular mechanism underlying IGF2-induced IR activation remains unclear. Here, we present cryo-EM structures of full-length human long isoform IR (IR-B) in both the inactive and IGF2-bound active states, and short isoform IR (IR-A) in the IGF2-bound active state. Under saturated IGF2 concentrations, both the IR-A and IR-B adopt predominantly asymmetric conformations with two or three IGF2s bound at site-1 and site-2, which differs from that insulin saturated IR forms an exclusively T-shaped symmetric conformation. IGF2 exhibits a relatively weak binding to IR site-2 compared to insulin, making it less potent in promoting full IR activation. Cell-based experiments validated the functional importance of IGF2 binding to two distinct binding sites in optimal IR signaling and trafficking. In the inactive state, the C-terminus of α-CT of IR-B contacts FnIII-2 domain of the same protomer, hindering its threading into the C-loop of IGF2, thus reducing the association rate of IGF2 with IR-B. Collectively, our studies demonstrate the activation mechanism of IR by IGF2 and reveal the molecular basis underlying the different affinity of IGF2 to IR-A and IR-B. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8u4b.cif.gz 8u4b.cif.gz | 423.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8u4b.ent.gz pdb8u4b.ent.gz | 314.1 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8u4b.json.gz 8u4b.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/u4/8u4b https://data.pdbj.org/pub/pdb/validation_reports/u4/8u4b ftp://data.pdbj.org/pub/pdb/validation_reports/u4/8u4b ftp://data.pdbj.org/pub/pdb/validation_reports/u4/8u4b | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  41877MC  8u4cC  8u4eC  8vjbC  8vjcC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
| #1: タンパク質 |  インスリン受容体 インスリン受容体分子量: 156518.328 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: INSR / 発現宿主: Homo sapiens (ヒト) / 遺伝子: INSR / 発現宿主:   Homo sapiens (ヒト) / 参照: UniProt: P06213 Homo sapiens (ヒト) / 参照: UniProt: P06213 |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Long form insulin receptor in the apo state / タイプ: COMPLEX / Entity ID: all / 由来: RECOMBINANT |
|---|---|
| 分子量 | 実験値: NO |
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 由来(組換発現) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 緩衝液 | pH: 7.5 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES |
| 試料支持 | グリッドの材料: GOLD / グリッドのサイズ: 300 divisions/in. / グリッドのタイプ: Quantifoil R1.2/1.3 |
急速凍結 | 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2600 nm / 最小 デフォーカス(公称値): 1600 nm / アライメント法: COMA FREE Bright-field microscopy / 最大 デフォーカス(公称値): 2600 nm / 最小 デフォーカス(公称値): 1600 nm / アライメント法: COMA FREE |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 電子線照射量: 60 e/Å2 フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) |
| 電子光学装置 | エネルギーフィルター名称 : GIF Bioquantum / エネルギーフィルタースリット幅: 20 eV : GIF Bioquantum / エネルギーフィルタースリット幅: 20 eV |
- 解析
解析
| EMソフトウェア |
| ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 1519317 | ||||||||||||||||||||||||||||||
| 対称性 | 点対称性 : C2 (2回回転対称 : C2 (2回回転対称 ) ) | ||||||||||||||||||||||||||||||
3次元再構成 | 解像度: 3.9 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 53302 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: RIGID BODY FIT / 空間: REAL |
 ムービー
ムービー コントローラー
コントローラー





 PDBj
PDBj











