+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8p5u | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
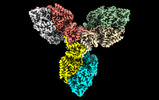
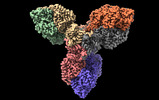
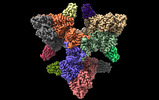
| タイトル | Single particle cryo-EM structure of homohexameric 2-oxoglutarate dehydrogenase OdhA from Corynebacterium glutamicum with Coenzyme A bound to the E2o domain | |||||||||
 要素 要素 | 2-oxoglutarate dehydrogenase E1/E2 component | |||||||||
 キーワード キーワード |  OXIDOREDUCTASE (酸化還元酵素) / OXIDOREDUCTASE (酸化還元酵素) /  2-oxoglutarate dehydrogenase / ODH 2-oxoglutarate dehydrogenase / ODH | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 oxoglutarate dehydrogenase (succinyl-transferring) / oxoglutarate dehydrogenase (succinyl-transferring) /  oxoglutarate dehydrogenase (succinyl-transferring) activity / oxoglutarate dehydrogenase (succinyl-transferring) activity /  dihydrolipoyllysine-residue succinyltransferase / dihydrolipoyllysine-residue succinyltransferase /  dihydrolipoyllysine-residue succinyltransferase activity / dihydrolipoyllysine-residue succinyltransferase activity /  thiamine pyrophosphate binding / thiamine pyrophosphate binding /  クエン酸回路 / magnesium ion binding クエン酸回路 / magnesium ion binding類似検索 - 分子機能 | |||||||||
| 生物種 |   Corynebacterium glutamicum ATCC 13032 (バクテリア) Corynebacterium glutamicum ATCC 13032 (バクテリア) | |||||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.17 Å クライオ電子顕微鏡法 / 解像度: 2.17 Å | |||||||||
 データ登録者 データ登録者 | Yang, L. / Mechaly, A.M. / Bellinzoni, M. | |||||||||
| 資金援助 |  フランス, 2件 フランス, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: High resolution cryo-EM and crystallographic snapshots of the actinobacterial two-in-one 2-oxoglutarate dehydrogenase. 著者: Lu Yang / Tristan Wagner / Ariel Mechaly / Alexandra Boyko / Eduardo M Bruch / Daniela Megrian / Francesca Gubellini / Pedro M Alzari / Marco Bellinzoni /     要旨: Actinobacteria possess unique ways to regulate the oxoglutarate metabolic node. Contrary to most organisms in which three enzymes compose the 2-oxoglutarate dehydrogenase complex (ODH), ...Actinobacteria possess unique ways to regulate the oxoglutarate metabolic node. Contrary to most organisms in which three enzymes compose the 2-oxoglutarate dehydrogenase complex (ODH), actinobacteria rely on a two-in-one protein (OdhA) in which both the oxidative decarboxylation and succinyl transferase steps are carried out by the same polypeptide. Here we describe high-resolution cryo-EM and crystallographic snapshots of representative enzymes from Mycobacterium smegmatis and Corynebacterium glutamicum, showing that OdhA is an 800-kDa homohexamer that assembles into a three-blade propeller shape. The obligate trimeric and dimeric states of the acyltransferase and dehydrogenase domains, respectively, are critical for maintaining the overall assembly, where both domains interact via subtle readjustments of their interfaces. Complexes obtained with substrate analogues, reaction products and allosteric regulators illustrate how these domains operate. Furthermore, we provide additional insights into the phosphorylation-dependent regulation of this enzymatic machinery by the signalling protein OdhI. #1:  ジャーナル: Biorxiv / 年: 2023 ジャーナル: Biorxiv / 年: 2023タイトル: High resolution cryo-EM and crystallographic snapshots of the large actinobacterial 2-oxoglutarate dehydrogenase: an all-in-one fusion with unique properties 著者: Yang, L. / Wagner, T. / Mechaly, A. / Boyko, A. / Bruch, E.M. / Megrian, D. / Gubellini, F. / Alzari, P.M. / Bellinzoni, M. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8p5u.cif.gz 8p5u.cif.gz | 1.3 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8p5u.ent.gz pdb8p5u.ent.gz | 1 MB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8p5u.json.gz 8p5u.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/p5/8p5u https://data.pdbj.org/pub/pdb/validation_reports/p5/8p5u ftp://data.pdbj.org/pub/pdb/validation_reports/p5/8p5u ftp://data.pdbj.org/pub/pdb/validation_reports/p5/8p5u | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  17453MC  8p5rC  8p5sC  8p5tC  8p5vC  8p5wC  8p5xC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 非結晶学的対称性 (NCS) | NCSドメイン:
NCSドメイン領域: Beg auth comp-ID: PRO / Beg label comp-ID: PRO / End auth comp-ID: GLU / End label comp-ID: GLU / Auth asym-ID: A / Label asym-ID: A / Auth seq-ID: 102 - 1220 / Label seq-ID: 104 - 1222
NCSアンサンブル:
|
- 要素
要素
| #1: タンパク質 | 分子量: 134972.078 Da / 分子数: 6 / 由来タイプ: 組換発現 由来: (組換発現)   Corynebacterium glutamicum ATCC 13032 (バクテリア) Corynebacterium glutamicum ATCC 13032 (バクテリア)遺伝子: odhA, Cgl1129, cg1280 / 発現宿主:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌)参照: UniProt: Q8NRC3,  oxoglutarate dehydrogenase (succinyl-transferring), oxoglutarate dehydrogenase (succinyl-transferring),  dihydrolipoyllysine-residue succinyltransferase dihydrolipoyllysine-residue succinyltransferase#2: 化合物 | ChemComp-MG / #3: 化合物 | ChemComp-COA /  補酵素A 補酵素A#4: 化合物 | ChemComp-ACO /  アセチルCoA アセチルCoA#5: 化合物 | ChemComp-TPP /  チアミンピロリン酸 チアミンピロリン酸研究の焦点であるリガンドがあるか | Y | |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Homohexameric 2-oxoglutarate dehydrogenase / タイプ: COMPLEX / Entity ID: #1 / 由来: RECOMBINANT | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 | 値: 0.808 MDa / 実験値: NO | ||||||||||||
| 由来(天然) | 生物種:   Corynebacterium glutamicum ATCC 13032 (バクテリア) Corynebacterium glutamicum ATCC 13032 (バクテリア) | ||||||||||||
| 由来(組換発現) | 生物種:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌) | ||||||||||||
| 緩衝液 | pH: 7.5 | ||||||||||||
| 緩衝液成分 |
| ||||||||||||
| 試料 | 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES | ||||||||||||
| 試料支持 | グリッドの材料: GOLD / グリッドのサイズ: 300 divisions/in. / グリッドのタイプ: UltrAuFoil R1.2/1.3 | ||||||||||||
急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE / 湿度: 100 % / 凍結前の試料温度: 277 K |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 倍率(公称値): 105000 X / 最大 デフォーカス(公称値): 2000 nm / 最小 デフォーカス(公称値): 800 nm Bright-field microscopy / 倍率(公称値): 105000 X / 最大 デフォーカス(公称値): 2000 nm / 最小 デフォーカス(公称値): 800 nm |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 平均露光時間: 1.82 sec. / 電子線照射量: 40 e/Å2 フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 撮影したグリッド数: 1 / 実像数: 12202 / 詳細: Each image was composed of 40 frames |
- 解析
解析
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 点対称性 : D3 (2回x3回 2面回転対称 : D3 (2回x3回 2面回転対称 ) ) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
3次元再構成 | 解像度: 2.17 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 1849406 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: FLEXIBLE FIT / 空間: REAL | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | PDB-ID: 8P5T Accession code: 8P5T / Source name: PDB / タイプ: experimental model | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | 解像度: 2.17→2.17 Å / Cor.coef. Fo:Fc: 0.878 / SU B: 5.148 / SU ML: 0.115 / ESU R: 0.125 立体化学のターゲット値: MAXIMUM LIKELIHOOD WITH PHASES 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 溶媒モデル: PARAMETERS FOR MASK CACLULATION | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 42.016 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: 1 / 合計: 52878 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー




 PDBj
PDBj













