+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8jj5 | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Porcine uroplakin complex | ||||||||||||||||||
 要素 要素 |
| ||||||||||||||||||
 キーワード キーワード |  MEMBRANE PROTEIN (膜タンパク質) / Urothelium / asymmetric unit membrane / urinary bladder / MEMBRANE PROTEIN (膜タンパク質) / Urothelium / asymmetric unit membrane / urinary bladder /  urinary tract infection (尿路感染症) / urinary tract infection (尿路感染症) /  lipid raft (脂質ラフト) lipid raft (脂質ラフト) | ||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報apical plasma membrane urothelial plaque / urea transport / urinary bladder development / water transport / sodium ion homeostasis / potassium ion homeostasis / epithelial cell differentiation /  kidney development / cell morphogenesis / kidney development / cell morphogenesis /  生体膜 / 生体膜 /  細胞膜 細胞膜類似検索 - 分子機能 | ||||||||||||||||||
| 生物種 |   Sus scrofa (ブタ) Sus scrofa (ブタ) | ||||||||||||||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.5 Å クライオ電子顕微鏡法 / 解像度: 3.5 Å | ||||||||||||||||||
 データ登録者 データ登録者 | Oda, T. / Yanagisawa, H. / Kikkawa, M. | ||||||||||||||||||
| 資金援助 |  日本, 5件 日本, 5件
| ||||||||||||||||||
 引用 引用 |  ジャーナル: Commun Biol / 年: 2023 ジャーナル: Commun Biol / 年: 2023タイトル: Cryo-EM elucidates the uroplakin complex structure within liquid-crystalline lipids in the porcine urothelial membrane. 著者: Haruaki Yanagisawa / Yoshihiro Kita / Toshiyuki Oda / Masahide Kikkawa /  要旨: The urothelium, a distinct epithelial tissue lining the urinary tract, serves as an essential component in preserving urinary tract integrity and thwarting infections. The asymmetric unit membrane ...The urothelium, a distinct epithelial tissue lining the urinary tract, serves as an essential component in preserving urinary tract integrity and thwarting infections. The asymmetric unit membrane (AUM), primarily composed of the uroplakin complex, constitutes a critical permeability barrier in fulfilling this role. However, the molecular architectures of both the AUM and the uroplakin complex have remained enigmatic due to the paucity of high-resolution structural data. In this study, we utilized cryo-electron microscopy to elucidate the three-dimensional structure of the uroplakin complex within the porcine AUM. While the global resolution achieved was 3.5 Å, we acknowledge that due to orientation bias, the resolution in the vertical direction was determined to be 6.3 Å. Our findings unveiled that the uroplakin complexes are situated within hexagonally arranged crystalline lipid membrane domains, rich in hexosylceramides. Moreover, our research rectifies a misconception in a previous model by confirming the existence of a domain initially believed to be absent, and pinpointing the accurate location of a crucial Escherichia coli binding site implicated in urinary tract infections. These discoveries offer valuable insights into the molecular underpinnings governing the permeability barrier function of the urothelium and the orchestrated lipid phase formation within the plasma membrane. | ||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8jj5.cif.gz 8jj5.cif.gz | 300.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8jj5.ent.gz pdb8jj5.ent.gz | 245.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8jj5.json.gz 8jj5.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/jj/8jj5 https://data.pdbj.org/pub/pdb/validation_reports/jj/8jj5 ftp://data.pdbj.org/pub/pdb/validation_reports/jj/8jj5 ftp://data.pdbj.org/pub/pdb/validation_reports/jj/8jj5 | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  36340MC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-タンパク質 , 4種, 4分子 ABCD
| #1: タンパク質 |  テトラスパニン テトラスパニン分子量: 28747.469 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Sus scrofa (ブタ) / 器官: Urinary bladder / 組織: Epithelium Sus scrofa (ブタ) / 器官: Urinary bladder / 組織: Epithelium 上皮細胞 / 参照: UniProt: Q06AT5 上皮細胞 / 参照: UniProt: Q06AT5 |
|---|---|
| #2: タンパク質 | 分子量: 19299.869 Da / 分子数: 1 / 由来タイプ: 天然 / 詳細: NCBI Reference Sequence: NP_999177.1 / 由来: (天然)   Sus scrofa (ブタ) / 器官: Urinary bladder / 組織: Epithelium Sus scrofa (ブタ) / 器官: Urinary bladder / 組織: Epithelium 上皮細胞 上皮細胞 |
| #3: タンパク質 | 分子量: 30339.234 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Sus scrofa (ブタ) / 器官: Urinary bladder / 組織: Epithelium Sus scrofa (ブタ) / 器官: Urinary bladder / 組織: Epithelium 上皮細胞 / 参照: UniProt: A0A287AEW0 上皮細胞 / 参照: UniProt: A0A287AEW0 |
| #4: タンパク質 | 分子量: 29857.492 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Sus scrofa (ブタ) / 器官: Urinary bladder / 組織: Epithelium Sus scrofa (ブタ) / 器官: Urinary bladder / 組織: Epithelium 上皮細胞 / 参照: UniProt: Q06AT4 上皮細胞 / 参照: UniProt: Q06AT4 |
-糖 , 4種, 6分子 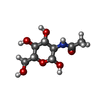
| #5: 多糖 |  オリゴ糖 / 分子量: 424.401 Da / 分子数: 2 / 由来タイプ: 組換発現 オリゴ糖 / 分子量: 424.401 Da / 分子数: 2 / 由来タイプ: 組換発現#6: 多糖 | beta-D-mannopyranose-(1-4)-2-acetamido-2-deoxy-beta-D-glucopyranose-(1-4)-2-acetamido-2-deoxy-beta- ...beta-D-mannopyranose-(1-4)-2-acetamido-2-deoxy-beta-D-glucopyranose-(1-4)-2-acetamido-2-deoxy-beta-D-glucopyranose |  オリゴ糖 / 分子量: 586.542 Da / 分子数: 1 / 由来タイプ: 組換発現 オリゴ糖 / 分子量: 586.542 Da / 分子数: 1 / 由来タイプ: 組換発現#7: 多糖 | alpha-D-mannopyranose-(1-3)-[alpha-D-mannopyranose-(1-6)]beta-D-mannopyranose-(1-4)-2-acetamido-2- ...alpha-D-mannopyranose-(1-3)-[alpha-D-mannopyranose-(1-6)]beta-D-mannopyranose-(1-4)-2-acetamido-2-deoxy-beta-D-glucopyranose-(1-4)-2-acetamido-2-deoxy-beta-D-glucopyranose |  オリゴ糖 / 分子量: 910.823 Da / 分子数: 1 / 由来タイプ: 組換発現 オリゴ糖 / 分子量: 910.823 Da / 分子数: 1 / 由来タイプ: 組換発現#8: 糖 |  N-アセチルグルコサミン N-アセチルグルコサミン |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | N |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: 2D ARRAY / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Uroplakin complex / タイプ: ORGANELLE OR CELLULAR COMPONENT 詳細: Asymmetric unit membrane isolated by sarkosyl extraction Entity ID: #1-#4 / 由来: NATURAL |
|---|---|
| 分子量 | 実験値: NO |
| 由来(天然) | 生物種:   Sus scrofa (ブタ) / 器官: Urinary bladder / 組織: Urothelium Sus scrofa (ブタ) / 器官: Urinary bladder / 組織: Urothelium |
| 緩衝液 | pH: 7.4 |
| 試料 | 濃度: 0.05 mg/ml / 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES |
| 試料支持 | グリッドの材料: COPPER/RHODIUM / グリッドのサイズ: 200 divisions/in. / グリッドのタイプ: Quantifoil R1.2/1.3 |
急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE / 湿度: 99 % / 凍結前の試料温度: 277 K |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 倍率(公称値): 64000 X / 最大 デフォーカス(公称値): 4500 nm / 最小 デフォーカス(公称値): 1100 nm / Cs Bright-field microscopy / 倍率(公称値): 64000 X / 最大 デフォーカス(公称値): 4500 nm / 最小 デフォーカス(公称値): 1100 nm / Cs : 2.7 mm : 2.7 mm |
| 試料ホルダ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 電子線照射量: 50 e/Å2 / フィルム・検出器のモデル: GATAN K3 (6k x 4k) |
- 解析
解析
| EMソフトウェア |
| |||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | |||||||||||||||||||||||||||
| 対称性 | 点対称性 : C6 (6回回転対称 : C6 (6回回転対称 ) ) | |||||||||||||||||||||||||||
3次元再構成 | 解像度: 3.5 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 609567 / 対称性のタイプ: POINT | |||||||||||||||||||||||||||
| 原子モデル構築 | Source name: AlphaFold / タイプ: in silico model | |||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー



 PDBj
PDBj
