+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8edu | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
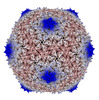
| タイトル | Mycobacteriophage Muddy capsid | |||||||||||||||
 要素 要素 | Capsid カプシド カプシド | |||||||||||||||
 キーワード キーワード |  VIRUS (ウイルス) / VIRUS (ウイルス) /  Bacteriophage (ファージ) / Bacteriophage (ファージ) /  Mycobacteriophage / HK97-fold / Mycobacteriophage / HK97-fold /  Capsid (カプシド) Capsid (カプシド) | |||||||||||||||
| 機能・相同性 | Phage capsid / Phage capsid family /  カプシド カプシド 機能・相同性情報 機能・相同性情報 | |||||||||||||||
| 生物種 |  Mycobacterium phage Muddy (ファージ) Mycobacterium phage Muddy (ファージ) | |||||||||||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.7 Å クライオ電子顕微鏡法 / 解像度: 2.7 Å | |||||||||||||||
 データ登録者 データ登録者 | Freeman, K.G. / White, S.J. / Huet, A. / Conway, J.F. | |||||||||||||||
| 資金援助 |  米国, 4件 米国, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Structure / 年: 2023 ジャーナル: Structure / 年: 2023タイトル: A structural dendrogram of the actinobacteriophage major capsid proteins provides important structural insights into the evolution of capsid stability. 著者: Jennifer M Podgorski / Krista Freeman / Sophia Gosselin / Alexis Huet / James F Conway / Mary Bird / John Grecco / Shreya Patel / Deborah Jacobs-Sera / Graham Hatfull / Johann Peter Gogarten ...著者: Jennifer M Podgorski / Krista Freeman / Sophia Gosselin / Alexis Huet / James F Conway / Mary Bird / John Grecco / Shreya Patel / Deborah Jacobs-Sera / Graham Hatfull / Johann Peter Gogarten / Janne Ravantti / Simon J White /   要旨: Many double-stranded DNA viruses, including tailed bacteriophages (phages) and herpesviruses, use the HK97-fold in their major capsid protein to make the capsomers of the icosahedral viral capsid. ...Many double-stranded DNA viruses, including tailed bacteriophages (phages) and herpesviruses, use the HK97-fold in their major capsid protein to make the capsomers of the icosahedral viral capsid. After the genome packaging at near-crystalline densities, the capsid is subjected to a major expansion and stabilization step that allows it to withstand environmental stresses and internal high pressure. Several different mechanisms for stabilizing the capsid have been structurally characterized, but how these mechanisms have evolved is still not understood. Using cryo-EM structure determination of 10 capsids, structural comparisons, phylogenetic analyses, and Alphafold predictions, we have constructed a detailed structural dendrogram describing the evolution of capsid structural stability within the actinobacteriophages. We show that the actinobacteriophage major capsid proteins can be classified into 15 groups based upon their HK97-fold. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8edu.cif.gz 8edu.cif.gz | 674.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8edu.ent.gz pdb8edu.ent.gz | 575.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  8edu.json.gz 8edu.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ed/8edu https://data.pdbj.org/pub/pdb/validation_reports/ed/8edu ftp://data.pdbj.org/pub/pdb/validation_reports/ed/8edu ftp://data.pdbj.org/pub/pdb/validation_reports/ed/8edu | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  28039MC  8e16C  8eb4C  8ec2C  8ec8C  8eciC  8ecjC  8eckC  8ecnC  8ecoC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 | x 60
|
| 2 |
|
| 3 | x 5
|
| 4 | x 6
|
| 5 | 
|
| 対称性 | 点対称性: (シェーンフリース記号 : I (正20面体型対称 : I (正20面体型対称 )) )) |
- 要素
要素
| #1: タンパク質 |  カプシド / gp9 カプシド / gp9分子量: 34670.102 Da / 分子数: 7 / 由来タイプ: 天然 / 由来: (天然)  Mycobacterium phage Muddy (ファージ) / 参照: UniProt: S5Y5B1 Mycobacterium phage Muddy (ファージ) / 参照: UniProt: S5Y5B1 |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Mycobacterium phage Muddy / タイプ: VIRUS 詳細: Phage Muddy particles generated by amplification on bacterial host and purification via CsCl gradient. Entity ID: all / 由来: NATURAL | |||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 | 実験値: NO | |||||||||||||||||||||||||
| 由来(天然) | 生物種:  Mycobacterium phage Muddy (ファージ) Mycobacterium phage Muddy (ファージ) | |||||||||||||||||||||||||
| ウイルスについての詳細 | 中空か: NO / エンベロープを持つか: NO / 単離: STRAIN / タイプ: VIRION | |||||||||||||||||||||||||
| 天然宿主 | 生物種: Mycolicibacterium smegmatis MC2 155 | |||||||||||||||||||||||||
| ウイルス殻 | 直径: 710 nm / 三角数 (T数): 7 | |||||||||||||||||||||||||
| 緩衝液 | pH: 7.5 | |||||||||||||||||||||||||
| 緩衝液成分 |
| |||||||||||||||||||||||||
| 試料 | 濃度: 10 mg/ml / 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES | |||||||||||||||||||||||||
| 試料支持 | グリッドの材料: COPPER / グリッドのサイズ: 300 divisions/in. / グリッドのタイプ: Quantifoil R2/1 | |||||||||||||||||||||||||
急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE-PROPANE / 湿度: 100 % / 凍結前の試料温度: 283 K |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 倍率(公称値): 75000 X / 最大 デフォーカス(公称値): 2500 nm / 最小 デフォーカス(公称値): 1000 nm / Cs Bright-field microscopy / 倍率(公称値): 75000 X / 最大 デフォーカス(公称値): 2500 nm / 最小 デフォーカス(公称値): 1000 nm / Cs : 2.7 mm / C2レンズ絞り径: 50 µm / アライメント法: COMA FREE : 2.7 mm / C2レンズ絞り径: 50 µm / アライメント法: COMA FREE |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 平均露光時間: 40 sec. / 電子線照射量: 50 e/Å2 / 検出モード: COUNTING フィルム・検出器のモデル: FEI FALCON III (4k x 4k) 撮影したグリッド数: 1 / 実像数: 1027 |
| 画像スキャン | サンプリングサイズ: 14 µm / 横: 4096 / 縦: 4096 |
- 解析
解析
| EMソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
CTF補正 | 詳細: Standard CTF correction inside RELION's reconstruction. タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | |||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 28207 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 点対称性 : I (正20面体型対称 : I (正20面体型対称 ) ) | |||||||||||||||||||||||||||||||||||||||||||||||||||||||
3次元再構成 | 解像度: 2.7 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 25244 / 対称性のタイプ: POINT | |||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: AB INITIO MODEL 詳細: Amino acid sequence built into the map for a single major capsid protein and refined with Phenix. Model then used for rest of asymmetric unit and refined with Phenix. Final step involved using Isolde. |
 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj

