+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 7qv9 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | CryoEM structure of bacterial transcription intermediate complex mediated by activator PspF | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード |  TRANSCRIPTION (転写 (生物学)) / TRANSCRIPTION (転写 (生物学)) /  RNA polymerase (RNAポリメラーゼ) / RNA polymerase (RNAポリメラーゼ) /  AAA protein / AAA protein /  transcription regulation / transcription regulation /  cryoEM (低温電子顕微鏡法) cryoEM (低温電子顕微鏡法) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 RNA polymerase complex / DNA-binding transcription activator activity / submerged biofilm formation / cellular response to cell envelope stress / cytosolic DNA-directed RNA polymerase complex / regulation of DNA-templated transcription initiation / bacterial-type flagellum assembly / RNA polymerase complex / DNA-binding transcription activator activity / submerged biofilm formation / cellular response to cell envelope stress / cytosolic DNA-directed RNA polymerase complex / regulation of DNA-templated transcription initiation / bacterial-type flagellum assembly /  sigma factor activity / bacterial-type flagellum-dependent cell motility / nitrate assimilation ... sigma factor activity / bacterial-type flagellum-dependent cell motility / nitrate assimilation ... RNA polymerase complex / DNA-binding transcription activator activity / submerged biofilm formation / cellular response to cell envelope stress / cytosolic DNA-directed RNA polymerase complex / regulation of DNA-templated transcription initiation / bacterial-type flagellum assembly / RNA polymerase complex / DNA-binding transcription activator activity / submerged biofilm formation / cellular response to cell envelope stress / cytosolic DNA-directed RNA polymerase complex / regulation of DNA-templated transcription initiation / bacterial-type flagellum assembly /  sigma factor activity / bacterial-type flagellum-dependent cell motility / nitrate assimilation / sigma factor activity / bacterial-type flagellum-dependent cell motility / nitrate assimilation /  nucleotidyltransferase activity / transcription elongation factor complex / nucleotidyltransferase activity / transcription elongation factor complex /  DNA-directed RNA polymerase complex / regulation of DNA-templated transcription elongation / transcription antitermination / DNA-directed RNA polymerase complex / regulation of DNA-templated transcription elongation / transcription antitermination /  運動性 / DNA-templated transcription initiation / 運動性 / DNA-templated transcription initiation /  ribonucleoside binding / DNA-directed 5'-3' RNA polymerase activity / ribonucleoside binding / DNA-directed 5'-3' RNA polymerase activity /  ポリメラーゼ / response to heat / protein-containing complex assembly / intracellular iron ion homeostasis / ポリメラーゼ / response to heat / protein-containing complex assembly / intracellular iron ion homeostasis /  protein dimerization activity / response to antibiotic / magnesium ion binding / protein dimerization activity / response to antibiotic / magnesium ion binding /  DNA binding / zinc ion binding / DNA binding / zinc ion binding /  生体膜 / 生体膜 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | ||||||
| 生物種 |   Escherichia coli K-12 (大腸菌) Escherichia coli K-12 (大腸菌)  Klebsiella pneumoniae (肺炎桿菌) Klebsiella pneumoniae (肺炎桿菌) | ||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.5 Å クライオ電子顕微鏡法 / 解像度: 3.5 Å | ||||||
 データ登録者 データ登録者 | Ye, F.Z. / Zhang, X.D. | ||||||
| 資金援助 |  英国, 1件 英国, 1件
| ||||||
 引用 引用 |  ジャーナル: Sci Adv / 年: 2022 ジャーナル: Sci Adv / 年: 2022タイトル: Mechanisms of DNA opening revealed in AAA+ transcription complex structures. 著者: Fuzhou Ye / Forson Gao / Xiaojiao Liu / Martin Buck / Xiaodong Zhang /  要旨: Gene transcription is carried out by RNA polymerase (RNAP) and requires the conversion of the initial closed promoter complex, where DNA is double stranded, to a transcription-competent open promoter ...Gene transcription is carried out by RNA polymerase (RNAP) and requires the conversion of the initial closed promoter complex, where DNA is double stranded, to a transcription-competent open promoter complex, where DNA is opened up. In bacteria, RNAP relies on σ factors for its promoter specificities. Using a special form of sigma factor (σ), which forms a stable closed complex and requires its activator that belongs to the AAA+ ATPases (ATPases associated with diverse cellular activities), we obtained cryo-electron microscopy structures of transcription initiation complexes that reveal a previously unidentified process of DNA melting opening. The σ amino terminus threads through the locally opened up DNA and then becomes enclosed by the AAA+ hexameric ring in the activator-bound intermediate complex. Our structures suggest how ATP hydrolysis by the AAA+ activator could remove the σ inhibition while helping to open up DNA, using σ amino-terminal peptide as a pry bar. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  7qv9.cif.gz 7qv9.cif.gz | 1002.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb7qv9.ent.gz pdb7qv9.ent.gz | 798.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  7qv9.json.gz 7qv9.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/qv/7qv9 https://data.pdbj.org/pub/pdb/validation_reports/qv/7qv9 ftp://data.pdbj.org/pub/pdb/validation_reports/qv/7qv9 ftp://data.pdbj.org/pub/pdb/validation_reports/qv/7qv9 | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  14171MC  7qwpC  7qxiC M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-DNA-directed RNA polymerase subunit ... , 4種, 5分子 ABCDE
| #1: タンパク質 |  ポリメラーゼ / RNAP subunit alpha / RNA polymerase subunit alpha / Transcriptase subunit alpha ポリメラーゼ / RNAP subunit alpha / RNA polymerase subunit alpha / Transcriptase subunit alpha分子量: 36558.680 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)   Escherichia coli K-12 (大腸菌) / 株: K12 / 遺伝子: rpoA, pez, phs, sez, b3295, JW3257 / プラスミド: pGEXABC / 発現宿主: Escherichia coli K-12 (大腸菌) / 株: K12 / 遺伝子: rpoA, pez, phs, sez, b3295, JW3257 / プラスミド: pGEXABC / 発現宿主:   Escherichia coli BL21(DE3) (大腸菌) / 株 (発現宿主): BL21(DE3) / Variant (発現宿主): Escherichia coli / 参照: UniProt: P0A7Z4, Escherichia coli BL21(DE3) (大腸菌) / 株 (発現宿主): BL21(DE3) / Variant (発現宿主): Escherichia coli / 参照: UniProt: P0A7Z4,  ポリメラーゼ ポリメラーゼ#2: タンパク質 | |  ポリメラーゼ / RNAP subunit beta / RNA polymerase subunit beta / Transcriptase subunit beta ポリメラーゼ / RNAP subunit beta / RNA polymerase subunit beta / Transcriptase subunit beta分子量: 150820.875 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Escherichia coli K-12 (大腸菌) / 株: K12 Escherichia coli K-12 (大腸菌) / 株: K12遺伝子: rpoB, groN, nitB, rif, ron, stl, stv, tabD, b3987, JW3950 プラスミド: pGEXABC / 発現宿主:   Escherichia coli BL21(DE3) (大腸菌) / 株 (発現宿主): BL21(DE3) / Variant (発現宿主): Escherichia coli / 参照: UniProt: P0A8V2, Escherichia coli BL21(DE3) (大腸菌) / 株 (発現宿主): BL21(DE3) / Variant (発現宿主): Escherichia coli / 参照: UniProt: P0A8V2,  ポリメラーゼ ポリメラーゼ#3: タンパク質 | |  ポリメラーゼ / RNAP subunit beta' / RNA polymerase subunit beta' / Transcriptase subunit beta' ポリメラーゼ / RNAP subunit beta' / RNA polymerase subunit beta' / Transcriptase subunit beta'分子量: 155366.781 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Escherichia coli K-12 (大腸菌) / 株: K12 / 遺伝子: rpoC, tabB, b3988, JW3951 / プラスミド: pGEXABC / 発現宿主: Escherichia coli K-12 (大腸菌) / 株: K12 / 遺伝子: rpoC, tabB, b3988, JW3951 / プラスミド: pGEXABC / 発現宿主:   Escherichia coli BL21(DE3) (大腸菌) / 株 (発現宿主): BL21(DE3) / Variant (発現宿主): Escherichia coli / 参照: UniProt: P0A8T7, Escherichia coli BL21(DE3) (大腸菌) / 株 (発現宿主): BL21(DE3) / Variant (発現宿主): Escherichia coli / 参照: UniProt: P0A8T7,  ポリメラーゼ ポリメラーゼ#4: タンパク質 | |  ポリメラーゼ / RNAP omega subunit / RNA polymerase omega subunit / Transcriptase subunit omega ポリメラーゼ / RNAP omega subunit / RNA polymerase omega subunit / Transcriptase subunit omega分子量: 10249.547 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Escherichia coli K-12 (大腸菌) / 株: K12 / 遺伝子: rpoZ, b3649, JW3624 / プラスミド: pGEXABC / 発現宿主: Escherichia coli K-12 (大腸菌) / 株: K12 / 遺伝子: rpoZ, b3649, JW3624 / プラスミド: pGEXABC / 発現宿主:   Escherichia coli BL21(DE3) (大腸菌) / 株 (発現宿主): BL21(DE3) / Variant (発現宿主): Escherichia coli / 参照: UniProt: P0A800, Escherichia coli BL21(DE3) (大腸菌) / 株 (発現宿主): BL21(DE3) / Variant (発現宿主): Escherichia coli / 参照: UniProt: P0A800,  ポリメラーゼ ポリメラーゼ |
|---|
-DNA鎖 , 2種, 2分子 NT
| #5: DNA鎖 | 分子量: 19439.387 Da / 分子数: 1 / 由来タイプ: 合成 / 由来: (合成)   Klebsiella pneumoniae (肺炎桿菌) Klebsiella pneumoniae (肺炎桿菌) |
|---|---|
| #6: DNA鎖 | 分子量: 19404.410 Da / 分子数: 1 / 由来タイプ: 合成 / 由来: (合成)   Klebsiella pneumoniae (肺炎桿菌) Klebsiella pneumoniae (肺炎桿菌) |
-タンパク質 , 2種, 7分子 abcdfeM
| #7: タンパク質 | 分子量: 33187.777 Da / 分子数: 6 / 由来タイプ: 組換発現 / 由来: (組換発現)   Escherichia coli K-12 (大腸菌) / 遺伝子: pspF / プラスミド: pET28b-PspF / 発現宿主: Escherichia coli K-12 (大腸菌) / 遺伝子: pspF / プラスミド: pET28b-PspF / 発現宿主:   Escherichia coli BL21(DE3) (大腸菌) / 株 (発現宿主): BL21(DE3) / Variant (発現宿主): Escherichia coli Escherichia coli BL21(DE3) (大腸菌) / 株 (発現宿主): BL21(DE3) / Variant (発現宿主): Escherichia coli#8: タンパク質 | | 分子量: 53978.656 Da / 分子数: 1 / 由来タイプ: 組換発現 詳細: MGSSHHHHHHSSGLVPRGSHMKQGLQLRLSQQLAMTPQLQQAIRLLQLSTLELQQELQQALESNPLLEQTDLHDEVEAKEVEDRESLDTVDALEQKEMPD ELPLDASWDEIYTAGTPSGNGVDYQDDELPVYQGETTQTLQDYLMWQVELTPFTDTDRAIATSIVDAVDDTGYLTIQIED ...詳細: MGSSHHHHHHSSGLVPRGSHMKQGLQLRLSQQLAMTPQLQQAIRLLQLSTLELQQELQQALESNPLLEQTDLHDEVEAKEVEDRESLDTVDALEQKEMPD ELPLDASWDEIYTAGTPSGNGVDYQDDELPVYQGETTQTLQDYLMWQVELTPFTDTDRAIATSIVDAVDDTGYLTIQIED IVDSIGDDEIGLEEVEAVLKRIQRFDPVGVAAKDLRDCLLIQLSQFAKETPWLEEARLIISDHLDLLANHDFRTLMRVTR LKEEVLKEAVNLIQSLDPRPGQSIQTSEPEYVIPDVLVRKVSGRWTVELNADSIPRLKINQQYAAMGNSARNDADGQFIR SNLQEARWLIKSLESRNDTLLRVSRCIVEQQQAFFEQGEEYMKPMVLADIAQAVEMHESTISRVTTQKYLHSPRGIFELK YFFSSHVNTEGGGEASSTAIRALVKKLIAAENPAKPLSDSKLTSMLSEQGIMVARRTVAKYRESLSIPPSNQRKQLV 由来: (組換発現)   Klebsiella pneumoniae (肺炎桿菌) Klebsiella pneumoniae (肺炎桿菌)遺伝子: rpoN, A7B01_01570, B4U61_10775, B5L96_24890, BANRA_03272, BL124_00020515, BN49_0508, BS595_02095, C3F39_17700, DDJ63_09965, DRB11_15360, EAO17_11045, FXN67_08250, G5637_17550, G7Z27_02650, ...遺伝子: rpoN, A7B01_01570, B4U61_10775, B5L96_24890, BANRA_03272, BL124_00020515, BN49_0508, BS595_02095, C3F39_17700, DDJ63_09965, DRB11_15360, EAO17_11045, FXN67_08250, G5637_17550, G7Z27_02650, GJJ08_002620, GJJ12_002565, GNE24_14685, GNG14_08115, GPZ86_02700, HV479_13895, NCTC13465_03419, NCTC3279_03959, NCTC5047_03164, SAMEA3499874_03190, SAMEA3499901_03507, SAMEA3500057_00079, SAMEA3512100_03793, SAMEA3538828_04129, SAMEA3649733_03935, SAMEA3649758_00080, SAMEA3720909_03353, SAMEA3727630_04208, SAMEA3727643_03388, SAMEA3727679_00080, SAMEA3729663_03804, SAMEA4364603_00630, SAMEA4873632_04262 プラスミド: pET28b-sigma54 / 発現宿主:   Escherichia coli BL21(DE3) (大腸菌) / 株 (発現宿主): Escherichia coli BL21(DE3 / 参照: UniProt: A0A0N9UTC1 Escherichia coli BL21(DE3) (大腸菌) / 株 (発現宿主): Escherichia coli BL21(DE3 / 参照: UniProt: A0A0N9UTC1 |
|---|
-非ポリマー , 2種, 9分子 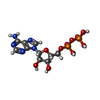


| #9: 化合物 | ChemComp-ADP /  アデノシン二リン酸 アデノシン二リン酸#10: 化合物 | ChemComp-AF3 /  フッ化アルミニウム フッ化アルミニウム |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Bacterial transcription intermediate complex mediated by PspF activator protein タイプ: COMPLEX / Entity ID: #1-#8 / 由来: MULTIPLE SOURCES | |||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 分子量 | 値: 0.71 MDa / 実験値: NO | |||||||||||||||||||||||||
| 緩衝液 | pH: 8 | |||||||||||||||||||||||||
| 緩衝液成分 |
| |||||||||||||||||||||||||
| 試料 | 濃度: 3.5 mg/ml / 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES | |||||||||||||||||||||||||
| 試料支持 | グリッドの材料: COPPER / グリッドのサイズ: 200 divisions/in. / グリッドのタイプ: Quantifoil R2/2 | |||||||||||||||||||||||||
急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE / 湿度: 95 % / 凍結前の試料温度: 277 K |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 倍率(公称値): 81000 X / 最大 デフォーカス(公称値): 2600 nm / 最小 デフォーカス(公称値): 1400 nm / Cs Bright-field microscopy / 倍率(公称値): 81000 X / 最大 デフォーカス(公称値): 2600 nm / 最小 デフォーカス(公称値): 1400 nm / Cs : 2.7 mm / C2レンズ絞り径: 50 µm / アライメント法: COMA FREE : 2.7 mm / C2レンズ絞り径: 50 µm / アライメント法: COMA FREE |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER |
| 撮影 | 平均露光時間: 4.1 sec. / 電子線照射量: 50 e/Å2 / フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 実像数: 14780 |
| 電子光学装置 | エネルギーフィルター名称 : GIF Bioquantum / エネルギーフィルタースリット幅: 20 eV : GIF Bioquantum / エネルギーフィルタースリット幅: 20 eV |
| 画像スキャン | 横: 5760 / 縦: 4092 |
- 解析
解析
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 2000000 | ||||||||||||||||||||||||||||||||||||||||
| 対称性 | 点対称性 : C1 (非対称) : C1 (非対称) | ||||||||||||||||||||||||||||||||||||||||
3次元再構成 | 解像度: 3.5 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 33285 / アルゴリズム: FOURIER SPACE / クラス平均像の数: 1 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | プロトコル: RIGID BODY FIT / 空間: REAL | ||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | PDB-ID: 5NSS |
 ムービー
ムービー コントローラー
コントローラー









 PDBj
PDBj











































