+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 6h9j | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Factor Inhibiting HIF (FIH) in complex with zinc, NOG and TRPV3 (229-255) | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード |  GENE REGULATION (遺伝子発現の調節) / NON-HEME / GENE REGULATION (遺伝子発現の調節) / NON-HEME /  DIOXYGENASE / DIOXYGENASE /  OXYGENASE (酸素添加酵素) / METAL-BINDING / OXYGENASE (酸素添加酵素) / METAL-BINDING /  TRANSCRIPTION (転写 (生物学)) / DSBH / FACIAL TRIAD / ASPARAGINYL/ASPARTYL HYDROXYLASE / TRANSCRIPTION (転写 (生物学)) / DSBH / FACIAL TRIAD / ASPARAGINYL/ASPARTYL HYDROXYLASE /  SIGNALING / ARD / SIGNALING / ARD /  BETA-HYDROXYLATION (ヒドロキシル化) / ACTIVATOR-INHIBITOR / OXIDOREDUCTASE-PEPTIDE COMPLEX / OXIDOREDUCTASE ANKYRIN / CHANNEL / BETA-HYDROXYLATION (ヒドロキシル化) / ACTIVATOR-INHIBITOR / OXIDOREDUCTASE-PEPTIDE COMPLEX / OXIDOREDUCTASE ANKYRIN / CHANNEL /  PROTEIN BINDING (タンパク質) / PROTEIN BINDING (タンパク質) /  ION CHANNEL (イオンチャネル) / TRPV CHANNEL / ION CHANNEL (イオンチャネル) / TRPV CHANNEL /  MEMBRANE PROTEIN (膜タンパク質) MEMBRANE PROTEIN (膜タンパク質) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 hypoxia-inducible factor-asparagine dioxygenase / hypoxia-inducible factor-asparagine dioxygenase /  : / [protein]-asparagine 3-dioxygenase activity / peptidyl-histidine dioxygenase activity / negative regulation of hair cycle / peptidyl-aspartic acid 3-dioxygenase activity / Cellular response to hypoxia / : / [protein]-asparagine 3-dioxygenase activity / peptidyl-histidine dioxygenase activity / negative regulation of hair cycle / peptidyl-aspartic acid 3-dioxygenase activity / Cellular response to hypoxia /  carboxylic acid binding / positive regulation of vasculogenesis / carboxylic acid binding / positive regulation of vasculogenesis /  ankyrin repeat binding ... ankyrin repeat binding ... hypoxia-inducible factor-asparagine dioxygenase / hypoxia-inducible factor-asparagine dioxygenase /  : / [protein]-asparagine 3-dioxygenase activity / peptidyl-histidine dioxygenase activity / negative regulation of hair cycle / peptidyl-aspartic acid 3-dioxygenase activity / Cellular response to hypoxia / : / [protein]-asparagine 3-dioxygenase activity / peptidyl-histidine dioxygenase activity / negative regulation of hair cycle / peptidyl-aspartic acid 3-dioxygenase activity / Cellular response to hypoxia /  carboxylic acid binding / positive regulation of vasculogenesis / carboxylic acid binding / positive regulation of vasculogenesis /  ankyrin repeat binding / ankyrin repeat binding /  oxygen sensor activity / Notch binding / oxygen sensor activity / Notch binding /  TRPチャネル / response to temperature stimulus / negative regulation of Notch signaling pathway / positive regulation of calcium ion import / TRPチャネル / response to temperature stimulus / negative regulation of Notch signaling pathway / positive regulation of calcium ion import /  NF-kappaB binding / positive regulation of myoblast differentiation / NF-kappaB binding / positive regulation of myoblast differentiation /  ferrous iron binding / calcium ion transmembrane transport / ferrous iron binding / calcium ion transmembrane transport /  calcium channel activity / transcription corepressor activity / calcium channel activity / transcription corepressor activity /  receptor complex / perinuclear region of cytoplasm / protein homodimerization activity / zinc ion binding / receptor complex / perinuclear region of cytoplasm / protein homodimerization activity / zinc ion binding /  核質 / identical protein binding / 核質 / identical protein binding /  細胞膜 / 細胞膜 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | ||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.83 Å 分子置換 / 解像度: 1.83 Å | ||||||
 データ登録者 データ登録者 | Leissing, T.M. / Clifton, I.J. / Saward, B.G. / Lu, X. / Hopkinson, R.J. / Schofield, C.J. | ||||||
| 資金援助 |  英国, 1件 英国, 1件
| ||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Factor Inhibiting HIF (FIH) in complex with zinc, NOG and TRPV3 (229-255) 著者: Leissing, T.M. / Clifton, I.J. / Saward, B.G. / Lu, X. / Hopkinson, R.J. / Schofield, C.J. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  6h9j.cif.gz 6h9j.cif.gz | 169 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb6h9j.ent.gz pdb6h9j.ent.gz | 131.5 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  6h9j.json.gz 6h9j.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/h9/6h9j https://data.pdbj.org/pub/pdb/validation_reports/h9/6h9j ftp://data.pdbj.org/pub/pdb/validation_reports/h9/6h9j ftp://data.pdbj.org/pub/pdb/validation_reports/h9/6h9j | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  1h2kS S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 / タンパク質・ペプチド , 2種, 2分子 AD
| #1: タンパク質 | 分子量: 39354.199 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) / 遺伝子: HIF1AN, FIH1 / プラスミド: pET28 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: HIF1AN, FIH1 / プラスミド: pET28 / 発現宿主:   Escherichia coli BL21(DE3) (大腸菌) Escherichia coli BL21(DE3) (大腸菌)参照: UniProt: Q9NWT6, hypoxia-inducible factor-asparagine dioxygenase, 酸化還元酵素; 電子対供与作用を持つ; 分子酸素を取り込むないしは分子酸素を還元する; 2- ...参照: UniProt: Q9NWT6,  hypoxia-inducible factor-asparagine dioxygenase, hypoxia-inducible factor-asparagine dioxygenase,  酸化還元酵素; 電子対供与作用を持つ; 分子酸素を取り込むないしは分子酸素を還元する; 2-オキソグルタル酸類を片方の電子供与体とする; 酸素分をそれぞれの電子供与体に取り込む 酸化還元酵素; 電子対供与作用を持つ; 分子酸素を取り込むないしは分子酸素を還元する; 2-オキソグルタル酸類を片方の電子供与体とする; 酸素分をそれぞれの電子供与体に取り込む |
|---|---|
| #2: タンパク質・ペプチド | 分子量: 2158.456 Da / 分子数: 1 / 由来タイプ: 合成 / 由来: (合成)   Homo sapiens (ヒト) / 参照: UniProt: Q8NET8 Homo sapiens (ヒト) / 参照: UniProt: Q8NET8 |
-非ポリマー , 5種, 168分子 

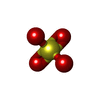






| #3: 化合物 | ChemComp-OGA /  N-Oxalylglycine N-Oxalylglycine | ||||||
|---|---|---|---|---|---|---|---|
| #4: 化合物 |  グリセリン グリセリン#5: 化合物 | ChemComp-SO4 /  硫酸塩 硫酸塩#6: 化合物 | ChemComp-ZN / | #7: 水 | ChemComp-HOH / |  水 水 |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.36 Å3/Da / 溶媒含有率: 63.43 % |
|---|---|
結晶化 | 温度: 293 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 7.5 詳細: 0.1 M Hepes pH = 7.5, 1.8 M ammonium sulfate, 4.5% PEG400 |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  Diamond Diamond  / ビームライン: I03 / 波長: 0.9795 Å / ビームライン: I03 / 波長: 0.9795 Å |
| 検出器 | タイプ: DECTRIS PILATUS 6M-F / 検出器: PIXEL / 日付: 2017年1月23日 |
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 0.9795 Å / 相対比: 1 : 0.9795 Å / 相対比: 1 |
| 反射 | 解像度: 1.83→38.635 Å / Num. obs: 50768 / % possible obs: 100 % / 冗長度: 19.4 % / Biso Wilson estimate: 41.46 Å2 / CC1/2: 0.9984 / Rmerge(I) obs: 0.1054 / Rpim(I) all: 0.025 / Rrim(I) all: 0.108 / Net I/σ(I): 13.74 |
| 反射 シェル | 解像度: 1.83→1.86 Å / 冗長度: 20.1 % / Rmerge(I) obs: 2.854 / Mean I/σ(I) obs: 1.2 / Num. unique obs: 2483 / CC1/2: 0.497 / Rpim(I) all: 0.649 / Rrim(I) all: 2.928 / % possible all: 100 |
-位相決定
位相決定 | 手法:  分子置換 分子置換 |
|---|
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: 1H2K 解像度: 1.83→38.635 Å / SU ML: 0.2 / 交差検証法: THROUGHOUT / σ(F): 1.34 / 位相誤差: 20.45
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso max: 121.4 Å2 / Biso mean: 55.7524 Å2 / Biso min: 31.84 Å2 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: final / 解像度: 1.83→38.635 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | Refine-ID: X-RAY DIFFRACTION / Rfactor Rfree error: 0 / Total num. of bins used: 18 / % reflection obs: 100 %
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 TLSグループ |
|
 ムービー
ムービー コントローラー
コントローラー























 PDBj
PDBj







