+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 5nug | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Motor domains from human cytoplasmic dynein-1 in the phi-particle conformation | |||||||||
 要素 要素 | Cytoplasmic dynein 1 heavy chain 1 | |||||||||
 キーワード キーワード |  MOTOR PROTEIN (モータータンパク質) / MOTOR PROTEIN (モータータンパク質) /  dynein (ダイニン) / dynein (ダイニン) /  motor domain (機関 (機械)) / motor domain (機関 (機械)) /  AAA+ AAA+ | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of intracellular transport / regulation of metaphase plate congression / establishment of spindle localization / positive regulation of spindle assembly /  dynein complex / dynein complex /  P-body assembly / COPI-independent Golgi-to-ER retrograde traffic / minus-end-directed microtubule motor activity / retrograde axonal transport / P-body assembly / COPI-independent Golgi-to-ER retrograde traffic / minus-end-directed microtubule motor activity / retrograde axonal transport /  cytoplasmic dynein complex ...positive regulation of intracellular transport / regulation of metaphase plate congression / establishment of spindle localization / positive regulation of spindle assembly / cytoplasmic dynein complex ...positive regulation of intracellular transport / regulation of metaphase plate congression / establishment of spindle localization / positive regulation of spindle assembly /  dynein complex / dynein complex /  P-body assembly / COPI-independent Golgi-to-ER retrograde traffic / minus-end-directed microtubule motor activity / retrograde axonal transport / P-body assembly / COPI-independent Golgi-to-ER retrograde traffic / minus-end-directed microtubule motor activity / retrograde axonal transport /  cytoplasmic dynein complex / dynein light intermediate chain binding / nuclear migration / dynein intermediate chain binding / cytoplasmic microtubule / Amplification of signal from unattached kinetochores via a MAD2 inhibitory signal / COPI-mediated anterograde transport / cytoplasmic dynein complex / dynein light intermediate chain binding / nuclear migration / dynein intermediate chain binding / cytoplasmic microtubule / Amplification of signal from unattached kinetochores via a MAD2 inhibitory signal / COPI-mediated anterograde transport /  stress granule assembly / cytoplasmic microtubule organization / Mitotic Prometaphase / EML4 and NUDC in mitotic spindle formation / axon cytoplasm / regulation of mitotic spindle organization / Loss of Nlp from mitotic centrosomes / Loss of proteins required for interphase microtubule organization from the centrosome / Recruitment of mitotic centrosome proteins and complexes / Resolution of Sister Chromatid Cohesion / Recruitment of NuMA to mitotic centrosomes / HSP90 chaperone cycle for steroid hormone receptors (SHR) in the presence of ligand / Anchoring of the basal body to the plasma membrane / MHC class II antigen presentation / AURKA Activation by TPX2 / mitotic spindle organization / stress granule assembly / cytoplasmic microtubule organization / Mitotic Prometaphase / EML4 and NUDC in mitotic spindle formation / axon cytoplasm / regulation of mitotic spindle organization / Loss of Nlp from mitotic centrosomes / Loss of proteins required for interphase microtubule organization from the centrosome / Recruitment of mitotic centrosome proteins and complexes / Resolution of Sister Chromatid Cohesion / Recruitment of NuMA to mitotic centrosomes / HSP90 chaperone cycle for steroid hormone receptors (SHR) in the presence of ligand / Anchoring of the basal body to the plasma membrane / MHC class II antigen presentation / AURKA Activation by TPX2 / mitotic spindle organization /  filopodium / RHO GTPases Activate Formins / Aggrephagy / HCMV Early Events / Separation of Sister Chromatids / filopodium / RHO GTPases Activate Formins / Aggrephagy / HCMV Early Events / Separation of Sister Chromatids /  Regulation of PLK1 Activity at G2/M Transition / azurophil granule lumen / positive regulation of cold-induced thermogenesis / Regulation of PLK1 Activity at G2/M Transition / azurophil granule lumen / positive regulation of cold-induced thermogenesis /  細胞皮質 / 細胞皮質 /  微小管 / 微小管 /  細胞分裂 / 細胞分裂 /  中心体 / Neutrophil degranulation / 中心体 / Neutrophil degranulation /  ATP hydrolysis activity / ATP hydrolysis activity /  RNA binding / extracellular exosome / extracellular region / RNA binding / extracellular exosome / extracellular region /  ATP binding / ATP binding /  生体膜 / 生体膜 /  細胞質基質 細胞質基質類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.8 Å クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | Zhang, K. / Foster, H.E. / Carter, A.P. | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2017 ジャーナル: Cell / 年: 2017タイトル: Cryo-EM Reveals How Human Cytoplasmic Dynein Is Auto-inhibited and Activated. 著者: Kai Zhang / Helen E Foster / Arnaud Rondelet / Samuel E Lacey / Nadia Bahi-Buisson / Alexander W Bird / Andrew P Carter /    要旨: Cytoplasmic dynein-1 binds dynactin and cargo adaptor proteins to form a transport machine capable of long-distance processive movement along microtubules. However, it is unclear why dynein-1 moves ...Cytoplasmic dynein-1 binds dynactin and cargo adaptor proteins to form a transport machine capable of long-distance processive movement along microtubules. However, it is unclear why dynein-1 moves poorly on its own or how it is activated by dynactin. Here, we present a cryoelectron microscopy structure of the complete 1.4-megadalton human dynein-1 complex in an inhibited state known as the phi-particle. We reveal the 3D structure of the cargo binding dynein tail and show how self-dimerization of the motor domains locks them in a conformation with low microtubule affinity. Disrupting motor dimerization with structure-based mutagenesis drives dynein-1 into an open form with higher affinity for both microtubules and dynactin. We find the open form is also inhibited for movement and that dynactin relieves this by reorienting the motor domains to interact correctly with microtubules. Our model explains how dynactin binding to the dynein-1 tail directly stimulates its motor activity. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  5nug.cif.gz 5nug.cif.gz | 1.2 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb5nug.ent.gz pdb5nug.ent.gz | 955.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  5nug.json.gz 5nug.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/nu/5nug https://data.pdbj.org/pub/pdb/validation_reports/nu/5nug ftp://data.pdbj.org/pub/pdb/validation_reports/nu/5nug ftp://data.pdbj.org/pub/pdb/validation_reports/nu/5nug | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  3698MC  3703C  3704C  3705C  3706C  3707C  5nvsC  5nvuC  5nw4C M: このデータのモデリングに利用したマップデータ C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 | 
|
| 2 | 
|
- 要素
要素
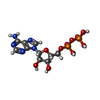
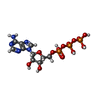
| #1: タンパク質 | 分子量: 533083.250 Da / 分子数: 2 / 由来タイプ: 組換発現 / 由来: (組換発現)   Homo sapiens (ヒト) Homo sapiens (ヒト)遺伝子: DYNC1H1, DHC1, DNCH1, DNCL, DNECL, DYHC, KIAA0325 発現宿主:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ)参照: UniProt: Q14204 #2: 化合物 | ChemComp-ADP /  アデノシン二リン酸 アデノシン二リン酸#3: 化合物 |  アデノシン三リン酸 アデノシン三リン酸#4: 化合物 | |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: Motor domains from human cytoplasmic dynein-1 in the phi-particle conformation タイプ: COMPLEX / Entity ID: #1 / 由来: RECOMBINANT |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 由来(組換発現) | 生物種:   Spodoptera frugiperda (ツマジロクサヨトウ) Spodoptera frugiperda (ツマジロクサヨトウ) |
| 緩衝液 | pH: 7.4 詳細: 20mM HEPES pH7.4, 150 mM KCl, 1mM MgCl2, 5 mM DTT, 0.1 mM ATP |
| 試料 | 濃度: 0.15 mg/ml / 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES詳細: vitrification was performed immediately after gel filtration. |
| 試料支持 | グリッドの材料: GOLD / グリッドのサイズ: 300 divisions/in. / グリッドのタイプ: Quantifoil R1.2/1.3 |
急速凍結 | 装置: FEI VITROBOT MARK IV / 凍結剤: ETHANE / 湿度: 100 % / 凍結前の試料温度: 277 K |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 倍率(公称値): 59000 X / 倍率(補正後): 106061 X / 最大 デフォーカス(公称値): 5000 nm / 最小 デフォーカス(公称値): 1500 nm / Calibrated defocus min: 1500 nm / 最大 デフォーカス(補正後): 5000 nm / Cs Bright-field microscopy / 倍率(公称値): 59000 X / 倍率(補正後): 106061 X / 最大 デフォーカス(公称値): 5000 nm / 最小 デフォーカス(公称値): 1500 nm / Calibrated defocus min: 1500 nm / 最大 デフォーカス(補正後): 5000 nm / Cs : 2.7 mm / C2レンズ絞り径: 70 µm / アライメント法: COMA FREE : 2.7 mm / C2レンズ絞り径: 70 µm / アライメント法: COMA FREE |
| 試料ホルダ | 凍結剤: NITROGEN 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER 最高温度: 100 K / 最低温度: 90 K |
| 撮影 | 平均露光時間: 2 sec. / 電子線照射量: 1.6 e/Å2 / 検出モード: INTEGRATING フィルム・検出器のモデル: FEI FALCON II (4k x 4k) 撮影したグリッド数: 6746 / 実像数: 312198 詳細: Images were also collected using K2 summit detetor in counting mode |
| 画像スキャン | サンプリングサイズ: 14 µm / 横: 4096 / 縦: 4096 / 動画フレーム数/画像: 34 / 利用したフレーム数/画像: 3-34 |
- 解析
解析
| ソフトウェア | 名称: REFMAC / バージョン: 5.8.0166 / 分類: 精密化 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EMソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 画像処理 | 詳細: Negative stain dataset was processed as | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 粒子像の選択 | 選択した粒子像数: 1 詳細: Negative stain dataset was processed to generate templates. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 点対称性 : C2 (2回回転対称 : C2 (2回回転対称 ) ) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
3次元再構成 | 解像度: 3.8 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 233227 / アルゴリズム: BACK PROJECTION / クラス平均像の数: 1 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | B value: 145 / プロトコル: AB INITIO MODEL / 空間: RECIPROCAL | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | 解像度: 3.8→232.32 Å / Cor.coef. Fo:Fc: 0.865 / SU B: 59.043 / SU ML: 0.777 / ESU R: 0.879 立体化学のターゲット値: MAXIMUM LIKELIHOOD WITH PHASES 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 溶媒モデル: PARAMETERS FOR MASK CACLULATION | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 145.21 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: 1 / 合計: 46232 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー









 PDBj
PDBj