+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1aq2 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | PHOSPHOENOLPYRUVATE CARBOXYKINASE | |||||||||
 要素 要素 | PHOSPHOENOLPYRUVATE CARBOXYKINASE ホスホエノールピルビン酸カルボキシキナーゼ ホスホエノールピルビン酸カルボキシキナーゼ | |||||||||
 キーワード キーワード |  KINASE (キナーゼ) / TRANSPHOSPHORYLATING / KINASE (キナーゼ) / TRANSPHOSPHORYLATING /  P-LOOP (Walkerモチーフ) / PROTEIN-ATP COMPLEX / NUCLEOTIDE-TRIPHOSPHATE HYDROLASE / P-LOOP (Walkerモチーフ) / PROTEIN-ATP COMPLEX / NUCLEOTIDE-TRIPHOSPHATE HYDROLASE /  LYASE (リアーゼ) / LYASE (リアーゼ) /  GLUCONEOGENESIS (糖新生) GLUCONEOGENESIS (糖新生) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 phosphoenolpyruvate carboxykinase (ATP) / phosphoenolpyruvate carboxykinase (ATP) /  phosphoenolpyruvate carboxykinase (ATP) activity / phosphoenolpyruvate carboxykinase (ATP) activity /  糖新生 / 糖新生 /  calcium ion binding / magnesium ion binding / calcium ion binding / magnesium ion binding /  ATP binding / ATP binding /  細胞質基質 細胞質基質類似検索 - 分子機能 | |||||||||
| 生物種 |   Escherichia coli (大腸菌) Escherichia coli (大腸菌) | |||||||||
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 1.9 Å 分子置換 / 解像度: 1.9 Å | |||||||||
 データ登録者 データ登録者 | Tari, L.W. / Matte, A. / Goldie, H. / Delbaere, L.T.J. | |||||||||
 引用 引用 |  ジャーナル: Nat.Struct.Biol. / 年: 1997 ジャーナル: Nat.Struct.Biol. / 年: 1997タイトル: Mg(2+)-Mn2+ clusters in enzyme-catalyzed phosphoryl-transfer reactions. 著者: Tari, L.W. / Matte, A. / Goldie, H. / Delbaere, L.T. #1:  ジャーナル: J.Mol.Biol. / 年: 1996 ジャーナル: J.Mol.Biol. / 年: 1996タイトル: Crystal Structure of Escherichia Coli Phosphoenolpyruvate Carboxykinase: A New Structural Family with the P-Loop Nucleoside Triphosphate Hydrolase Fold 著者: Matte, A. / Goldie, H. / Sweet, R.M. / Delbaere, L.T. #2:  ジャーナル: Nat.Struct.Biol. / 年: 1996 ジャーナル: Nat.Struct.Biol. / 年: 1996タイトル: Snapshot of an Enzyme Reaction Intermediate in the Structure of the ATP-Mg2+-Oxalate Ternary Complex of Escherichia Coli Pep Carboxykinase 著者: Tari, L.W. / Matte, A. / Pugazhenthi, U. / Goldie, H. / Delbaere, L.T. #3:  ジャーナル: J.Mol.Biol. / 年: 1991 ジャーナル: J.Mol.Biol. / 年: 1991タイトル: Crystallization of the Calcium-Activated Phosphoenolpyruvate Carboxykinase from Escherichia Coli K12 著者: Delbaere, L.T. / Vandonselaar, M. / Glaeske, D. / Jabs, C. / Goldie, H. #4:  ジャーナル: J.Bacteriol. / 年: 1990 ジャーナル: J.Bacteriol. / 年: 1990タイトル: Sequence of the Pcka Gene of Escherichia Coli K-12: Relevance to Genetic and Allosteric Regulation and Homology of E. Coli Phosphoenolpyruvate Carboxykinase with the Enzymes from ...タイトル: Sequence of the Pcka Gene of Escherichia Coli K-12: Relevance to Genetic and Allosteric Regulation and Homology of E. Coli Phosphoenolpyruvate Carboxykinase with the Enzymes from Trypanosoma Brucei and Saccharomyces Cerevisiae 著者: Medina, V. / Pontarollo, R. / Glaeske, D. / Tabel, H. / Goldie, H. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1aq2.cif.gz 1aq2.cif.gz | 123.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1aq2.ent.gz pdb1aq2.ent.gz | 93.1 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1aq2.json.gz 1aq2.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/aq/1aq2 https://data.pdbj.org/pub/pdb/validation_reports/aq/1aq2 ftp://data.pdbj.org/pub/pdb/validation_reports/aq/1aq2 ftp://data.pdbj.org/pub/pdb/validation_reports/aq/1aq2 | HTTPS FTP |
|---|
-関連構造データ
| 関連構造データ |  1aylS S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 1種, 1分子 A
| #1: タンパク質 |  ホスホエノールピルビン酸カルボキシキナーゼ / ATP-OXALOACETATE CARBOXY-LYASE (ATP) ホスホエノールピルビン酸カルボキシキナーゼ / ATP-OXALOACETATE CARBOXY-LYASE (ATP)分子量: 59709.176 Da / 分子数: 1 / 由来タイプ: 組換発現 詳細: ORDERED MAGNESIUM AND MANGANESE IONS OBSERVED BOUND TO ATP 由来: (組換発現)   Escherichia coli (大腸菌) / 株: K12 Escherichia coli (大腸菌) / 株: K12解説: NATIVE PROMOTER OF E. COLI PCKA GENE, HIGHLY AEROBIC GROWTH TO EARLY STATIONARY PHASE IN THE ABSENCE OF CARBOHYDRATES 細胞内の位置: CYTOPLASM  細胞質 / 遺伝子 (発現宿主): PCKA / 発現宿主: 細胞質 / 遺伝子 (発現宿主): PCKA / 発現宿主:   Escherichia coli (大腸菌) Escherichia coli (大腸菌)参照: UniProt: P22259,  phosphoenolpyruvate carboxykinase (ATP) phosphoenolpyruvate carboxykinase (ATP) |
|---|
-非ポリマー , 5種, 236分子 

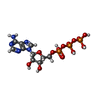






| #2: 化合物 | ChemComp-MN / |
|---|---|
| #3: 化合物 | ChemComp-MG / |
| #4: 化合物 | ChemComp-ATP /  アデノシン三リン酸 アデノシン三リン酸 |
| #5: 化合物 | ChemComp-PYR /  ピルビン酸 ピルビン酸 |
| #6: 水 | ChemComp-HOH /  水 水 |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.38 Å3/Da / 溶媒含有率: 50 % |
|---|---|
結晶化 | pH: 4.4 / 詳細: pH 4.4 |
-データ収集
| 回折 | 平均測定温度: 300 K |
|---|---|
| 放射光源 | タイプ: ENRAF-NONIUS / 波長: 1.5418 |
| 検出器 | タイプ: ENRAF-NONIUS FAST / 検出器: DIFFRACTOMETER / 日付: 1996年10月15日 |
| 放射 | モノクロメーター: GRAPHITE(002) / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長 : 1.5418 Å / 相対比: 1 : 1.5418 Å / 相対比: 1 |
| 反射 | 解像度: 1.9→60 Å / Num. obs: 39324 / % possible obs: 90 % / Observed criterion σ(I): 1 / 冗長度: 2.6 % / Rsym value: 0.118 / Net I/σ(I): 4.3 |
| 反射 シェル | 解像度: 1.9→3 Å / 冗長度: 2.4 % / Mean I/σ(I) obs: 2.1 / Rsym value: 0.34 / % possible all: 85 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法 : :  分子置換 分子置換開始モデル: 1AYL 解像度: 1.9→6 Å / 交差検証法: FREE-R / σ(F): 2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze | Luzzati coordinate error obs: 0.2 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.9→6 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj





