+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: EMDB / ID: EMD-8698 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | EBOV GPdMuc:ADI-16061 | |||||||||
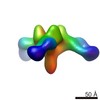
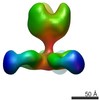
 マップデータ マップデータ | Complex of recombinant Ebola virus glycoprotein and recombinant human IgG1 Fab ADI-16061 from survivor of infection. | |||||||||
 試料 試料 |
| |||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  ネガティブ染色法 / 解像度: 25.3 Å ネガティブ染色法 / 解像度: 25.3 Å | |||||||||
 データ登録者 データ登録者 | Murin CD / Ward AB / Turner HL | |||||||||
 引用 引用 |  ジャーナル: Cell / 年: 2017 ジャーナル: Cell / 年: 2017タイトル: Antibodies from a Human Survivor Define Sites of Vulnerability for Broad Protection against Ebolaviruses. 著者: Anna Z Wec / Andrew S Herbert / Charles D Murin / Elisabeth K Nyakatura / Dafna M Abelson / J Maximilian Fels / Shihua He / Rebekah M James / Marc-Antoine de La Vega / Wenjun Zhu / Russell R ...著者: Anna Z Wec / Andrew S Herbert / Charles D Murin / Elisabeth K Nyakatura / Dafna M Abelson / J Maximilian Fels / Shihua He / Rebekah M James / Marc-Antoine de La Vega / Wenjun Zhu / Russell R Bakken / Eileen Goodwin / Hannah L Turner / Rohit K Jangra / Larry Zeitlin / Xiangguo Qiu / Jonathan R Lai / Laura M Walker / Andrew B Ward / John M Dye / Kartik Chandran / Zachary A Bornholdt /   要旨: Experimental monoclonal antibody (mAb) therapies have shown promise for treatment of lethal Ebola virus (EBOV) infections, but their species-specific recognition of the viral glycoprotein (GP) has ...Experimental monoclonal antibody (mAb) therapies have shown promise for treatment of lethal Ebola virus (EBOV) infections, but their species-specific recognition of the viral glycoprotein (GP) has limited their use against other divergent ebolaviruses associated with human disease. Here, we mined the human immune response to natural EBOV infection and identified mAbs with exceptionally potent pan-ebolavirus neutralizing activity and protective efficacy against three virulent ebolaviruses. These mAbs recognize an inter-protomer epitope in the GP fusion loop, a critical and conserved element of the viral membrane fusion machinery, and neutralize viral entry by targeting a proteolytically primed, fusion-competent GP intermediate (GP) generated in host cell endosomes. Only a few somatic hypermutations are required for broad antiviral activity, and germline-approximating variants display enhanced GP recognition, suggesting that such antibodies could be elicited more efficiently with suitably optimized GP immunogens. Our findings inform the development of both broadly effective immunotherapeutics and vaccines against filoviruses. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | EMマップ:  SurfView SurfView Molmil Molmil Jmol/JSmol Jmol/JSmol |
| 添付画像 |
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_8698.map.gz emd_8698.map.gz | 758.8 KB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-8698-v30.xml emd-8698-v30.xml emd-8698.xml emd-8698.xml | 14 KB 14 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_8698.png emd_8698.png | 30.8 KB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-8698 http://ftp.pdbj.org/pub/emdb/structures/EMD-8698 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8698 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-8698 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_8698.map.gz / 形式: CCP4 / 大きさ: 1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_8698.map.gz / 形式: CCP4 / 大きさ: 1 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Complex of recombinant Ebola virus glycoprotein and recombinant human IgG1 Fab ADI-16061 from survivor of infection. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 4.1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
CCP4マップ ヘッダ情報:
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
-添付データ
- 試料の構成要素
試料の構成要素
-全体 : EBOVGPdMuc:ADI-16061
| 全体 | 名称: EBOVGPdMuc:ADI-16061 |
|---|---|
| 要素 |
|
-超分子 #1: EBOVGPdMuc:ADI-16061
| 超分子 | 名称: EBOVGPdMuc:ADI-16061 / タイプ: complex / ID: 1 / 親要素: 0 詳細: Complex of recombinant Ebola virus glycoprotein and recombinant human IgG1 Fab from survivor of infection. |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Homo sapiens (ヒト) / 組換細胞: HEK 293F / 組換プラスミド: pPPI4 Homo sapiens (ヒト) / 組換細胞: HEK 293F / 組換プラスミド: pPPI4 |
-超分子 #2: EBOV GPdMuc
| 超分子 | 名称: EBOV GPdMuc / タイプ: complex / ID: 2 / 親要素: 1 詳細: Recombinant mucin-deleted Ebola virus glycoprotein expressed in mammalian cells, 293F. |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Homo sapiens (ヒト) / 組換細胞: HEK 293F / 組換プラスミド: pPPI4 Homo sapiens (ヒト) / 組換細胞: HEK 293F / 組換プラスミド: pPPI4 |
-超分子 #3: ADI-16061 Fab
| 超分子 | 名称: ADI-16061 Fab / タイプ: complex / ID: 3 / 親要素: 1 詳細: Fab fragment generated by proteolytic cleavage of IgG antibody, expressed in plant system. |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 組換発現 | 生物種:   Nicotiana benthamiana (ベンサミアナタバコ) Nicotiana benthamiana (ベンサミアナタバコ) |
-実験情報
-構造解析
| 手法 |  ネガティブ染色法 ネガティブ染色法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.03 mg/mL | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 構成要素:
詳細: Solutions were made from a 10X stock and filtered sterilized. | |||||||||
| 染色 | タイプ: NEGATIVE / 材質: Uranyl Formate 詳細: Negatively stained EM specimens were prepared using a side-blotting technique and uranyl-formate stain. | |||||||||
| グリッド | モデル: Protochip / 材質: COPPER / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: PLASMA CLEANING / 前処理 - 雰囲気: OTHER | |||||||||
| 詳細 | Monodisperse sample deposited on negative stain grid. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TECNAI SPIRIT |
|---|---|
| 電子線 | 加速電圧: 120 kV / 電子線源: LAB6 |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最小 デフォーカス(公称値): 1.0 µm Bright-field microscopy / 最小 デフォーカス(公称値): 1.0 µm |
| 試料ステージ | 試料ホルダーモデル: SIDE ENTRY, EUCENTRIC |
| 撮影 | フィルム・検出器のモデル: TVIPS TEMCAM-F416 (4k x 4k) 撮影したグリッド数: 1 / 実像数: 53 / 平均電子線量: 2.0 e/Å2 |
| 実験機器 |  モデル: Tecnai Spirit / 画像提供: FEI Company |
- 画像解析
画像解析
| 粒子像選択 | 選択した数: 11650 |
|---|---|
| 初期モデル | モデルのタイプ: OTHER 詳細: Common lines start model obtained from negative stain class averages of the data. |
| 初期 角度割当 | タイプ: PROJECTION MATCHING / ソフトウェア - 名称: EMAN (ver. 2.2) |
| 最終 3次元分類 | クラス数: 82 / 平均メンバー数/クラス: 50 / ソフトウェア - 名称: IMAGIC / 詳細: Iterative MSA/MRA |
| 最終 角度割当 | タイプ: PROJECTION MATCHING / ソフトウェア - 名称: EMAN (ver. 2.2) |
| 最終 再構成 | 想定した対称性 - 点群: C3 (3回回転対称 ) / 解像度のタイプ: BY AUTHOR / 解像度: 25.3 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: EMAN (ver. 2.2) / 使用した粒子像数: 6677 ) / 解像度のタイプ: BY AUTHOR / 解像度: 25.3 Å / 解像度の算出法: FSC 0.5 CUT-OFF / ソフトウェア - 名称: EMAN (ver. 2.2) / 使用した粒子像数: 6677 |
 ムービー
ムービー コントローラー
コントローラー