+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM Structure of the Helicobacter pylori cagYdAP PR | ||||||||||||
 マップデータ マップデータ | |||||||||||||
 試料 試料 |
| ||||||||||||
 キーワード キーワード |  T4SS / T4SS /  protein translocation / protein translocation /  MEMBRANE PROTEIN (膜タンパク質) MEMBRANE PROTEIN (膜タンパク質) | ||||||||||||
| 生物種 |   Helicobacter pylori 26695 (ピロリ菌) Helicobacter pylori 26695 (ピロリ菌) | ||||||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 6.6 Å クライオ電子顕微鏡法 / 解像度: 6.6 Å | ||||||||||||
 データ登録者 データ登録者 | Roberts JR | ||||||||||||
| 資金援助 |  米国, 3件 米国, 3件
| ||||||||||||
 引用 引用 |  ジャーナル: Life Science Alliance / 年: 2024 ジャーナル: Life Science Alliance / 年: 2024タイトル: Subdomains of the H. pylori Cag T4SS outer membrane core complex exhibit structural independence 著者: Roberts JR / Tran SC / Frick-Cheng AF / Bryant KN / Okoye CD / Cover TL / Ohi MD | ||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_42392.map.gz emd_42392.map.gz | 978.8 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-42392-v30.xml emd-42392-v30.xml emd-42392.xml emd-42392.xml | 16.5 KB 16.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_42392_fsc.xml emd_42392_fsc.xml | 21.6 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_42392.png emd_42392.png | 69.1 KB | ||
| Filedesc metadata |  emd-42392.cif.gz emd-42392.cif.gz | 5.6 KB | ||
| その他 |  emd_42392_half_map_1.map.gz emd_42392_half_map_1.map.gz emd_42392_half_map_2.map.gz emd_42392_half_map_2.map.gz | 964.5 MB 964.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-42392 http://ftp.pdbj.org/pub/emdb/structures/EMD-42392 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42392 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-42392 | HTTPS FTP |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_42392.map.gz / 形式: CCP4 / 大きさ: 1 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_42392.map.gz / 形式: CCP4 / 大きさ: 1 GB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_42392_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
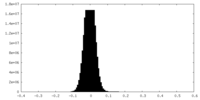
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_42392_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
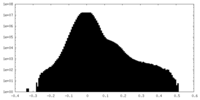
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Helicobacter pylori CagYdAP PR
| 全体 | 名称: Helicobacter pylori CagYdAP PR |
|---|---|
| 要素 |
|
-超分子 #1: Helicobacter pylori CagYdAP PR
| 超分子 | 名称: Helicobacter pylori CagYdAP PR / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Helicobacter pylori 26695 (ピロリ菌) / 株: 26695 / 細胞中の位置: membrane Helicobacter pylori 26695 (ピロリ菌) / 株: 26695 / 細胞中の位置: membrane |
-分子 #1: Cag pathogenicity island protein (Cag8)
| 分子 | 名称: Cag pathogenicity island protein (Cag8) / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Helicobacter pylori 26695 (ピロリ菌) Helicobacter pylori 26695 (ピロリ菌) |
| 配列 | 文字列: MGQAFFKKIV GCFCLGYLF L SSAIEAAA LD IKNFNRG RVK VVNKKI AYLG DEKPI TIWTS LDNV TVIQLE KDE TISYITT GF NKGWSIVP N SNHIFIQPK SVKSNLMFEK EAVNFALMT R DYQEFLKT KK LIVDAPD PKE LEEQKK ALEK EKEAK ...文字列: MGQAFFKKIV GCFCLGYLF L SSAIEAAA LD IKNFNRG RVK VVNKKI AYLG DEKPI TIWTS LDNV TVIQLE KDE TISYITT GF NKGWSIVP N SNHIFIQPK SVKSNLMFEK EAVNFALMT R DYQEFLKT KK LIVDAPD PKE LEEQKK ALEK EKEAK EQAQK AQKD KREKRK EER AKNRANL EN LTNAMSNP Q NLSNNKNLS EFIKQQRENE LDQMERLED M QEQAQANA LK QIEELNK KQA EETIKQ RAKD KINIK TDKPQ KSPE DNSIEL SPS DSAWRTN LV VRTNKALY Q FILRIAQKD NFASAYLTVK LEYPQRHEV S SVIEELKK RE EAKRQKE LIK QENLNT TAYI NRVMM ASNEQ IINK EKIREE KQK IILDQAK AL ETQYVHNA L KRNPVPRNY NYYQAPEKRS KHIMPSEIF D DGTFTYFG FK NITLQPA IFV VQPDGK LSMT DAAID PNMTN SGLR WYRVNE IAE KFKLIKD KA LVTVINKG Y GKNPLTKNY NIKNYGELER VIKKLPEVR D K |
-分子 #2: Cag pathogenicity island protein (Cag7)
| 分子 | 名称: Cag pathogenicity island protein (Cag7) / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Helicobacter pylori 26695 (ピロリ菌) Helicobacter pylori 26695 (ピロリ菌) |
| 配列 | 文字列: MNEENDKLET SKKAQQDSPQ DLSNEEATEA NHFENLLKES KESSDHHLDN PTETQTHFDG DKSEETQTQM DSEGNETSES SNGSLADKLF KKARKLVDNK KPFTQQKNLD EETQELNEED DQENNEYQEE TQTDLIDDET SKKTQQHSPQ DLSNEEATEA NHFENLLKES ...文字列: MNEENDKLET SKKAQQDSPQ DLSNEEATEA NHFENLLKES KESSDHHLDN PTETQTHFDG DKSEETQTQM DSEGNETSES SNGSLADKLF KKARKLVDNK KPFTQQKNLD EETQELNEED DQENNEYQEE TQTDLIDDET SKKTQQHSPQ DLSNEEATEA NHFENLLKES KESSDHHLDN PTETQTNFDG DKSEETQTQM DSEGNETSES SNGSLADKLF KKARKLVDNK KPFTQQKNLD EETQELNEED DQENNEYQEE TQTDLIDDET SKKTQQHSPQ DLSNEEATEA NHFENLLKES KESSDHHLDN PTETQTNFDG DKSEEITDDS NDQEIIKGSK KKYIIGGIVV AVLIVIILFS RSIFHYFMPL EDKSSRFSKD RNLYVNDEIQ IRQEYNRLLK ERNEKGNMID KNLFFNDDPN RTLYNYLNIA EIEDKNPLRA FYECISNGGN YEECLKLIKD KKLQDQMKKT LEAYNDCIKN AKTEEERIKC LDLIKDENLK KSLLNQQKVQ VALDCLKNAK TDEERNECLK LINDPEIREK FRKELELQKE LQEYKDCIKN AKTEAEKNKC LKGLSKEAIE RLKQQALDCL KNAKTDEERN ECLKNIPQDL QKELLADMSV KAYKDCVSKA RNEKEKQECE KLLTPEARKK LEQQVLDCLK NAKTDEERKK CLKDLPKDLQ SDILAKESLK AYKDCVSQAK TEAEKKECEK LLTPEAKKLL EEEAKESVKA YLDCVSQAKT EAEKKECEKL LTPEAKKKLE EAKKSVKAYL DCVSRARNEK EKKECEKLLT PEAKKLLEQQ ALDCLKNAKT DKERKKCLKD LPKDLQKKVL AKESVKAYLD CVSQAKTEAE KKECEKLLTP EARKLLEEAK KSVKAYLDCV SQAKTEAEKK ECEKLLTPEA RKLLEEXAKE SVKAYLDCVS QAKNEAEKKE CEKLLTLESK KKLEEAKKSV KAYLDCVSQA KTEAEKKECE KLLTPEAKKL LEQQALDCLK NAKTEADKKR CVKDLPKDLQ KKVLAKESLK AYKDCVSKAR NEKEKKECEK LLTPEAKKLL EEAKKSVKAY LDCVSQAKTE AEKKECEKLL TPEARKLLEE AKESVKAYKD CVSKARNEKE KKECEKLLTP EAKKLLEQQV LDCLKNAKTE ADKKRCVKDL PKDLQKKVLA KESVKAYLDC VSRARNEKEK KECEKLLTPE AKKLLEEAKE SLKAYKDCLS QARNEEERRA CEKLLTPEAR KLLEQEVKKS IKAYLDCVSR ARNEKEKKEC EKLLTPEARK FLAKQVLNCL EKAGNEEERK ACLKNLPKDL QENILAKESL KAYKDCLSQA RNEEERRACE KLLTPEARKL LEQEVKKSVK AYLDCVSRAR NEKEKKECEK LLTPEARKFL AKELQQKDKA IKDCLKNADP NDRAAIMKCL DGLSDEEKLK YLQEAREKAV ADCLAMAKTD EEKRKCQNLY SDLIQEIQNK RTQNKQNQLS KTERLHQASE CLDNLDDPTD QEAIEQCLEG LSDSERALIL GIKRQADEVD LIYSDLRNRK TFDNMAAKGY PLLPMDFKNG GDIATINATN VDADKIASDN PIYASIEPDI AKQYETEKTI KDKNLEAKLA KALGGNKKDD DKEKSKKSTA EAKAENNKID KDVAETAKNI SEIALKNKKE KSGEFVDENG NPIDDKKKAE KQDETSPVKQ AFIGKSDPTF VLAQYTPIEI TLTSKVDATL TGIVSGVVAK DVWNMNGTMI LLDKGTKVYG NYQSVKGGTP IMTRLMIVFT KAITPDGVII PLANAQAAGM LGEAGVDGYV NNMNIPPSFY KNEGDSIKIL TMDDIDFSGV YDVKITNKSV VDEIIKQSTK TLSREHEEIT TSPKGGN |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.4 mg/mL |
|---|---|
| 緩衝液 | pH: 7 |
| 凍結 | 凍結剤: NITROGEN |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm Bright-field microscopy / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 60.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




 Z
Z Y
Y X
X