+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 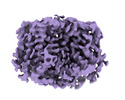 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | E. coli MraY mutant-T23P | |||||||||
 マップデータ マップデータ | sharpened map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  peptidoglycan (ペプチドグリカン) / peptidoglycan (ペプチドグリカン) /  phosphotransferase / phosphotransferase /  MEMBRANE PROTEIN (膜タンパク質) MEMBRANE PROTEIN (膜タンパク質) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 phospho-N-acetylmuramoyl-pentapeptide-transferase / UDP-N-acetylmuramoyl-L-alanyl-D-glutamyl-meso-2,6-diaminopimelyl-D-alanyl-D-alanine:undecaprenyl-phosphate transferase activity / phospho-N-acetylmuramoyl-pentapeptide-transferase / UDP-N-acetylmuramoyl-L-alanyl-D-glutamyl-meso-2,6-diaminopimelyl-D-alanyl-D-alanine:undecaprenyl-phosphate transferase activity /  phospho-N-acetylmuramoyl-pentapeptide-transferase activity / cell wall macromolecule biosynthetic process / peptidoglycan biosynthetic process / cell wall organization / regulation of cell shape / phospho-N-acetylmuramoyl-pentapeptide-transferase activity / cell wall macromolecule biosynthetic process / peptidoglycan biosynthetic process / cell wall organization / regulation of cell shape /  細胞周期 / 細胞周期 /  細胞分裂 / 細胞分裂 /  生体膜 ... 生体膜 ... phospho-N-acetylmuramoyl-pentapeptide-transferase / UDP-N-acetylmuramoyl-L-alanyl-D-glutamyl-meso-2,6-diaminopimelyl-D-alanyl-D-alanine:undecaprenyl-phosphate transferase activity / phospho-N-acetylmuramoyl-pentapeptide-transferase / UDP-N-acetylmuramoyl-L-alanyl-D-glutamyl-meso-2,6-diaminopimelyl-D-alanyl-D-alanine:undecaprenyl-phosphate transferase activity /  phospho-N-acetylmuramoyl-pentapeptide-transferase activity / cell wall macromolecule biosynthetic process / peptidoglycan biosynthetic process / cell wall organization / regulation of cell shape / phospho-N-acetylmuramoyl-pentapeptide-transferase activity / cell wall macromolecule biosynthetic process / peptidoglycan biosynthetic process / cell wall organization / regulation of cell shape /  細胞周期 / 細胞周期 /  細胞分裂 / 細胞分裂 /  生体膜 / 生体膜 /  metal ion binding / metal ion binding /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Escherichia coli K-12 (大腸菌) Escherichia coli K-12 (大腸菌) | |||||||||
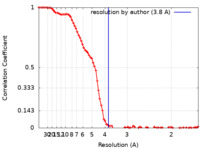
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.8 Å クライオ電子顕微鏡法 / 解像度: 3.8 Å | |||||||||
 データ登録者 データ登録者 | Orta AK / Li YE / Clemons WM | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Microbiol / 年: 2024 ジャーナル: Nat Microbiol / 年: 2024タイトル: Synthesis of lipid-linked precursors of the bacterial cell wall is governed by a feedback control mechanism in Pseudomonas aeruginosa. 著者: Lindsey S Marmont / Anna K Orta / Becca W A Baileeves / David Sychantha / Ana Fernández-Galliano / Yancheng E Li / Neil G Greene / Robin A Corey / Phillip J Stansfeld / William M Clemons / ...著者: Lindsey S Marmont / Anna K Orta / Becca W A Baileeves / David Sychantha / Ana Fernández-Galliano / Yancheng E Li / Neil G Greene / Robin A Corey / Phillip J Stansfeld / William M Clemons / Thomas G Bernhardt /    要旨: Many bacterial surface glycans such as the peptidoglycan (PG) cell wall are built from monomeric units linked to a polyprenyl lipid carrier. How this limiting carrier is distributed among competing ...Many bacterial surface glycans such as the peptidoglycan (PG) cell wall are built from monomeric units linked to a polyprenyl lipid carrier. How this limiting carrier is distributed among competing pathways has remained unclear. Here we describe the isolation of hyperactive variants of Pseudomonas aeruginosa MraY, the enzyme that forms the first lipid-linked PG precursor. These variants result in the elevated production of the final PG precursor lipid II in cells and are hyperactive in vitro. The activated MraY variants have substitutions that map to a cavity on the extracellular side of the dimer interface, far from the active site. Our structural and molecular dynamics results suggest that this cavity is a binding site for externalized lipid II. Overall, our results support a model in which excess externalized lipid II allosterically inhibits MraY, providing a feedback mechanism that prevents the sequestration of lipid carrier in the PG biogenesis pathway. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_41373.map.gz emd_41373.map.gz | 48.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-41373-v30.xml emd-41373-v30.xml emd-41373.xml emd-41373.xml | 19.2 KB 19.2 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_41373_fsc.xml emd_41373_fsc.xml | 11.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_41373.png emd_41373.png | 103.2 KB | ||
| マスクデータ |  emd_41373_msk_1.map emd_41373_msk_1.map | 52.7 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-41373.cif.gz emd-41373.cif.gz | 6.1 KB | ||
| その他 |  emd_41373_additional_1.map.gz emd_41373_additional_1.map.gz emd_41373_half_map_1.map.gz emd_41373_half_map_1.map.gz emd_41373_half_map_2.map.gz emd_41373_half_map_2.map.gz | 49.6 MB 49 MB 49 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-41373 http://ftp.pdbj.org/pub/emdb/structures/EMD-41373 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41373 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-41373 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8tluMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_41373.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_41373.map.gz / 形式: CCP4 / 大きさ: 52.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sharpened map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 0.832 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_41373_msk_1.map emd_41373_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: unsharpened map
| ファイル | emd_41373_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpened map | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_41373_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_41373_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : E. coli MraY T23P dimer
| 全体 | 名称: E. coli MraY T23P dimer |
|---|---|
| 要素 |
|
-超分子 #1: E. coli MraY T23P dimer
| 超分子 | 名称: E. coli MraY T23P dimer / タイプ: organelle_or_cellular_component / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli K-12 (大腸菌) Escherichia coli K-12 (大腸菌) |
| 分子量 | 理論値: 39.875 KDa |
-分子 #1: Phospho-N-acetylmuramoyl-pentapeptide-transferase
| 分子 | 名称: Phospho-N-acetylmuramoyl-pentapeptide-transferase / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli K-12 (大腸菌) Escherichia coli K-12 (大腸菌) |
| 分子量 | 理論値: 39.905551 KDa |
| 組換発現 | 生物種:   Escherichia coli K-12 (大腸菌) Escherichia coli K-12 (大腸菌) |
| 配列 | 文字列: MLVWLAEHLV KYYSGFNVFS YLPFRAIVSL LTALFISLWM GPRMIAHLQK LSFGQVVRND GPESHFSKRG TPTMGGIMIL TAIVISVLL WAYPSNPYVW CVLVVLVGYG VIGFVDDYRK VVRKDTKGLI ARWKYFWMSV IALGVAFALY LAGKDTPATQ L VVPFFKDV ...文字列: MLVWLAEHLV KYYSGFNVFS YLPFRAIVSL LTALFISLWM GPRMIAHLQK LSFGQVVRND GPESHFSKRG TPTMGGIMIL TAIVISVLL WAYPSNPYVW CVLVVLVGYG VIGFVDDYRK VVRKDTKGLI ARWKYFWMSV IALGVAFALY LAGKDTPATQ L VVPFFKDV MPQLGLFYIL LAYFVIVGTG NAVNLTDGLD GLAIMPTVFV AGGFALVAWA TGNMNFASYL HIPYLRHAGE LV IVCTAIV GAGLGFLWFN TYPAQVFMGD VGSLALGGAL GIIAVLLRQE FLLVIMGGVF VVETLSVILQ VGSFKLRGQR IFR MAPIHH HYELKGWPEP RVIVRFWIIS LMLVLIGLAT LKVR UniProtKB:  Phospho-N-acetylmuramoyl-pentapeptide-transferase Phospho-N-acetylmuramoyl-pentapeptide-transferase |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 5 mg/mL |
|---|---|
| 緩衝液 | pH: 7.5 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 60 sec. |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277.15 K / 装置: FEI VITROBOT MARK IV |
| 詳細 | MraY was pulled down using the YES complex. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: OTHER / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 105000 Bright-field microscopy / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 105000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 撮影したグリッド数: 1 / 平均電子線量: 60.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




 Z
Z Y
Y X
X