+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Cryo-EM structure of cyanobacteria phosphoketolase | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | Carbon metabolism / TPP-dependent enzyme /  CYTOSOLIC PROTEIN (細胞質基質) CYTOSOLIC PROTEIN (細胞質基質) | |||||||||
| 機能・相同性 |  : :  機能・相同性情報 機能・相同性情報 | |||||||||
| 生物種 |   Synechococcus (バクテリア) / Synechococcus (バクテリア) /   Synechococcus elongatus (strain ATCC 33912 / PCC 7942 / FACHB-805) (バクテリア) Synechococcus elongatus (strain ATCC 33912 / PCC 7942 / FACHB-805) (バクテリア) | |||||||||
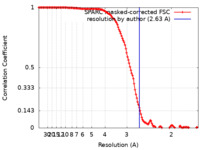
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.63 Å クライオ電子顕微鏡法 / 解像度: 2.63 Å | |||||||||
 データ登録者 データ登録者 | Chang C-W / Tsai M-D | |||||||||
| 資金援助 |  台湾, 2件 台湾, 2件
| |||||||||
 引用 引用 |  ジャーナル: Nat Metab / 年: 2023 ジャーナル: Nat Metab / 年: 2023タイトル: An ATP-sensitive phosphoketolase regulates carbon fixation in cyanobacteria. 著者: Kuan-Jen Lu / Chiung-Wen Chang / Chun-Hsiung Wang / Frederic Y-H Chen / Irene Y Huang / Pin-Hsuan Huang / Cheng-Han Yang / Hsiang-Yi Wu / Wen-Jin Wu / Kai-Cheng Hsu / Meng-Chiao Ho / Ming-Daw ...著者: Kuan-Jen Lu / Chiung-Wen Chang / Chun-Hsiung Wang / Frederic Y-H Chen / Irene Y Huang / Pin-Hsuan Huang / Cheng-Han Yang / Hsiang-Yi Wu / Wen-Jin Wu / Kai-Cheng Hsu / Meng-Chiao Ho / Ming-Daw Tsai / James C Liao /  要旨: Regulation of CO fixation in cyanobacteria is important both for the organism and global carbon balance. Here we show that phosphoketolase in Synechococcus elongatus PCC7942 (SeXPK) possesses a ...Regulation of CO fixation in cyanobacteria is important both for the organism and global carbon balance. Here we show that phosphoketolase in Synechococcus elongatus PCC7942 (SeXPK) possesses a distinct ATP-sensing mechanism, where a drop in ATP level allows SeXPK to divert precursors of the RuBisCO substrate away from the Calvin-Benson-Bassham cycle. Deleting the SeXPK gene increased CO fixation particularly during light-dark transitions. In high-density cultures, the Δxpk strain showed a 60% increase in carbon fixation and unexpectedly resulted in sucrose secretion without any pathway engineering. Using cryo-EM analysis, we discovered that these functions were enabled by a unique allosteric regulatory site involving two subunits jointly binding two ATP, which constantly suppresses the activity of SeXPK until the ATP level drops. This magnesium-independent ATP allosteric site is present in many species across all three domains of life, where it may also play important regulatory functions. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_35613.map.gz emd_35613.map.gz | 108.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-35613-v30.xml emd-35613-v30.xml emd-35613.xml emd-35613.xml | 17.1 KB 17.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_35613_fsc.xml emd_35613_fsc.xml | 13.3 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_35613.png emd_35613.png | 60.8 KB | ||
| Filedesc metadata |  emd-35613.cif.gz emd-35613.cif.gz | 6.5 KB | ||
| その他 |  emd_35613_half_map_1.map.gz emd_35613_half_map_1.map.gz emd_35613_half_map_2.map.gz emd_35613_half_map_2.map.gz | 200.5 MB 200.5 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-35613 http://ftp.pdbj.org/pub/emdb/structures/EMD-35613 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35613 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-35613 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_35613.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_35613.map.gz / 形式: CCP4 / 大きさ: 216 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 0.82 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_35613_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_35613_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Cyanobacteria XPK complexed with TPP/Mg2+
| 全体 | 名称: Cyanobacteria XPK complexed with TPP/Mg2+ |
|---|---|
| 要素 |
|
-超分子 #1: Cyanobacteria XPK complexed with TPP/Mg2+
| 超分子 | 名称: Cyanobacteria XPK complexed with TPP/Mg2+ / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Synechococcus (バクテリア) Synechococcus (バクテリア) |
| 分子量 | 理論値: 890 KDa |
-分子 #1: Probable phosphoketolase
| 分子 | 名称: Probable phosphoketolase / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Synechococcus elongatus (strain ATCC 33912 / PCC 7942 / FACHB-805) (バクテリア) Synechococcus elongatus (strain ATCC 33912 / PCC 7942 / FACHB-805) (バクテリア)株: ATCC 33912 / PCC 7942 / FACHB-805 |
| 分子量 | 理論値: 89.133211 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MTSTLQATDI ATLSPNEQAA IDAWWRAANY LSVGQIYLRD NPLLQEPLRP EHIKQRLLGH WGSDPGLSFV YVHLNRLIRR LDLNLIYVT GPGHGAPALL ANAWLEGTYS EVYPNCQQST AGLQQFFKQF SFPGGIGSHC TPETPGSIHE GGELGYSLSH A FGAALDNP ...文字列: MTSTLQATDI ATLSPNEQAA IDAWWRAANY LSVGQIYLRD NPLLQEPLRP EHIKQRLLGH WGSDPGLSFV YVHLNRLIRR LDLNLIYVT GPGHGAPALL ANAWLEGTYS EVYPNCQQST AGLQQFFKQF SFPGGIGSHC TPETPGSIHE GGELGYSLSH A FGAALDNP DLIVACVIGD GEAETGPLAT SWHSNKFLNP AQDGAVLPIL HLNGYKIANP TLLSRISHEE LRSLFIGYGY EP FFVEGND PAILHGVMAS TLATCVQKIQ AIQAAARSGE SSDRPMWPMI VLRTPKGWTG PATIKGHVVE GSWRSHQVPM ADV LTNPEH LQLLEDWLRS YRPEELFDAS GAPVAELQAI APIGDRRMSA NPVTNGGLLR RALTLPDFRD QAVSVPAPGK SRAD STRPL GQFLREVIRH NPDNFRLFGP DETASNRLDA VYEVTSKVWL GDRIPEDEDG GHLSDRGRVM EILSEHTLEG WLEAY LLTG RHGFFATYEA FAHVIDSMVN QHAKWLDVSK REVDWRAPVS SLNILLSSTV WRQDHNGFSH QDPGFIDLVT NKSARV TRI YLPPDANCLL SVADHCLRST DYINVIVADK QSHLQYLDAE AAARHCAKGI GIWDWASNDQ GASPDVVIAS CGDVVTL EA LAATALLREH FPDLKIRFVN VVDLFRLQPD TEHPHGLSDR DFDSLFTVDK PIIFNFHGYP WLIHKLAYRR HNHNNLHV R GYKEVGNINT PLELAIRNQV DRFNLAIDVI DRVPHLRDRG AHVKEWLKDQ IHDHIQYAYQ EGIDRPEINQ WQWPF UniProtKB:  UNIPROTKB: A0A8T9U4A0 UNIPROTKB: A0A8T9U4A0 |
-分子 #2: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 2 / コピー数: 2 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #3: THIAMINE DIPHOSPHATE
| 分子 | 名称: THIAMINE DIPHOSPHATE / タイプ: ligand / ID: 3 / コピー数: 2 / 式: TPP |
|---|---|
| 分子量 | 理論値: 425.314 Da |
| Chemical component information |  ChemComp-TPP: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 0.4 mg/mL |
|---|---|
| 緩衝液 | pH: 7.2 |
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 40 sec. |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 165000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 1.5 µm / 倍率(公称値): 165000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 撮影したグリッド数: 2 / 実像数: 3983 / 平均露光時間: 4.5 sec. / 平均電子線量: 59.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー















 Z
Z Y
Y X
X