+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Substrate-engaged TOM complex from yeast | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  Mitochondrial protein import (ミトコンドリア) / TOM / Mitochondrial protein import (ミトコンドリア) / TOM /  protein translocation / protein translocation /  PROTEIN TRANSPORT PROTEIN TRANSPORT | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報mitochondrial outer membrane translocase complex assembly / mitochondrial outer membrane translocase complex / protein import into mitochondrial matrix / protein targeting to mitochondrion / protein insertion into mitochondrial outer membrane /  porin activity / pore complex / protein transmembrane transporter activity / monoatomic ion transport / porin activity / pore complex / protein transmembrane transporter activity / monoatomic ion transport /  ミトコンドリア ...mitochondrial outer membrane translocase complex assembly / mitochondrial outer membrane translocase complex / protein import into mitochondrial matrix / protein targeting to mitochondrion / protein insertion into mitochondrial outer membrane / ミトコンドリア ...mitochondrial outer membrane translocase complex assembly / mitochondrial outer membrane translocase complex / protein import into mitochondrial matrix / protein targeting to mitochondrion / protein insertion into mitochondrial outer membrane /  porin activity / pore complex / protein transmembrane transporter activity / monoatomic ion transport / porin activity / pore complex / protein transmembrane transporter activity / monoatomic ion transport /  ミトコンドリア / mitochondrial outer membrane / ミトコンドリア / mitochondrial outer membrane /  ミトコンドリア / ミトコンドリア /  細胞質基質 細胞質基質類似検索 - 分子機能 | |||||||||
| 生物種 |   Saccharomyces cerevisiae S288C (パン酵母) / Saccharomyces cerevisiae S288C (パン酵母) /   Aequorea victoria (オワンクラゲ) Aequorea victoria (オワンクラゲ) | |||||||||
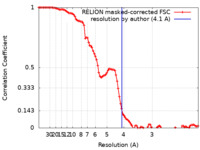
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 4.1 Å クライオ電子顕微鏡法 / 解像度: 4.1 Å | |||||||||
 データ登録者 データ登録者 | Zhou XY / Yang YQ / Wang GP / Wang SS | |||||||||
| 資金援助 |  中国, 1件 中国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2023 ジャーナル: Nat Struct Mol Biol / 年: 2023タイトル: Molecular pathway of mitochondrial preprotein import through the TOM-TIM23 supercomplex. 著者: Xueyin Zhou / Yuqi Yang / Guopeng Wang / Shanshan Wang / Dongjie Sun / Xiaomin Ou / Yuke Lian / Long Li /  要旨: Over half of mitochondrial proteins are imported from the cytosol via the pre-sequence pathway, controlled by the TOM complex in the outer membrane and the TIM23 complex in the inner membrane. The ...Over half of mitochondrial proteins are imported from the cytosol via the pre-sequence pathway, controlled by the TOM complex in the outer membrane and the TIM23 complex in the inner membrane. The mechanisms through which proteins are translocated via the TOM and TIM23 complexes remain unclear. Here we report the assembly of the active TOM-TIM23 supercomplex of Saccharomyces cerevisiae with translocating polypeptide substrates. Electron cryo-microscopy analyses reveal that the polypeptide substrates pass the TOM complex through the center of a Tom40 subunit, interacting with a glutamine-rich region. Structural and biochemical analyses show that the TIM23 complex contains a heterotrimer of the subunits Tim23, Tim17 and Mgr2. The polypeptide substrates are shielded from lipids by Mgr2 and Tim17, which creates a translocation pathway characterized by a negatively charged entrance and a central hydrophobic region. These findings reveal an unexpected pre-sequence pathway through the TOM-TIM23 supercomplex spanning the double membranes of mitochondria. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_34660.map.gz emd_34660.map.gz | 6.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-34660-v30.xml emd-34660-v30.xml emd-34660.xml emd-34660.xml | 18.8 KB 18.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_34660_fsc.xml emd_34660_fsc.xml | 10 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_34660.png emd_34660.png | 24.8 KB | ||
| Filedesc metadata |  emd-34660.cif.gz emd-34660.cif.gz | 6.5 KB | ||
| その他 |  emd_34660_half_map_1.map.gz emd_34660_half_map_1.map.gz emd_34660_half_map_2.map.gz emd_34660_half_map_2.map.gz | 65.4 MB 65.4 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-34660 http://ftp.pdbj.org/pub/emdb/structures/EMD-34660 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34660 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34660 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8hcoMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_34660.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_34660.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_34660_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_34660_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Substrate-engaged TOM complex from yeast
| 全体 | 名称: Substrate-engaged TOM complex from yeast |
|---|---|
| 要素 |
|
-超分子 #1: Substrate-engaged TOM complex from yeast
| 超分子 | 名称: Substrate-engaged TOM complex from yeast / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae S288C (パン酵母) Saccharomyces cerevisiae S288C (パン酵母) |
-分子 #1: Mitochondrial import receptor subunit TOM40
| 分子 | 名称: Mitochondrial import receptor subunit TOM40 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae S288C (パン酵母) / 株: S288c Saccharomyces cerevisiae S288C (パン酵母) / 株: S288c |
| 分子量 | 理論値: 42.071141 KDa |
| 配列 | 文字列: MSAPTPLAEA SQIPTIPALS PLTAKQSKGN FFSSNPISSF VVDTYKQLHS HRQSLELVNP GTVENLNKEV SRDVFLSQYF FTGLRADLN KAFSMNPAFQ TSHTFSIGSQ ALPKYAFSAL FANDNLFAQG NIDNDLSVSG RLNYGWDKKN ISKVNLQISD G QPTMCQLE ...文字列: MSAPTPLAEA SQIPTIPALS PLTAKQSKGN FFSSNPISSF VVDTYKQLHS HRQSLELVNP GTVENLNKEV SRDVFLSQYF FTGLRADLN KAFSMNPAFQ TSHTFSIGSQ ALPKYAFSAL FANDNLFAQG NIDNDLSVSG RLNYGWDKKN ISKVNLQISD G QPTMCQLE QDYQASDFSV NVKTLNPSFS EKGEFTGVAV ASFLQSVTPQ LALGLETLYS RTDGSAPGDA GVSYLTRYVS KK QDWIFSG QLQANGALIA SLWRKVAQNV EAGIETTLQA GMVPITDPLM GTPIGIQPTV EGSTTIGAKY EYRQSVYRGT LDS NGKVAC FLERKVLPTL SVLFCGEIDH FKNDTKIGCG LQFETAGNQE LLMLQQGLDA DGNPLQALPQ L UniProtKB: Mitochondrial import receptor subunit TOM40 |
-分子 #2: Mitochondrial import receptor subunit TOM22
| 分子 | 名称: Mitochondrial import receptor subunit TOM22 / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae S288C (パン酵母) / 株: ATCC 204508 / S288c Saccharomyces cerevisiae S288C (パン酵母) / 株: ATCC 204508 / S288c |
| 分子量 | 理論値: 16.801373 KDa |
| 配列 | 文字列: MVELTEIKDD VVQLDEPQFS RNQAIVEEKA SATNNDVVDD EDDSDSDFED EFDENETLLD RIVALKDIVP PGKRQTISNF FGFTSSFVR NAFTKSGNLA WTLTTTALLL GVPLSLSILA EQQLIEMEKT FDLQSDANNI LAQGEKDAAA TAN UniProtKB: Mitochondrial import receptor subunit TOM22 |
-分子 #3: Mitochondrial import receptor subunit TOM5
| 分子 | 名称: Mitochondrial import receptor subunit TOM5 / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae S288C (パン酵母) / 株: ATCC 204508 / S288c Saccharomyces cerevisiae S288C (パン酵母) / 株: ATCC 204508 / S288c |
| 分子量 | 理論値: 5.993924 KDa |
| 配列 | 文字列: MFGLPQQEVS EEEKRAHQEQ TEKTLKQAAY VAAFLWVSPM IWHLVKKQWK UniProtKB: Mitochondrial import receptor subunit TOM5 |
-分子 #4: Mitochondrial import receptor subunit TOM6
| 分子 | 名称: Mitochondrial import receptor subunit TOM6 / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae S288C (パン酵母) / 株: ATCC 204508 / S288c Saccharomyces cerevisiae S288C (パン酵母) / 株: ATCC 204508 / S288c |
| 分子量 | 理論値: 6.41046 KDa |
| 配列 | 文字列: MDGMFAMPGA AAGAASPQQP KSRFQAFKES PLYTIALNGA FFVAGVAFIQ SPLMDMLAPQ L UniProtKB: Mitochondrial import receptor subunit TOM6 |
-分子 #5: Mitochondrial import receptor subunit TOM7
| 分子 | 名称: Mitochondrial import receptor subunit TOM7 / タイプ: protein_or_peptide / ID: 5 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae S288C (パン酵母) / 株: S288c Saccharomyces cerevisiae S288C (パン酵母) / 株: S288c |
| 分子量 | 理論値: 6.876955 KDa |
| 配列 | 文字列: MSFLPSFILS DESKERISKI LTLTHNVAHY GWIPFVLYLG WAHTSNRPNF LNLLSPLPSV UniProtKB: Mitochondrial import receptor subunit TOM7 |
-分子 #6: sfGFP
| 分子 | 名称: sfGFP / タイプ: protein_or_peptide / ID: 6 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Aequorea victoria (オワンクラゲ) Aequorea victoria (オワンクラゲ) |
| 分子量 | 理論値: 61.514633 KDa |
| 組換発現 | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 配列 | 文字列: YGSTVPKSKS FEQDSSSVAY LNWHNGQIDN EPKLDMNKQK ISPAEVAKHN KPDDCWVVIN GYVYDLTRFL PNHPGMISLI AALAVDRVI GMENAMPWNL PADLAWFKRN TLNKPVIMGR HTWESIGRPL PGRKNIILSS QPGTDDRVTW VKSVDEAIAA S GDVPEIMV ...文字列: YGSTVPKSKS FEQDSSSVAY LNWHNGQIDN EPKLDMNKQK ISPAEVAKHN KPDDCWVVIN GYVYDLTRFL PNHPGMISLI AALAVDRVI GMENAMPWNL PADLAWFKRN TLNKPVIMGR HTWESIGRPL PGRKNIILSS QPGTDDRVTW VKSVDEAIAA S GDVPEIMV IGGGRVYEQF LPKAQKLYLT HIDAEVEGDT HFPDYEPDDW ESVFSEFHDA DAQNSHSYSF EILERRGQDV IK FNAGKDV TAIFEPLHAP NVIDKYIAPE KKLGPLQGSG SGSKGEELFT GVVPILVELD GDVNGHKFSV RGEGEGDATN GKL TLKFIC TTGKLPVPWP TLVTTF(GYS)VQC FSRYPDHMKR HDFFKSAMPE GYVQERTISF KDDGTYKTRA EVKFEGDTLV NRIELKGID FKEDGNILGH KLEYNFNSHN VYITADKQKN GIKANFKIRH NVEDGSVQLA DHYQQNTPIG DGPVLLPDNH Y LSTQSVLS KDPNEKRDHM VLLEFVTAAG ITHGSAGLEV LFQGPANGGS AWSHPQFEKG GGSGGGSGGG SWSHPQFEK |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 |
|---|---|
| グリッド | モデル: Quantifoil R1.2/1.3 / 材質: GOLD / メッシュ: 300 |
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 1.2 µm / 最小 デフォーカス(公称値): 1.0 µm Bright-field microscopy / 最大 デフォーカス(公称値): 1.2 µm / 最小 デフォーカス(公称値): 1.0 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 BIOQUANTUM (6k x 4k) 平均電子線量: 60.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー





 Z
Z Y
Y X
X