+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 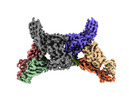 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
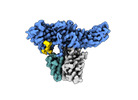
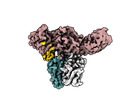
| タイトル | Structure of beta-arrestin2 in complex with a phosphopeptide corresponding to the human Atypical chemokine receptor 2, ACKR2 (D6R) | |||||||||||||||
 マップデータ マップデータ | Full map with pixel size 1.5375 | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード |  GPCR (Gタンパク質共役受容体) / GPCR (Gタンパク質共役受容体) /  Arrestin (アレスチン) / Arrestin (アレスチン) /  SIGNALING PROTEIN / SIGNALING PROTEIN /  SIGNALING PROTEIN-IMMUNE SYSTEM complex SIGNALING PROTEIN-IMMUNE SYSTEM complex | |||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 angiotensin receptor binding / angiotensin receptor binding /  chemokine receptor activity / desensitization of G protein-coupled receptor signaling pathway / C-C chemokine receptor activity / chemokine receptor activity / desensitization of G protein-coupled receptor signaling pathway / C-C chemokine receptor activity /  inositol hexakisphosphate binding / C-C chemokine binding / scavenger receptor activity / G protein-coupled receptor internalization / Chemokine receptors bind chemokines / phosphatidylinositol-3,4,5-trisphosphate binding ... inositol hexakisphosphate binding / C-C chemokine binding / scavenger receptor activity / G protein-coupled receptor internalization / Chemokine receptors bind chemokines / phosphatidylinositol-3,4,5-trisphosphate binding ... angiotensin receptor binding / angiotensin receptor binding /  chemokine receptor activity / desensitization of G protein-coupled receptor signaling pathway / C-C chemokine receptor activity / chemokine receptor activity / desensitization of G protein-coupled receptor signaling pathway / C-C chemokine receptor activity /  inositol hexakisphosphate binding / C-C chemokine binding / scavenger receptor activity / G protein-coupled receptor internalization / Chemokine receptors bind chemokines / phosphatidylinositol-3,4,5-trisphosphate binding / positive regulation of receptor internalization / endocytic vesicle / inositol hexakisphosphate binding / C-C chemokine binding / scavenger receptor activity / G protein-coupled receptor internalization / Chemokine receptors bind chemokines / phosphatidylinositol-3,4,5-trisphosphate binding / positive regulation of receptor internalization / endocytic vesicle /  クラスリン / cell chemotaxis / クラスリン / cell chemotaxis /  phosphatidylinositol binding / phosphatidylinositol binding /  マイクロフィラメント / calcium-mediated signaling / マイクロフィラメント / calcium-mediated signaling /  receptor internalization / recycling endosome / receptor internalization / recycling endosome /  protein transport / positive regulation of cytosolic calcium ion concentration / protein transport / positive regulation of cytosolic calcium ion concentration /  核膜 / positive regulation of ERK1 and ERK2 cascade / molecular adaptor activity / 核膜 / positive regulation of ERK1 and ERK2 cascade / molecular adaptor activity /  エンドソーム / intracellular signal transduction / エンドソーム / intracellular signal transduction /  免疫応答 / 免疫応答 /  炎症 / external side of plasma membrane / intracellular membrane-bounded organelle / 炎症 / external side of plasma membrane / intracellular membrane-bounded organelle /  シグナル伝達 / シグナル伝達 /  核質 / 核質 /  細胞核 / 細胞核 /  細胞膜 / 細胞膜 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||||||||
| 生物種 |   Bos taurus (ウシ) / Bos taurus (ウシ) /   Mus musculus (ハツカネズミ) / Mus musculus (ハツカネズミ) /   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.35 Å クライオ電子顕微鏡法 / 解像度: 3.35 Å | |||||||||||||||
 データ登録者 データ登録者 | Maharana J / Sarma P / Yadav MK / Banerjee R / Shukla AK | |||||||||||||||
| 資金援助 |  インド, 4件 インド, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Science / 年: 2024 ジャーナル: Science / 年: 2024タイトル: Molecular insights into atypical modes of β-arrestin interaction with seven transmembrane receptors. 著者: Jagannath Maharana / Fumiya K Sano / Parishmita Sarma / Manish K Yadav / Longhan Duan / Tomasz M Stepniewski / Madhu Chaturvedi / Ashutosh Ranjan / Vinay Singh / Sayantan Saha / Gargi Mahajan ...著者: Jagannath Maharana / Fumiya K Sano / Parishmita Sarma / Manish K Yadav / Longhan Duan / Tomasz M Stepniewski / Madhu Chaturvedi / Ashutosh Ranjan / Vinay Singh / Sayantan Saha / Gargi Mahajan / Mohamed Chami / Wataru Shihoya / Jana Selent / Ka Young Chung / Ramanuj Banerjee / Osamu Nureki / Arun K Shukla /      要旨: β-arrestins (βarrs) are multifunctional proteins involved in signaling and regulation of seven transmembrane receptors (7TMRs), and their interaction is driven primarily by agonist-induced receptor ...β-arrestins (βarrs) are multifunctional proteins involved in signaling and regulation of seven transmembrane receptors (7TMRs), and their interaction is driven primarily by agonist-induced receptor activation and phosphorylation. Here, we present seven cryo-electron microscopy structures of βarrs either in the basal state, activated by the muscarinic receptor subtype 2 (M2R) through its third intracellular loop, or activated by the βarr-biased decoy D6 receptor (D6R). Combined with biochemical, cellular, and biophysical experiments, these structural snapshots allow the visualization of atypical engagement of βarrs with 7TMRs and also reveal a structural transition in the carboxyl terminus of βarr2 from a β strand to an α helix upon activation by D6R. Our study provides previously unanticipated molecular insights into the structural and functional diversity encoded in 7TMR-βarr complexes with direct implications for exploring novel therapeutic avenues. | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_34174.map.gz emd_34174.map.gz | 59.5 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-34174-v30.xml emd-34174-v30.xml emd-34174.xml emd-34174.xml | 23.7 KB 23.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_34174.png emd_34174.png | 79.5 KB | ||
| Filedesc metadata |  emd-34174.cif.gz emd-34174.cif.gz | 7.2 KB | ||
| その他 |  emd_34174_half_map_1.map.gz emd_34174_half_map_1.map.gz emd_34174_half_map_2.map.gz emd_34174_half_map_2.map.gz | 59.2 MB 59.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-34174 http://ftp.pdbj.org/pub/emdb/structures/EMD-34174 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34174 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-34174 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8go9MC  8j8rC  8j8vC  8j8zC  8j97C  8j9kC  8ja3C  8jafC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_34174.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_34174.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Full map with pixel size 1.5375 | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.5375 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half map A with pixel size 1.5375
| ファイル | emd_34174_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A with pixel size 1.5375 | ||||||||||||
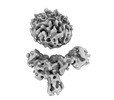
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map B with pixel size 1.5375
| ファイル | emd_34174_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B with pixel size 1.5375 | ||||||||||||
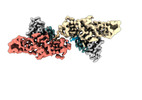
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Peptide bound beta-arrestin2 in complex with Fab30
| 全体 | 名称: Peptide bound beta-arrestin2 in complex with Fab30 |
|---|---|
| 要素 |
|
-超分子 #1: Peptide bound beta-arrestin2 in complex with Fab30
| 超分子 | 名称: Peptide bound beta-arrestin2 in complex with Fab30 / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: all |
|---|---|
| 分子量 | 理論値: 190 KDa |
-超分子 #2: beta-arrestin2
| 超分子 | 名称: beta-arrestin2 / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Bos taurus (ウシ) Bos taurus (ウシ) |
-超分子 #3: Fab30
| 超分子 | 名称: Fab30 / タイプ: complex / ID: 3 / 親要素: 2 / 含まれる分子: #2-#3 |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
-超分子 #4: Atypical chemokine receptor 2
| 超分子 | 名称: Atypical chemokine receptor 2 / タイプ: complex / ID: 4 / 親要素: 3 / 含まれる分子: #4 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) / Synthetically produced: Yes Homo sapiens (ヒト) / Synthetically produced: Yes |
-分子 #1: Beta-arrestin-2
| 分子 | 名称: Beta-arrestin-2 / タイプ: protein_or_peptide / ID: 1 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Bos taurus (ウシ) Bos taurus (ウシ) |
| 分子量 | 理論値: 47.217676 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MGEKPGTRVF KKSSPNGKLT VYLGKRDFVD HLDKVDPVDG VVLVDPDYLK DRKVFVTLTV AFRYGREDCD VLGLSFRKDL FIANYQAFP PTPNPPRPPT RLQERLLRKL GQHAHPFFFT IPQNLPSSVT LQPGPEDTGK ALGVDFEIRA FVAKSLEEKS H KRNSVRLV ...文字列: MGEKPGTRVF KKSSPNGKLT VYLGKRDFVD HLDKVDPVDG VVLVDPDYLK DRKVFVTLTV AFRYGREDCD VLGLSFRKDL FIANYQAFP PTPNPPRPPT RLQERLLRKL GQHAHPFFFT IPQNLPSSVT LQPGPEDTGK ALGVDFEIRA FVAKSLEEKS H KRNSVRLV IRKVQFAPEK PGPQPSAETT RHFLMSDRSL HLEASLDKEL YYHGEPLNVN VHVTNNSTKT VKKIKVSVRQ YA DIVLFST AQYKVPVAQV EQDDQVSPSS TFSKVYTITP FLANNREKRG LALDGKLKHE DTNLASSTIV KEGANKEVLG ILV SYRVKV KLVVSRGGDV SVELPFVLMH PKPHDHIALP RPQSAATHPP TLLPSAVPET DAPVDTNLIE FETNYATDDD IVFE DFARL RLKGLKDEDY DDQFC UniProtKB:  Beta-arrestin-2 Beta-arrestin-2 |
-分子 #2: Fab30 Heavy Chain
| 分子 | 名称: Fab30 Heavy Chain / タイプ: protein_or_peptide / ID: 2 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
| 分子量 | 理論値: 25.512354 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: EISEVQLVES GGGLVQPGGS LRLSCAASGF NVYSSSIHWV RQAPGKGLEW VASISSYYGY TYYADSVKGR FTISADTSKN TAYLQMNSL RAEDTAVYYC ARSRQFWYSG LDYWGQGTLV TVSSASTKGP SVFPLAPSSK STSGGTAALG CLVKDYFPEP V TVSWNSGA ...文字列: EISEVQLVES GGGLVQPGGS LRLSCAASGF NVYSSSIHWV RQAPGKGLEW VASISSYYGY TYYADSVKGR FTISADTSKN TAYLQMNSL RAEDTAVYYC ARSRQFWYSG LDYWGQGTLV TVSSASTKGP SVFPLAPSSK STSGGTAALG CLVKDYFPEP V TVSWNSGA LTSGVHTFPA VLQSSGLYSL SSVVTVPSSS LGTQTYICNV NHKPSNTKVD KKVEPKSCDK THHHHHHHH |
-分子 #3: Fab30 Light Chain
| 分子 | 名称: Fab30 Light Chain / タイプ: protein_or_peptide / ID: 3 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
| 分子量 | 理論値: 23.435064 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: SDIQMTQSPS SLSASVGDRV TITCRASQSV SSAVAWYQQK PGKAPKLLIY SASSLYSGVP SRFSGSRSGT DFTLTISSLQ PEDFATYYC QQYKYVPVTF GQGTKVEIKR TVAAPSVFIF PPSDSQLKSG TASVVCLLNN FYPREAKVQW KVDNALQSGN S QESVTEQD ...文字列: SDIQMTQSPS SLSASVGDRV TITCRASQSV SSAVAWYQQK PGKAPKLLIY SASSLYSGVP SRFSGSRSGT DFTLTISSLQ PEDFATYYC QQYKYVPVTF GQGTKVEIKR TVAAPSVFIF PPSDSQLKSG TASVVCLLNN FYPREAKVQW KVDNALQSGN S QESVTEQD SKDSTYSLSS TLTLSKADYE KHKVYACEVT HQGLSSPVTK SFNRGEC |
-分子 #4: Atypical chemokine receptor 2
| 分子 | 名称: Atypical chemokine receptor 2 / タイプ: protein_or_peptide / ID: 4 / コピー数: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 2.352668 KDa |
| 配列 | 文字列: G(TPO)AQA(SEP)L(SEP)(SEP)C (SEP)E(SEP)(SEP)IL(TPO)A UniProtKB: Atypical chemokine receptor 2 |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 構成要素:
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 300 / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 45 sec. | |||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 90 % / チャンバー内温度: 283.15 K / 装置: LEICA EM GP / 詳細: Blotted for 3 seconds before plunging.. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 165000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm / 倍率(公称値): 165000 |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 SUMMIT (4k x 4k) 検出モード: COUNTING / 実像数: 10028 / 平均電子線量: 56.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 粒子像選択 | 選択した数: 1827629 |
|---|---|
| 初期モデル | モデルのタイプ: PDB ENTRY PDBモデル - PDB ID: |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 3次元分類 | ソフトウェア - 名称: cryoSPARC (ver. 3.3.1) |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD |
| 最終 再構成 | 想定した対称性 - 点群: C2 (2回回転対称 ) / 解像度のタイプ: BY AUTHOR / 解像度: 3.35 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC (ver. 3.3.1) / 使用した粒子像数: 94497 ) / 解像度のタイプ: BY AUTHOR / 解像度: 3.35 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC (ver. 3.3.1) / 使用した粒子像数: 94497 |
 ムービー
ムービー コントローラー
コントローラー




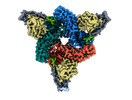
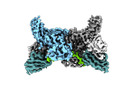
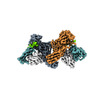










 Z
Z Y
Y X
X


















