+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 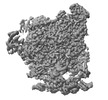 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | EsN-dhsU36duplex full map (map 1) | |||||||||
 マップデータ マップデータ | Raw map | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード | promoter-bound / initiation /  TRANSCRIPTION (転写 (生物学)) TRANSCRIPTION (転写 (生物学)) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 RNA polymerase complex / submerged biofilm formation / cellular response to cell envelope stress / cytosolic DNA-directed RNA polymerase complex / regulation of DNA-templated transcription initiation / bacterial-type flagellum assembly / bacterial-type flagellum-dependent cell motility / nitrate assimilation / transcription elongation factor complex / regulation of DNA-templated transcription elongation ... RNA polymerase complex / submerged biofilm formation / cellular response to cell envelope stress / cytosolic DNA-directed RNA polymerase complex / regulation of DNA-templated transcription initiation / bacterial-type flagellum assembly / bacterial-type flagellum-dependent cell motility / nitrate assimilation / transcription elongation factor complex / regulation of DNA-templated transcription elongation ... RNA polymerase complex / submerged biofilm formation / cellular response to cell envelope stress / cytosolic DNA-directed RNA polymerase complex / regulation of DNA-templated transcription initiation / bacterial-type flagellum assembly / bacterial-type flagellum-dependent cell motility / nitrate assimilation / transcription elongation factor complex / regulation of DNA-templated transcription elongation / transcription antitermination / RNA polymerase complex / submerged biofilm formation / cellular response to cell envelope stress / cytosolic DNA-directed RNA polymerase complex / regulation of DNA-templated transcription initiation / bacterial-type flagellum assembly / bacterial-type flagellum-dependent cell motility / nitrate assimilation / transcription elongation factor complex / regulation of DNA-templated transcription elongation / transcription antitermination /  運動性 / DNA-templated transcription initiation / 運動性 / DNA-templated transcription initiation /  ribonucleoside binding / DNA-directed 5'-3' RNA polymerase activity / ribonucleoside binding / DNA-directed 5'-3' RNA polymerase activity /  ポリメラーゼ / response to heat / protein-containing complex assembly / intracellular iron ion homeostasis / ポリメラーゼ / response to heat / protein-containing complex assembly / intracellular iron ion homeostasis /  protein dimerization activity / response to antibiotic / magnesium ion binding / protein dimerization activity / response to antibiotic / magnesium ion binding /  DNA binding / zinc ion binding / DNA binding / zinc ion binding /  生体膜 / 生体膜 /  細胞質基質 / 細胞質基質 /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |   Escherichia coli (大腸菌) / Escherichia coli (大腸菌) /    Aquifex aeolicus (バクテリア) Aquifex aeolicus (バクテリア) | |||||||||
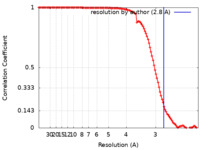
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.8 Å クライオ電子顕微鏡法 / 解像度: 2.8 Å | |||||||||
 データ登録者 データ登録者 | Mueller AU / Chen J / Darst SA | |||||||||
| 資金援助 |  米国, 1件 米国, 1件
| |||||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2023 ジャーナル: Proc Natl Acad Sci U S A / 年: 2023タイトル: A general mechanism for transcription bubble nucleation in bacteria. 著者: Andreas U Mueller / James Chen / Mengyu Wu / Courtney Chiu / B Tracy Nixon / Elizabeth A Campbell / Seth A Darst /  要旨: Bacterial transcription initiation requires σ factors for nucleation of the transcription bubble. The canonical housekeeping σ factor, σ, nucleates DNA melting via recognition of conserved bases ...Bacterial transcription initiation requires σ factors for nucleation of the transcription bubble. The canonical housekeeping σ factor, σ, nucleates DNA melting via recognition of conserved bases of the promoter -10 motif, which are unstacked and captured in pockets of σ. By contrast, the mechanism of transcription bubble nucleation and formation during the unrelated σ-mediated transcription initiation is poorly understood. Herein, we combine structural and biochemical approaches to establish that σ, like σ, captures a flipped, unstacked base in a pocket formed between its N-terminal region I (RI) and extra-long helix features. Strikingly, RI inserts into the nascent bubble to stabilize the nucleated bubble prior to engagement of the obligate ATPase activator. Our data suggest a general paradigm of transcription initiation that requires σ factors to nucleate an early melted intermediate prior to productive RNA synthesis. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28786.map.gz emd_28786.map.gz | 65.3 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28786-v30.xml emd-28786-v30.xml emd-28786.xml emd-28786.xml | 31 KB 31 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_28786_fsc.xml emd_28786_fsc.xml | 11.2 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_28786.png emd_28786.png | 105.9 KB | ||
| マスクデータ |  emd_28786_msk_1.map emd_28786_msk_1.map emd_28786_msk_2.map emd_28786_msk_2.map | 129.7 MB 129.7 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-28786.cif.gz emd-28786.cif.gz | 8.2 KB | ||
| その他 |  emd_28786_additional_1.map.gz emd_28786_additional_1.map.gz emd_28786_additional_2.map.gz emd_28786_additional_2.map.gz emd_28786_additional_3.map.gz emd_28786_additional_3.map.gz emd_28786_half_map_1.map.gz emd_28786_half_map_1.map.gz emd_28786_half_map_2.map.gz emd_28786_half_map_2.map.gz | 122.4 MB 4 MB 4.6 MB 120.6 MB 120.6 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28786 http://ftp.pdbj.org/pub/emdb/structures/EMD-28786 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28786 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28786 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8f1iC  8f1jC  8f1kC C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28786.map.gz / 形式: CCP4 / 大きさ: 129.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28786.map.gz / 形式: CCP4 / 大きさ: 129.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Raw map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.083 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_28786_msk_1.map emd_28786_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-マスク #2
| ファイル |  emd_28786_msk_2.map emd_28786_msk_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: B-factor sharpened map (-121.3)
| ファイル | emd_28786_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | B-factor sharpened map (-121.3) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Locally filtered map (blocfilt)
| ファイル | emd_28786_additional_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Locally filtered map (blocfilt) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-追加マップ: Local resolution map (blocres)
| ファイル | emd_28786_additional_3.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Local resolution map (blocres) | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map A
| ファイル | emd_28786_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map B
| ファイル | emd_28786_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : EsN-dhsU36duplex
| 全体 | 名称: EsN-dhsU36duplex |
|---|---|
| 要素 |
|
-超分子 #1: EsN-dhsU36duplex
| 超分子 | 名称: EsN-dhsU36duplex / タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#7 |
|---|
-分子 #1: RNA polymerase alpha subunit
| 分子 | 名称: RNA polymerase alpha subunit / タイプ: protein_or_peptide / ID: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MQGSVTEFLK PRLVDIEQVS STHAKVTLEP LERGFGHTLG NALRRILLSS MPGCAVTEVE IDGVLHEYST KEGVQEDILE ILLNLKGLA VRVQGKDEVI LTLNKSGIGP VTAADITHDG DVEIVKPQHV ICHLTDENAS ISMRIKVQRG RGYVPASTRI H SEEDERPI ...文字列: MQGSVTEFLK PRLVDIEQVS STHAKVTLEP LERGFGHTLG NALRRILLSS MPGCAVTEVE IDGVLHEYST KEGVQEDILE ILLNLKGLA VRVQGKDEVI LTLNKSGIGP VTAADITHDG DVEIVKPQHV ICHLTDENAS ISMRIKVQRG RGYVPASTRI H SEEDERPI GRLLVDACYS PVERIAYNVE AARVEQRTDL DKLVIEMETN GTIDPEEAIR RAATILAEQL EAFVDLRDVR QP EVKEEKP EFDPILLRPV DDLELTVRSA NCLKAEAIHY IGDLVQRTEV ELLKTPNLGK KSLTEIKDVL ASRGLSLGMR LEN WPPASI ADE UniProtKB:  DNA-directed RNA polymerase subunit alpha DNA-directed RNA polymerase subunit alpha |
-分子 #2: RNA polymerase beta subunit
| 分子 | 名称: RNA polymerase beta subunit / タイプ: protein_or_peptide / ID: 2 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MVYSYTEKKR IRKDFGKRPQ VLDVPYLLSI QLDSFQKFIE QDPEGQYGLE AAFRSVFPIQ SYSGNSELQY VSYRLGEPVF DVQECQIRG VTYSAPLRVK LRLVIYEREA PEGTVKDIKE QEVYMGEIPL MTDNGTFVIN GTERVIVSQL HRSPGVFFDS D KGKTHSSG ...文字列: MVYSYTEKKR IRKDFGKRPQ VLDVPYLLSI QLDSFQKFIE QDPEGQYGLE AAFRSVFPIQ SYSGNSELQY VSYRLGEPVF DVQECQIRG VTYSAPLRVK LRLVIYEREA PEGTVKDIKE QEVYMGEIPL MTDNGTFVIN GTERVIVSQL HRSPGVFFDS D KGKTHSSG KVLYNARIIP YRGSWLDFEF DPKDNLFVRI DRRRKLPATI ILRALNYTTE QILDLFFEKV IFEIRDNKLQ ME LVPERLR GETASFDIEA NGKVYVEKGR RITARHIRQL EKDDVKLIEV PVEYIAGKVV AKDYIDESTG ELICAANMEL SLD LLAKLS QSGHKRIETL FTNDLDHGPY ISETLRVDPT NDRLSALVEI YRMMRPGEPP TREAAESLFE NLFFSEDRYD LSAV GRMKF NRSLLREEIE GSGILSKDDI IDVMKKLIDI RNGKGEVDDI DHLGNRRIRS VGEMAENQFR VGLVRVERAV KERLS LGDL DTLMPQDMIN AKPISAAVKE FFGSSQLSQF MDQNNPLSEI THKRRISALG PGGLTRERAG FEVRDVHPTH YGRVCP IET PEGPNIGLIN SLSVYAQTNE YGFLETPYRK VTDGVVTDEI HYLSAIEEGN YVIAQANSNL DEEGHFVEDL VTCRSKG ES SLFSRDQVDY MDVSTQQVVS VGASLIPFLE HDDANRALMG ANMQRQAVPT LRADKPLVGT GMERAVAVDS GVTAVAKR G GVVQYVDASR IVIKVNEDEM YPGEAGIDIY NLTKYTRSNQ NTCINQMPCV SLGEPVERGD VLADGPSTDL GELALGQNM RVAFMPWNGY NFEDSILVSE RVVQEDRFTT IHIQELACVS RDTKLGPEEI TADIPNVGEA ALSKLDESGI VYIGAEVTGG DILVGKVTP KGETQLTPEE KLLRAIFGEK ASDVKDSSLR VPNGVSGTVI DVQVFTRDGV EKDKRALEIE EMQLKQAKKD L SEELQILE AGLFSRIRAV LVAGGVEAEK LDKLPRDRWL ELGLTDEEKQ NQLEQLAEQY DELKHEFEKK LEAKRRKITQ GD DLAPGVL KIVKVYLAVK RRIQPGDKMA GRHGNKGVIS KINPIEDMPY DENGTPVDIV LNPLGVPSRM NIGQILETHL GMA AKGIGD KINAMLKQQQ EVAKLREFIQ RAYDLGADVR QKVDLSTFSD EEVMRLAENL RKGMPIATPV FDGAKEAEIK ELLK LGDLP TSGQIRLYDG RTGEQFERPV TVGYMYMLKL NHLVDDKMHA RSTGSYSLVT QQPLGGKAQF GGQRFGEMEV WALEA YGAA YTLQEMLTVK SDDVNGRTKM YKNIVDGNHQ MEPGMPESFN VLLKEIRSLG INIELEDE UniProtKB:  DNA-directed RNA polymerase subunit beta DNA-directed RNA polymerase subunit beta |
-分子 #3: RNA polymerase beta' subunit
| 分子 | 名称: RNA polymerase beta' subunit / タイプ: protein_or_peptide / ID: 3 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MKDLLKFLKA QTKTEEFDAI KIALASPDMI RSWSFGEVKK PETINYRTFK PERDGLFCAR IFGPVKDYEC LCGKYKRLKH RGVICEKCG VEVTQTKVRR ERMGHIELAS PTAHIWFLKS LPSRIGLLLD MPLRDIERVL YFESYVVIEG GMTNLERQQI L TEEQYLDA ...文字列: MKDLLKFLKA QTKTEEFDAI KIALASPDMI RSWSFGEVKK PETINYRTFK PERDGLFCAR IFGPVKDYEC LCGKYKRLKH RGVICEKCG VEVTQTKVRR ERMGHIELAS PTAHIWFLKS LPSRIGLLLD MPLRDIERVL YFESYVVIEG GMTNLERQQI L TEEQYLDA LEEFGDEFDA KMGAEAIQAL LKSMDLEQEC EQLREELNET NSETKRKKLT KRIKLLEAFV QSGNKPEWMI LT VLPVLPP DLRPLVPLDG GRFATSDLND LYRRVINRNN RLKRLLDLAA PDIIVRNEKR MLQEAVDALL DNGRRGRAIT GSN KRPLKS LADMIKGKQG RFRQNLLGKR VDYSGRSVIT VGPYLRLHQC GLPKKMALEL FKPFIYGKLE LRGLATTIKA AKKM VEREE AVVWDILDEV IREHPVLLNR APTLHRLGIQ AFEPVLIEGK AIQLHPLVCA AYNADFDGDQ MAVHVPLTLE AQLEA RALM MSTNNILSPA NGEPIIVPSQ DVVLGLYYMT RDCVNAKGEG MVLTGPKEAE RLYRSGLASL HARVKVRITE YEKDAN GEL VAKTSLKDTT VGRAILWMIV PKGLPYSIVN QALGKKAISK MLNTCYRILG LKPTVIFADQ IMYTGFAYAA RSGASVG ID DMVIPEKKHE IISEAEAEVA EIQEQFQSGL VTAGERYNKV IDIWAAANDR VSKAMMDNLQ TETVINRDGQ EEKQVSFN S IYMMADSGAR GSAAQIRQLA GMRGLMAKPD GSIIETPITA NFREGLNVLQ YFISTHGARK GLADTALKTA NSGYLTRRL VDVAQDLVVT EDDCGTHEGI MMTPVIEGGD VKEPLRDRVL GRVTAEDVLK PGTADILVPR NTLLHEQWCD LLEENSVDAV KVRSVVSCD TDFGVCAHCY GRDLARGHII NKGEAIGVIA AQSIGEPGTQ LTMRTFHIGG AASRAAAESS IQVKNKGSIK L SNVKSVVN SSGKLVITSR NTELKLIDEF GRTKESYKVP YGAVLAKGDG EQVAGGETVA NWDPHTMPVI TEVSGFVRFT DM IDGQTIT RQTDELTGLS SLVVLDSAER TAGGKDLRPA LKIVDAQGND VLIPGTDMPA QYFLPGKAIV QLEDGVQISS GDT LARIPQ ESGGTKDITG GLPRVADLFE ARRPKEPAIL AEISGIVSFG KETKGKRRLV ITPVDGSDPY EEMIPKWRQL NVFE GERVE RGDVISDGPE APHDILRLRG VHAVTRYIVN EVQDVYRLQG VKINDKHIEV IVRQMLRKAT IVNAGSSDFL EGEQV EYSR VKIANRELEA NGKVGATYSR DLLGITKASL ATESFISAAS FQETTRVLTE AAVAGKRDEL RGLKENVIVG RLIPAG TGY AYHQDRMRRR AAGEAPAAPQ VTAEDASASL AELLNAGLGG SDNELELEVL FQGPSSGHHH HHHHHHH UniProtKB:  DNA-directed RNA polymerase subunit beta' DNA-directed RNA polymerase subunit beta' |
-分子 #4: RNA polymerase omega subunit
| 分子 | 名称: RNA polymerase omega subunit / タイプ: protein_or_peptide / ID: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MARVTVQDAV EKIGNRFDLV LVAARRARQM QVGGKDPLVP EENDKTTVIA LREIEEGLIN NQILDVRERQ EQQEQEAAEL QAVTAIAEGR R UniProtKB:  DNA-directed RNA polymerase subunit omega DNA-directed RNA polymerase subunit omega |
-分子 #7: RNA polymerase sigma-54 factor
| 分子 | 名称: RNA polymerase sigma-54 factor / タイプ: protein_or_peptide / ID: 7 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: SEFMKQGLQL RLSQQLAMTP QLQQAIRLLQ LSTLELQQEL QQALESNPLL EQIDTHEEID TRETQDSETL DTADALEQKE MPEELPLDA SWDTIYTAGT PSGTSGDYID DELPVYQGET TQTLQDYLMW QVELTPFSDT DRAIATSIVD AVDETGYLTV P LEDILESI ...文字列: SEFMKQGLQL RLSQQLAMTP QLQQAIRLLQ LSTLELQQEL QQALESNPLL EQIDTHEEID TRETQDSETL DTADALEQKE MPEELPLDA SWDTIYTAGT PSGTSGDYID DELPVYQGET TQTLQDYLMW QVELTPFSDT DRAIATSIVD AVDETGYLTV P LEDILESI GDEEIDIDEV EAVLKRIQRF DPVGVAAKDL RDCLLIQLSQ FDKTTPWLEE ARLIISDHLD LLANHDFRTL MR VTRLKED VLKEAVNLIQ SLDPRPGQSI QTGEPEYVIP DVLVRKHNGH WTVELNSDSI PRLQINQHYA SMCNNARNDG DSQ FIRSNL QDAKWLIKSL ESRNDTLLRV SRCIVEQQQA FFEQGEEYMK PMVLADIAQA VEMHESTISR VTTQKYLHSP RGIF ELKYF FSSHVNTEGG GEASSTAIRA LVKKLIAAEN PAKPLSDSKL TSLLSEQGIM VARRTVAKYR ESLSIPPSNQ RKQLV |
-分子 #5: dhsU36duplex top strand
| 分子 | 名称: dhsU36duplex top strand / タイプ: dna / ID: 5 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:    Aquifex aeolicus (バクテリア) Aquifex aeolicus (バクテリア) |
| 配列 | 文字列: (DC)(DC)(DA)(DG)(DA)(DA)(DA)(DT)(DT)(DG) (DG)(DC)(DA)(DC)(DG)(DA)(DA)(DA)(DA)(DT) (DT)(DG)(DC)(DA)(DA)(DT)(DA)(DA)(DA) (DT)(DA)(DC)(DA)(DA)(DC)(DG) |
-分子 #6: dhsU36duplex bottom strand
| 分子 | 名称: dhsU36duplex bottom strand / タイプ: dna / ID: 6 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:    Aquifex aeolicus (バクテリア) Aquifex aeolicus (バクテリア) |
| 配列 | 文字列: (DC)(DG)(DT)(DT)(DG)(DT)(DA)(DT)(DT)(DT) (DA)(DT)(DT)(DG)(DC)(DA)(DA)(DT)(DT)(DT) (DT)(DC)(DG)(DT)(DG)(DC)(DC)(DA)(DA) (DT)(DT)(DT)(DC)(DT)(DG)(DG) |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 8 構成要素:
| |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| グリッド | モデル: C-flat-1.2/1.3 / 材質: GOLD / メッシュ: 400 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: HOLEY / 前処理 - タイプ: GLOW DISCHARGE / 前処理 - 時間: 25 sec. / 前処理 - 雰囲気: AIR 詳細: Self-built glow discharge chamber was evacuated with a vacuum pump prior to application of voltage (no pressure and/or voltage readings available) | |||||||||||||||
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 295 K / 装置: FEI VITROBOT MARK IV 詳細: Octyl beta-D-glucopyranoside (beta-OG) was added to the sample to 0.1%w/v final concentration (from 10x stock) just prior to plunge vitrification. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.8 µm Bright-field microscopy / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 実像数: 6088 / 平均露光時間: 2.0 sec. / 平均電子線量: 51.32 e/Å2 詳細: dose-fractionation with 0.05 seconds per frame (=40 frames) |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー













 Z
Z Y
Y X
X