+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of single homo-hexameric Holliday junction ATP-dependent DNA helicase RuvB motor | |||||||||
 マップデータ マップデータ | ||||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報 Holliday junction resolvase complex / four-way junction helicase activity / four-way junction DNA binding / DNA recombination / Holliday junction resolvase complex / four-way junction helicase activity / four-way junction DNA binding / DNA recombination /  ヘリカーゼ / ヘリカーゼ /  DNA修復 / DNA修復 /  ATP hydrolysis activity / ATP hydrolysis activity /  ATP binding / ATP binding /  細胞質 細胞質類似検索 - 分子機能 | |||||||||
| 生物種 |    Thermus thermophilus HB8 (サーマス・サーモフィルス) Thermus thermophilus HB8 (サーマス・サーモフィルス) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.97 Å クライオ電子顕微鏡法 / 解像度: 2.97 Å | |||||||||
 データ登録者 データ登録者 | Shen ZF / Rish AD / Fu TM | |||||||||
| 資金援助 | 1件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: Structure of single homo-hexameric Holliday junction ATP-dependent DNA helicase RuvB motor 著者: Shen ZF / Rish AD / Fu TM | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_28101.map.gz emd_28101.map.gz | 36.2 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-28101-v30.xml emd-28101-v30.xml emd-28101.xml emd-28101.xml | 15.7 KB 15.7 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_28101_fsc.xml emd_28101_fsc.xml | 7.1 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_28101.png emd_28101.png | 66.3 KB | ||
| その他 |  emd_28101_half_map_1.map.gz emd_28101_half_map_1.map.gz emd_28101_half_map_2.map.gz emd_28101_half_map_2.map.gz | 35.7 MB 35.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-28101 http://ftp.pdbj.org/pub/emdb/structures/EMD-28101 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28101 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-28101 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8efvMC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_28101.map.gz / 形式: CCP4 / 大きさ: 38.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_28101.map.gz / 形式: CCP4 / 大きさ: 38.4 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_28101_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_28101_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Single homo-hexameric AAA+ ATPase RuvB motor
| 全体 | 名称: Single homo-hexameric AAA+ ATPase RuvB motor |
|---|---|
| 要素 |
|
-超分子 #1: Single homo-hexameric AAA+ ATPase RuvB motor
| 超分子 | 名称: Single homo-hexameric AAA+ ATPase RuvB motor / タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: #1-#3 |
|---|---|
| 由来(天然) | 生物種:    Thermus thermophilus HB8 (サーマス・サーモフィルス) Thermus thermophilus HB8 (サーマス・サーモフィルス) |
-分子 #1: Holliday junction ATP-dependent DNA helicase RuvB
| 分子 | 名称: Holliday junction ATP-dependent DNA helicase RuvB / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO / EC番号:  ヘリカーゼ ヘリカーゼ |
|---|---|
| 由来(天然) | 生物種:    Thermus thermophilus HB8 (サーマス・サーモフィルス) Thermus thermophilus HB8 (サーマス・サーモフィルス)株: ATCC 27634 / DSM 579 / HB8 |
| 分子量 | 理論値: 36.024688 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: MEDLALRPKT LDEYIGQERL KQKLRVYLEA AKARKEPLEH LLLFGPPGLG KTTLAHVIAH ELGVNLRVTS GPAIEKPGDL AAILANSLE EGDILFIDEI HRLSRQAEEH LYPAMEDFVM DIVIGQGPAA RTIRLELPRF TLIGATTRPG LITAPLLSRF G IVEHLEYY ...文字列: MEDLALRPKT LDEYIGQERL KQKLRVYLEA AKARKEPLEH LLLFGPPGLG KTTLAHVIAH ELGVNLRVTS GPAIEKPGDL AAILANSLE EGDILFIDEI HRLSRQAEEH LYPAMEDFVM DIVIGQGPAA RTIRLELPRF TLIGATTRPG LITAPLLSRF G IVEHLEYY TPEELAQGVM RDARLLGVRI TEEAALEIGR RSRGTMRVAK RLFRRVRDFA QVAGEEVITR ERALEALAAL GL DELGLEK RDREILEVLI LRFGGGPVGL ATLATALSED PGTLEEVHEP YLIRQGLLKR TPRGRVATEL AYRHLGYPPP VGP LLEP |
-分子 #2: 49-mer DNA
| 分子 | 名称: 49-mer DNA / タイプ: dna / ID: 2 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:    Thermus thermophilus HB8 (サーマス・サーモフィルス) Thermus thermophilus HB8 (サーマス・サーモフィルス) |
| 分子量 | 理論値: 15.146732 KDa |
| 配列 | 文字列: (DA)(DG)(DA)(DA)(DT)(DC)(DT)(DG)(DC)(DC) (DG)(DA)(DG)(DA)(DG)(DA)(DC)(DC)(DG)(DA) (DG)(DC)(DA)(DG)(DA)(DA)(DT)(DT)(DC) (DT)(DA)(DT)(DG)(DT)(DG)(DT)(DT)(DT)(DA) (DC) (DC)(DA)(DA)(DG)(DC)(DG)(DC)(DT) (DG) |
-分子 #3: 51-mer DNA
| 分子 | 名称: 51-mer DNA / タイプ: dna / ID: 3 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種:    Thermus thermophilus HB8 (サーマス・サーモフィルス) Thermus thermophilus HB8 (サーマス・サーモフィルス) |
| 分子量 | 理論値: 15.69107 KDa |
| 配列 | 文字列: (DC)(DA)(DG)(DC)(DG)(DC)(DT)(DT)(DG)(DG) (DT)(DA)(DA)(DA)(DC)(DA)(DC)(DA)(DT)(DA) (DG)(DA)(DA)(DT)(DT)(DC)(DT)(DG)(DC) (DT)(DC)(DT)(DC)(DG)(DG)(DT)(DC)(DT)(DG) (DA) (DG)(DC)(DC)(DG)(DT) ...文字列: (DC)(DA)(DG)(DC)(DG)(DC)(DT)(DT)(DG)(DG) (DT)(DA)(DA)(DA)(DC)(DA)(DC)(DA)(DT)(DA) (DG)(DA)(DA)(DT)(DT)(DC)(DT)(DG)(DC) (DT)(DC)(DT)(DC)(DG)(DG)(DT)(DC)(DT)(DG) (DA) (DG)(DC)(DC)(DG)(DT)(DC)(DT)(DA) (DA)(DG)(DA) |
-分子 #4: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER
| 分子 | 名称: PHOSPHOTHIOPHOSPHORIC ACID-ADENYLATE ESTER / タイプ: ligand / ID: 4 / コピー数: 3 / 式: AGS |
|---|---|
| 分子量 | 理論値: 523.247 Da |
| Chemical component information | 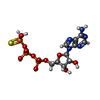 ChemComp-AGS: |
-分子 #5: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 5 / コピー数: 4 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #6: ADENOSINE-5'-DIPHOSPHATE
| 分子 | 名称: ADENOSINE-5'-DIPHOSPHATE / タイプ: ligand / ID: 6 / コピー数: 3 / 式: ADP |
|---|---|
| 分子量 | 理論値: 427.201 Da |
| Chemical component information | 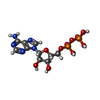 ChemComp-ADP: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.5 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm Bright-field microscopy / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.5 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 50.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




 Z
Z Y
Y X
X