+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 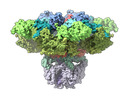 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Structure of the full-length IP3R1 channel determined at high Ca2+ | ||||||||||||||||||||||||
 マップデータ マップデータ | |||||||||||||||||||||||||
 試料 試料 |
| ||||||||||||||||||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Effects of PIP2 hydrolysis / Antigen activates B Cell Receptor (BCR) leading to generation of second messengers / inositol 1,4,5-trisphosphate receptor activity involved in regulation of postsynaptic cytosolic calcium levels / Elevation of cytosolic Ca2+ levels / cGMP effects / smooth endoplasmic reticulum membrane / platelet dense tubular network / negative regulation of calcium-mediated signaling /  calcineurin complex / platelet dense granule membrane ...Effects of PIP2 hydrolysis / Antigen activates B Cell Receptor (BCR) leading to generation of second messengers / inositol 1,4,5-trisphosphate receptor activity involved in regulation of postsynaptic cytosolic calcium levels / Elevation of cytosolic Ca2+ levels / cGMP effects / smooth endoplasmic reticulum membrane / platelet dense tubular network / negative regulation of calcium-mediated signaling / calcineurin complex / platelet dense granule membrane ...Effects of PIP2 hydrolysis / Antigen activates B Cell Receptor (BCR) leading to generation of second messengers / inositol 1,4,5-trisphosphate receptor activity involved in regulation of postsynaptic cytosolic calcium levels / Elevation of cytosolic Ca2+ levels / cGMP effects / smooth endoplasmic reticulum membrane / platelet dense tubular network / negative regulation of calcium-mediated signaling /  calcineurin complex / platelet dense granule membrane / epithelial fluid transport / ion channel modulating, G protein-coupled receptor signaling pathway / phospholipase C-activating G protein-coupled acetylcholine receptor signaling pathway / inositol 1,4,5-trisphosphate-gated calcium channel activity / regulation of postsynaptic cytosolic calcium ion concentration / voluntary musculoskeletal movement / calcineurin complex / platelet dense granule membrane / epithelial fluid transport / ion channel modulating, G protein-coupled receptor signaling pathway / phospholipase C-activating G protein-coupled acetylcholine receptor signaling pathway / inositol 1,4,5-trisphosphate-gated calcium channel activity / regulation of postsynaptic cytosolic calcium ion concentration / voluntary musculoskeletal movement /  inositol 1,4,5 trisphosphate binding / positive regulation of calcium ion transport / Glucagon-like Peptide-1 (GLP1) regulates insulin secretion / endoplasmic reticulum calcium ion homeostasis / positive regulation of hepatocyte proliferation / nuclear inner membrane / transport vesicle membrane / Ion homeostasis / dendrite development / intracellularly gated calcium channel activity / ligand-gated ion channel signaling pathway / intrinsic apoptotic signaling pathway in response to endoplasmic reticulum stress / GABA-ergic synapse / calcium channel inhibitor activity / cellular response to cAMP / release of sequestered calcium ion into cytosol / inositol 1,4,5 trisphosphate binding / positive regulation of calcium ion transport / Glucagon-like Peptide-1 (GLP1) regulates insulin secretion / endoplasmic reticulum calcium ion homeostasis / positive regulation of hepatocyte proliferation / nuclear inner membrane / transport vesicle membrane / Ion homeostasis / dendrite development / intracellularly gated calcium channel activity / ligand-gated ion channel signaling pathway / intrinsic apoptotic signaling pathway in response to endoplasmic reticulum stress / GABA-ergic synapse / calcium channel inhibitor activity / cellular response to cAMP / release of sequestered calcium ion into cytosol /  phosphatidylinositol binding / post-embryonic development / secretory granule membrane / phosphatidylinositol binding / post-embryonic development / secretory granule membrane /  筋小胞体 / 筋小胞体 /  synaptic membrane / synaptic membrane /  liver regeneration / calcium-mediated signaling / calcium ion transmembrane transport / Schaffer collateral - CA1 synapse / cell morphogenesis / positive regulation of insulin secretion / positive regulation of neuron projection development / calcium ion transport / presynapse / liver regeneration / calcium-mediated signaling / calcium ion transmembrane transport / Schaffer collateral - CA1 synapse / cell morphogenesis / positive regulation of insulin secretion / positive regulation of neuron projection development / calcium ion transport / presynapse /  核膜 / phospholipase C-activating G protein-coupled receptor signaling pathway / positive regulation of cytosolic calcium ion concentration / cellular response to hypoxia / postsynapse / 核膜 / phospholipase C-activating G protein-coupled receptor signaling pathway / positive regulation of cytosolic calcium ion concentration / cellular response to hypoxia / postsynapse /  protein phosphatase binding / transmembrane transporter binding / protein phosphatase binding / transmembrane transporter binding /  postsynaptic density / response to hypoxia / positive regulation of apoptotic process / protein domain specific binding / neuronal cell body / postsynaptic density / response to hypoxia / positive regulation of apoptotic process / protein domain specific binding / neuronal cell body /  樹状突起 / 樹状突起 /  シナプス / シナプス /  calcium ion binding / protein-containing complex binding / endoplasmic reticulum membrane / calcium ion binding / protein-containing complex binding / endoplasmic reticulum membrane /  核小体 / negative regulation of apoptotic process / perinuclear region of cytoplasm / 核小体 / negative regulation of apoptotic process / perinuclear region of cytoplasm /  小胞体 / protein-containing complex / 小胞体 / protein-containing complex /  生体膜 / identical protein binding / 生体膜 / identical protein binding /  細胞膜 / 細胞膜 /  細胞質 細胞質類似検索 - 分子機能 | ||||||||||||||||||||||||
| 生物種 |   Rattus norvegicus (ドブネズミ) / Rattus norvegicus (ドブネズミ) /   Norway rat (ドブネズミ) Norway rat (ドブネズミ) | ||||||||||||||||||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.26 Å クライオ電子顕微鏡法 / 解像度: 3.26 Å | ||||||||||||||||||||||||
 データ登録者 データ登録者 | Fan G / Baker MR / Terry LE / Arige V / Chen M / Seryshev AB / Baker ML / Ludtke SJ / Yule DI / Serysheva II | ||||||||||||||||||||||||
| 資金援助 |  米国, 7件 米国, 7件
| ||||||||||||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2022 ジャーナル: Nat Commun / 年: 2022タイトル: Conformational motions and ligand-binding underlying gating and regulation in IPR channel. 著者: Guizhen Fan / Mariah R Baker / Lara E Terry / Vikas Arige / Muyuan Chen / Alexander B Seryshev / Matthew L Baker / Steven J Ludtke / David I Yule / Irina I Serysheva /  要旨: Inositol-1,4,5-trisphosphate receptors (IPRs) are activated by IP and Ca and their gating is regulated by various intracellular messengers that finely tune the channel activity. Here, using single ...Inositol-1,4,5-trisphosphate receptors (IPRs) are activated by IP and Ca and their gating is regulated by various intracellular messengers that finely tune the channel activity. Here, using single particle cryo-EM analysis we determined 3D structures of the nanodisc-reconstituted IPR1 channel in two ligand-bound states. These structures provide unprecedented details governing binding of IP, Ca and ATP, revealing conformational changes that couple ligand-binding to channel opening. Using a deep-learning approach and 3D variability analysis we extracted molecular motions of the key protein domains from cryo-EM density data. We find that IP binding relies upon intrinsic flexibility of the ARM2 domain in the tetrameric channel. Our results highlight a key role of dynamic side chains in regulating gating behavior of IPR channels. This work represents a stepping-stone to developing mechanistic understanding of conformational pathways underlying ligand-binding, activation and regulation of the channel. | ||||||||||||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
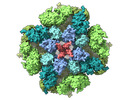
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27982.map.gz emd_27982.map.gz | 135.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27982-v30.xml emd-27982-v30.xml emd-27982.xml emd-27982.xml | 23.8 KB 23.8 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_27982.png emd_27982.png | 114.9 KB | ||
| その他 |  emd_27982_half_map_1.map.gz emd_27982_half_map_1.map.gz emd_27982_half_map_2.map.gz emd_27982_half_map_2.map.gz | 109.1 MB 109.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27982 http://ftp.pdbj.org/pub/emdb/structures/EMD-27982 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27982 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27982 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8eaqMC  8earC M: このマップから作成された原子モデル C: 同じ文献を引用 ( |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27982.map.gz / 形式: CCP4 / 大きさ: 144.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27982.map.gz / 形式: CCP4 / 大きさ: 144.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ボクセルのサイズ | X=Y=Z: 1.07 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: #2
| ファイル | emd_27982_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_27982_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : Type 1 inositol 1,4,5-trisphosphate receptor tetrameric protein c...
| 全体 | 名称: Type 1 inositol 1,4,5-trisphosphate receptor tetrameric protein complex |
|---|---|
| 要素 |
|
-超分子 #1: Type 1 inositol 1,4,5-trisphosphate receptor tetrameric protein c...
| 超分子 | 名称: Type 1 inositol 1,4,5-trisphosphate receptor tetrameric protein complex タイプ: complex / キメラ: Yes / ID: 1 / 親要素: 0 / 含まれる分子: #1 |
|---|---|
| 由来(天然) | 生物種:   Rattus norvegicus (ドブネズミ) / 器官: cerebellum / Organelle: endoplasmic reticulum / 細胞中の位置: membrane Rattus norvegicus (ドブネズミ) / 器官: cerebellum / Organelle: endoplasmic reticulum / 細胞中の位置: membrane |
| 分子量 | 理論値: 1.3 MDa |
-分子 #1: Inositol 1,4,5-trisphosphate receptor type 1
| 分子 | 名称: Inositol 1,4,5-trisphosphate receptor type 1 / タイプ: protein_or_peptide / ID: 1 / コピー数: 4 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Norway rat (ドブネズミ) / 器官: cerebellum Norway rat (ドブネズミ) / 器官: cerebellum |
| 分子量 | 理論値: 313.657406 KDa |
| 配列 | 文字列: MSDKMSSFLH IGDICSLYAE GSTNGFISTL GLVDDRCVVQ PEAGDLNNPP KKFRDCLFKL CPMNRYSAQK QFWKAAKPGA NSTTDAVLL NKLHHAADLE KKQNETENRK LLGTVIQYGN VIQLLHLKSN KYLTVNKRLP ALLEKNAMRV TLDEAGNEGS W FYIQPFYK ...文字列: MSDKMSSFLH IGDICSLYAE GSTNGFISTL GLVDDRCVVQ PEAGDLNNPP KKFRDCLFKL CPMNRYSAQK QFWKAAKPGA NSTTDAVLL NKLHHAADLE KKQNETENRK LLGTVIQYGN VIQLLHLKSN KYLTVNKRLP ALLEKNAMRV TLDEAGNEGS W FYIQPFYK LRSIGDSVVI GDKVVLNPVN AGQPLHASSH QLVDNPGCNE VNSVNCNTSW KIVLFMKWSD NKDDILKGGD VV RLFHAEQ EKFLTCDEHR KKQHVFLRTT GRQSATSATS SKALWEVEVV QHDPCRGGAG YWNSLFRFKH LATGHYLAAE VDP DFEEEC LEFQPSVDPD QDASRSRLRN AQEKMVYSLV SVPEGNDISS IFELDPTTLR GGDSLVPRNS YVRLRHLCTN TWVH STNIP IDKEEEKPVM LKIGTSPLKE DKEAFAIVPV SPAEVRDLDF ANDASKVLGS IAGKLEKGTI TQNERRSVTK LLEDL VYFV TGGTNSGQDV LEVVFSKPNR ERQKLMREQN ILKQIFKLLQ APFTDCGDGP MLRLEELGDQ RHAPFRHICR LCYRVL RHS QQDYRKNQEY IAKQFGFMQK QIGYDVLAED TITALLHNNR KLLEKHITAA EIDTFVSLVR KNREPRFLDY LSDLCVS MN KSIPVTQELI CKAVLNPTNA DILIETKLVL SRFEFEGVST GENALEAGED EEEVWLFWRD SNKEIRSKSV RELAQDAK E GQKEDRDVLS YYRYQLNLFA RMCLDRQYLA INEISGQLDV DLILRCMSDE NLPYDLRASF CRLMLHMHVD RDPQEQVTP VKYARLWSEI PSEIAIDDYD SSGASKDEIK ERFAQTMEFV EEYLRDVVCQ RFPFSDKEKN KLTFEVVNLA RNLIYFGFYN FSDLLRLTK ILLAILDCVH VTTIFPISKM TKGEENKGSN VMRSIHGVGE LMTQVVLRGG GFLPMTPMAA APEGNVKQAE P EKEDIMVM DTKLKIIEIL QFILNVRLDY RISCLLCIFK REFDESNSQS SETSSGNSSQ EGPSNVPGAL DFEHIEEQAE GI FGGSEEN TPLDLDDHGG RTFLRVLLHL TMHDYPPLVS GALQLLFRHF SQRQEVLQAF KQVQLLVTSQ DVDNYKQIKQ DLD QLRSIV EKSELWVYKG QGPDEPMDGA SGENEHKKTE EGTSKPLKHE STSSYNYRVV KEILIRLSKL CVQESASVRK SRKQ QQRLL RNMGAHAVVL ELLQIPYEKA EDTKMQEIMR LAHEFLQNFC AGNQQNQALL HKHINLFLNP GILEAVTMQH IFMNN FQLC SEINERVVQH FVHCIETHGR NVQYIKFLQT IVKAEGKFIK KCQDMVMAEL VNSGEDVLVF YNDRASFQTL IQMMRS ERD RMDENSPLFM YHIHLVELLA VCTEGKNVYT EIKCNSLLPL DDIVRVVTHE DCIPEVKIAY INFLNHCYVD TEVEMKE IY TSNHMWKLFE NFLVDICRAC NNTSDRKHAD SVLEKYVTEI VMSIVTTFFS SPFSDQSTTL QTRQPVFVQL LQGVFRVY H CNWLMPSQKA SVESCIRVLS DVAKSRAIAI PVDLDSQVNN LFLKSHNIVQ KTAMNWRLSA RNAARRDSVL AASRDYRNI IERLQDIVSA LEDRLRPLVQ AELSVLVDVL HRPELLFPEN TDARRKCESG GFICKLIKHT KQLLEENEEK LCIKVLQTLR EMMTKDRGY GEKQISIDEL ENAELPQPPE AENSTEQELE PSPPLRQLED HKRGEALRQI LVNRYYGNIR PSGRRESLTS F GNGPLSPG GPSKPGGGGG GPGSGSTSRG EMSLAEVQCH LDKEGASNLV IDLIMNASSD RVFHESILLA IALLEGGNTT IQ HSFFCRL TEDKKSEKFF KVFYDRMKVA QQEIKATVTV NTSDLGNKKK DDEVDRDAPS RKKAKEPTTQ ITEEVRDQLL EAS AATRKA FTTFRREADP DDHYQSGEGT QATTDKAKDD LEMSAVITIM QPILRFLQLL CENHNRDLQN FLRCQNNKTN YNLV CETLQ FLDCICGSTT GGLGLLGLYI NEKNVALINQ TLESLTEYCQ GPCHENQNCI ATHESNGIDI ITALILNDIN PLGKK RMDL VLELKNNASK LLLAIMESRH DSENAERILY NMRPKELVEV IKKAYMQGEV EFEDGENGED GAASPRNVGH NIYILA HQL ARHNKELQTM LKPGGQVDGD EALEFYAKHT AQIEIVRLDR TMEQIVFPVP SICEFLTKES KLRIYYTTER DEQGSKI ND FFLRSEDLFN EMNWQKKLRA QPVLYWCARN MSFWSSISFN LAVLMNLLVA FFYPFKGVRG GTLEPHWSGL LWTAMLIS L AIVIALPKPH GIRALIASTI LRLIFSVGLQ PTLFLLGAFN VCNKIIFLMS FVGNCGTFTR GYRAMVLDVE FLYHLLYLL ICAMGLFVHE FFYSLLLFDL VYREETLLNV IKSVTRNGRP IILTAALALI LVYLFSIVGY LFFKDDFILE VDRLPNETAG PETGESLAN DFLYSDVCRV ETGENCTSPA PKEELLPVEE TEQDKEHTCE TLLMCIVTVL SHGLRSGGGV GDVLRKPSKE E PLFAARVI YDLLFFFMVI IIVLNLIFGV IIDTFADLRS EKQKKEEILK TTCFICGLER DKFDNKTVTF EEHIKEEHNM WH YLCFIVL VKVKDSTEYT GPESYVAEMI RERNLDWFPR MRAMSLVSSD SEGEQNELRN LQEKLESTMK LVTNLSGQLS ELK DQMTEQ RKQKQRIGLL GHPPHMNVNP QQPA |
-分子 #2: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 2 / コピー数: 4 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-分子 #3: CALCIUM ION
| 分子 | 名称: CALCIUM ION / タイプ: ligand / ID: 3 / コピー数: 20 / 式: CA |
|---|---|
| 分子量 | 理論値: 40.078 Da |
-分子 #4: (9R,11S)-9-({[(1S)-1-HYDROXYHEXADECYL]OXY}METHYL)-2,2-DIMETHYL-5,...
| 分子 | 名称: (9R,11S)-9-({[(1S)-1-HYDROXYHEXADECYL]OXY}METHYL)-2,2-DIMETHYL-5,7,10-TRIOXA-2LAMBDA~5~-AZA-6LAMBDA~5~-PHOSPHAOCTACOSANE-6,6,11-TRIOL タイプ: ligand / ID: 4 / コピー数: 28 / 式: PLX |
|---|---|
| 分子量 | 理論値: 767.132 Da |
| Chemical component information |  ChemComp-PLX: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 濃度 | 1 mg/mL |
|---|---|
| 緩衝液 | pH: 7.4 |
| グリッド | モデル: Quantifoil R2/1 / 材質: COPPER / メッシュ: 200 / 支持フィルム - 材質: CARBON / 支持フィルム - トポロジー: CONTINUOUS / 前処理 - タイプ: GLOW DISCHARGE |
| 凍結 | 凍結剤: ETHANE / チャンバー内湿度: 100 % / チャンバー内温度: 277 K / 装置: FEI VITROBOT MARK IV |
| 詳細 | reconstituted in lipid nanodisc |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | TFS KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 倍率(補正後): 46943 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 130000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.5 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 130000 |
| 特殊光学系 | エネルギーフィルター - 名称: GIF Bioquantum / エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: GATAN K2 QUANTUM (4k x 4k) 検出モード: SUPER-RESOLUTION / デジタル化 - サイズ - 横: 3840 pixel / デジタル化 - サイズ - 縦: 3712 pixel / デジタル化 - サンプリング間隔: 5.0 µm / 実像数: 25125 / 平均露光時間: 0.2 sec. / 平均電子線量: 49.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 粒子像選択 | 選択した数: 1955320 / 詳細: NeuralNet autopicking in EMAN2 |
|---|---|
| CTF補正 | ソフトウェア - 名称: Gctf |
| 初期モデル | モデルのタイプ: EMDB MAP EMDB ID: 詳細: filtered to 60 angstroms |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: RELION (ver. 3) |
| 最終 3次元分類 | ソフトウェア - 名称: RELION (ver. 3) |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: RELION (ver. 3) |
| 最終 再構成 | 想定した対称性 - 点群: C4 (4回回転対称 ) / 解像度のタイプ: BY AUTHOR / 解像度: 3.26 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 3) / 使用した粒子像数: 346731 ) / 解像度のタイプ: BY AUTHOR / 解像度: 3.26 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: RELION (ver. 3) / 使用した粒子像数: 346731 |
 ムービー
ムービー コントローラー
コントローラー







 Z
Z Y
Y X
X



















