+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | SsoMCM hexamer bound to Mg/ADP-BeFx and 46-mer DNA strand. Class 1 | |||||||||
 マップデータ マップデータ | unsharpened map | |||||||||
 試料 試料 |
| |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報MCM complex / DNA duplex unwinding /  helicase activity / helicase activity /  single-stranded DNA binding / single-stranded DNA binding /  ヘリカーゼ / ヘリカーゼ /  DNA複製 / DNA複製 /  ATP hydrolysis activity / ATP hydrolysis activity /  ATP binding / identical protein binding ATP binding / identical protein binding類似検索 - 分子機能 | |||||||||
| 生物種 |   Saccharolobus solfataricus P2 (古細菌) / synthetic construct (人工物) Saccharolobus solfataricus P2 (古細菌) / synthetic construct (人工物) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 2.45 Å クライオ電子顕微鏡法 / 解像度: 2.45 Å | |||||||||
 データ登録者 データ登録者 | Meagher M / Myasnikov A / Enemark EJ | |||||||||
| 資金援助 |  米国, 2件 米国, 2件
| |||||||||
 引用 引用 |  ジャーナル: Int J Mol Sci / 年: 2022 ジャーナル: Int J Mol Sci / 年: 2022タイトル: Two Distinct Modes of DNA Binding by an MCM Helicase Enable DNA Translocation. 著者: Martin Meagher / Alexander Myasnikov / Eric J Enemark /  要旨: A six-subunit ATPase ring forms the central hub of the replication forks in all domains of life. This ring performs a helicase function to separate the two complementary DNA strands to be replicated ...A six-subunit ATPase ring forms the central hub of the replication forks in all domains of life. This ring performs a helicase function to separate the two complementary DNA strands to be replicated and drives the replication machinery along the DNA. Disruption of this helicase/ATPase ring is associated with genetic instability and diseases such as cancer. The helicase/ATPase rings of eukaryotes and archaea consist of six minichromosome maintenance (MCM) proteins. Prior structural studies have shown that MCM rings bind one encircled strand of DNA in a spiral staircase, suggesting that the ring pulls this strand of DNA through its central pore in a hand-over-hand mechanism where the subunit at the bottom of the staircase dissociates from DNA and re-binds DNA one step above the staircase. With high-resolution cryo-EM, we show that the MCM ring of the archaeal organism binds an encircled DNA strand in two different modes with different numbers of subunits engaged to DNA, illustrating a plausible mechanism for the alternating steps of DNA dissociation and re-association that occur during DNA translocation. | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_27978.map.gz emd_27978.map.gz | 31.6 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-27978-v30.xml emd-27978-v30.xml emd-27978.xml emd-27978.xml | 22.5 KB 22.5 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_27978_fsc.xml emd_27978_fsc.xml | 8.4 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_27978.png emd_27978.png | 122.7 KB | ||
| マスクデータ |  emd_27978_msk_1.map emd_27978_msk_1.map | 64 MB |  マスクマップ マスクマップ | |
| その他 |  emd_27978_additional_1.map.gz emd_27978_additional_1.map.gz emd_27978_half_map_1.map.gz emd_27978_half_map_1.map.gz emd_27978_half_map_2.map.gz emd_27978_half_map_2.map.gz | 59.6 MB 59.2 MB 59.2 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-27978 http://ftp.pdbj.org/pub/emdb/structures/EMD-27978 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27978 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-27978 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  8eajMC  8eafC  8eagC  8eahC  8eaiC  8eakC  8ealC  8eamC C: 同じ文献を引用 ( M: このマップから作成された原子モデル |
|---|---|
| 類似構造データ | 類似検索 - 機能・相同性  F&H 検索 F&H 検索 |
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_27978.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_27978.map.gz / 形式: CCP4 / 大きさ: 64 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | unsharpened map | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.08 Å | ||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_27978_msk_1.map emd_27978_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
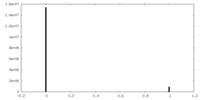
| 投影像・断面図 |
| ||||||||||||
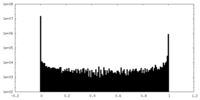
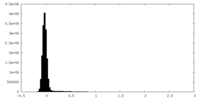
| 密度ヒストグラム |
-追加マップ: sharpened map
| ファイル | emd_27978_additional_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | sharpened map | ||||||||||||
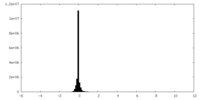
| 投影像・断面図 |
| ||||||||||||
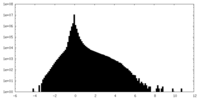
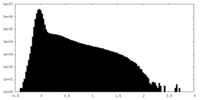
| 密度ヒストグラム |
-ハーフマップ: half-map A
| ファイル | emd_27978_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half-map A | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
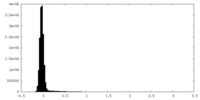
| 密度ヒストグラム |
-ハーフマップ: half-map B
| ファイル | emd_27978_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | half-map B | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
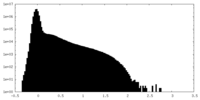
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : SsoMCM hexamer bound to Mg/ADP-BeFx and 46-mer DNA strand. Class 1.
| 全体 | 名称: SsoMCM hexamer bound to Mg/ADP-BeFx and 46-mer DNA strand. Class 1. |
|---|---|
| 要素 |
|
-超分子 #1: SsoMCM hexamer bound to Mg/ADP-BeFx and 46-mer DNA strand. Class 1.
| 超分子 | 名称: SsoMCM hexamer bound to Mg/ADP-BeFx and 46-mer DNA strand. Class 1. タイプ: complex / ID: 1 / キメラ: Yes / 親要素: 0 / 含まれる分子: #1-#2 |
|---|---|
| 由来(天然) | 生物種:   Saccharolobus solfataricus P2 (古細菌) Saccharolobus solfataricus P2 (古細菌) |
-分子 #1: Minichromosome maintenance protein MCM
| 分子 | 名称: Minichromosome maintenance protein MCM / タイプ: protein_or_peptide / ID: 1 / コピー数: 6 / 光学異性体: LEVO / EC番号:  ヘリカーゼ ヘリカーゼ |
|---|---|
| 由来(天然) | 生物種:   Saccharolobus solfataricus P2 (古細菌) / 株: ATCC 35092 / DSM 1617 / JCM 11322 / P2 Saccharolobus solfataricus P2 (古細菌) / 株: ATCC 35092 / DSM 1617 / JCM 11322 / P2 |
| 分子量 | 理論値: 68.641961 KDa |
| 組換発現 | 生物種:   Escherichia coli (大腸菌) Escherichia coli (大腸菌) |
| 配列 | 文字列: SLEIPSKQID YRDVFIEFLT TFKGNNNQNK YIERINELVA YRKKSLIIEF SDVLSFNENL AYEIINNTKI ILPILEGALY DHILQLDPT YQRDIEKVHV RIVGIPRVIE LRKIRSTDIG KLITIDGILV KVTPVKERIY KATYKHIHPD CMQEFEWPED E EMPEVLEM ...文字列: SLEIPSKQID YRDVFIEFLT TFKGNNNQNK YIERINELVA YRKKSLIIEF SDVLSFNENL AYEIINNTKI ILPILEGALY DHILQLDPT YQRDIEKVHV RIVGIPRVIE LRKIRSTDIG KLITIDGILV KVTPVKERIY KATYKHIHPD CMQEFEWPED E EMPEVLEM PTICPKCGKP GQFRLIPEKT KLIDWQKAVI QERPEEVPSG QLPRQLEIIL EDDLVDSARP GDRVKVTGIL DI KQDSPVK RGSRAVFDIY MKVSSIEVSG GSGGSSEEDE KKIKDLAKDP WIRDRIISSI APSIYGHWEL KEALALALFG GVP KVLEDT RIRGDIHILI IGDPGTAKSQ MLQFISRVAP RAVYTTGKGS TAAGLTAAVV REKGTGEYYL EAGALVLADG GIAV IDEID KMRDEDRVAI HEAMEQQTVS IAKAGIVAKL NARAAVIAAG NPKFGRYISE RPVSDNINLP PTILSRFDLI FILKD QPGE QDRELANYIL DVHSGKSTKN IIDIDTLRKY IAYARKYVTP KITSEAKNLI TDFFVEMRKK SSETPDSPIL ITPRQL EAL IRISEAYAKM ALKAEVTRED AERAINIMRL FLESVGVDME SGKIDID |
-分子 #2: 46-mer DNA strand
| 分子 | 名称: 46-mer DNA strand / タイプ: dna / ID: 2 / コピー数: 1 / 分類: DNA |
|---|---|
| 由来(天然) | 生物種: synthetic construct (人工物) |
| 分子量 | 理論値: 13.975942 KDa |
| 配列 | 文字列: (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DC)(DT)(DA)(DT)(DA)(DG)(DT)(DT)(DT) (DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT)(DT) (DT) (DT)(DT)(DT)(DT)(DT)(DT) |
-分子 #3: ZINC ION
| 分子 | 名称: ZINC ION / タイプ: ligand / ID: 3 / コピー数: 6 / 式: ZN |
|---|---|
| 分子量 | 理論値: 65.409 Da |
-分子 #4: MAGNESIUM ION
| 分子 | 名称: MAGNESIUM ION / タイプ: ligand / ID: 4 / コピー数: 5 / 式: MG |
|---|---|
| 分子量 | 理論値: 24.305 Da |
-分子 #5: [[[(2R,3S,4R,5R)-5-(6-aminopurin-9-yl)-3,4-bis(oxidanyl)oxolan-2-...
| 分子 | 名称: [[[(2R,3S,4R,5R)-5-(6-aminopurin-9-yl)-3,4-bis(oxidanyl)oxolan-2-yl]methoxy-oxidanyl-phosphoryl]oxy-oxidanyl-phosphoryl]oxy-tris(fluoranyl)beryllium タイプ: ligand / ID: 5 / コピー数: 5 / 式: 08T |
|---|---|
| 分子量 | 理論値: 492.201 Da |
| Chemical component information | 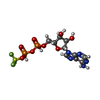 ChemComp-08T: |
-分子 #6: water
| 分子 | 名称: water / タイプ: ligand / ID: 6 / コピー数: 15 / 式: HOH |
|---|---|
| 分子量 | 理論値: 18.015 Da |
| Chemical component information |  ChemComp-HOH: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.6 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.8 µm Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 1.8 µm / 最小 デフォーカス(公称値): 0.8 µm |
| 撮影 | フィルム・検出器のモデル: GATAN K3 (6k x 4k) / 平均電子線量: 78.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
-原子モデル構築 1
| 初期モデル | PDB ID: Chain - Chain ID: B |
|---|---|
| 詳細 | Bond distance and angle restraints for a tetrahedral Zn2+ coordination were applied. Bond distance and angle restraints for a octahedral Mg2+ coordination were applied. |
| 精密化 | プロトコル: OTHER |
| 得られたモデル |  PDB-8eaj: |
 ムービー
ムービー コントローラー
コントローラー












 Z
Z Y
Y X
X