+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 |  | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Locally refined SARS-CoV-2 BA-2.86 Spike receptor binding domain (RBD) complexed with angiotensin converting enzyme 2 (ACE2) | |||||||||
 マップデータ マップデータ | Sharpened refined map of BA-2.86 spike with ACE2. | |||||||||
 試料 試料 |
| |||||||||
 キーワード キーワード |  Viral protein (ウイルスタンパク質) / Viral protein (ウイルスタンパク質) /  immune system (免疫系) / immune system (免疫系) /  SARS-CoV-2 (SARSコロナウイルス2) / SARS-CoV-2 (SARSコロナウイルス2) /  ACE2 (アンジオテンシン変換酵素2) / RBD / Spike / ACE2 (アンジオテンシン変換酵素2) / RBD / Spike /  glycoprotein (糖タンパク質) / BA.2.86 / glycoprotein (糖タンパク質) / BA.2.86 /  angiotensin converting enzyme 2 (アンジオテンシン変換酵素2) / angiotensin converting enzyme 2 (アンジオテンシン変換酵素2) /  receptor (受容体) / receptor (受容体) /  coronavirus-2 (オルトコロナウイルス亜科) coronavirus-2 (オルトコロナウイルス亜科) | |||||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of amino acid transport /  アンジオテンシン変換酵素2 / positive regulation of L-proline import across plasma membrane / アンジオテンシン変換酵素2 / positive regulation of L-proline import across plasma membrane /  加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; 金属プロテアーゼ / angiotensin-mediated drinking behavior / tryptophan transport / positive regulation of gap junction assembly / regulation of systemic arterial blood pressure by renin-angiotensin / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; 金属プロテアーゼ / angiotensin-mediated drinking behavior / tryptophan transport / positive regulation of gap junction assembly / regulation of systemic arterial blood pressure by renin-angiotensin /  regulation of vasoconstriction / regulation of cardiac conduction ...positive regulation of amino acid transport / regulation of vasoconstriction / regulation of cardiac conduction ...positive regulation of amino acid transport /  アンジオテンシン変換酵素2 / positive regulation of L-proline import across plasma membrane / アンジオテンシン変換酵素2 / positive regulation of L-proline import across plasma membrane /  加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; 金属プロテアーゼ / angiotensin-mediated drinking behavior / tryptophan transport / positive regulation of gap junction assembly / regulation of systemic arterial blood pressure by renin-angiotensin / 加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; 金属プロテアーゼ / angiotensin-mediated drinking behavior / tryptophan transport / positive regulation of gap junction assembly / regulation of systemic arterial blood pressure by renin-angiotensin /  regulation of vasoconstriction / regulation of cardiac conduction / peptidyl-dipeptidase activity / angiotensin maturation / maternal process involved in female pregnancy / Metabolism of Angiotensinogen to Angiotensins / regulation of vasoconstriction / regulation of cardiac conduction / peptidyl-dipeptidase activity / angiotensin maturation / maternal process involved in female pregnancy / Metabolism of Angiotensinogen to Angiotensins /  metallocarboxypeptidase activity / Attachment and Entry / metallocarboxypeptidase activity / Attachment and Entry /  carboxypeptidase activity / negative regulation of signaling receptor activity / positive regulation of cardiac muscle contraction / regulation of cytokine production / carboxypeptidase activity / negative regulation of signaling receptor activity / positive regulation of cardiac muscle contraction / regulation of cytokine production /  ウイルスのライフサイクル / blood vessel diameter maintenance / regulation of transmembrane transporter activity / brush border membrane / negative regulation of smooth muscle cell proliferation / ウイルスのライフサイクル / blood vessel diameter maintenance / regulation of transmembrane transporter activity / brush border membrane / negative regulation of smooth muscle cell proliferation /  繊毛 / negative regulation of ERK1 and ERK2 cascade / endocytic vesicle membrane / 繊毛 / negative regulation of ERK1 and ERK2 cascade / endocytic vesicle membrane /  metallopeptidase activity / positive regulation of reactive oxygen species metabolic process / virus receptor activity / regulation of cell population proliferation / metallopeptidase activity / positive regulation of reactive oxygen species metabolic process / virus receptor activity / regulation of cell population proliferation /  regulation of inflammatory response / Maturation of spike protein / viral translation / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space / regulation of inflammatory response / Maturation of spike protein / viral translation / Translation of Structural Proteins / Virion Assembly and Release / host cell surface / host extracellular space /  endopeptidase activity / suppression by virus of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / Potential therapeutics for SARS / entry receptor-mediated virion attachment to host cell / host cell endoplasmic reticulum-Golgi intermediate compartment membrane / receptor-mediated endocytosis of virus by host cell / Attachment and Entry / endopeptidase activity / suppression by virus of host tetherin activity / Induction of Cell-Cell Fusion / structural constituent of virion / Potential therapeutics for SARS / entry receptor-mediated virion attachment to host cell / host cell endoplasmic reticulum-Golgi intermediate compartment membrane / receptor-mediated endocytosis of virus by host cell / Attachment and Entry /  membrane fusion / positive regulation of viral entry into host cell / receptor-mediated virion attachment to host cell / membrane fusion / positive regulation of viral entry into host cell / receptor-mediated virion attachment to host cell /  receptor ligand activity / host cell surface receptor binding / symbiont entry into host cell / receptor ligand activity / host cell surface receptor binding / symbiont entry into host cell /  脂質ラフト / apical plasma membrane / fusion of virus membrane with host plasma membrane / 脂質ラフト / apical plasma membrane / fusion of virus membrane with host plasma membrane /  小胞体 / fusion of virus membrane with host endosome membrane / 小胞体 / fusion of virus membrane with host endosome membrane /  エンベロープ (ウイルス) / symbiont-mediated suppression of host type I interferon-mediated signaling pathway / virion attachment to host cell / SARS-CoV-2 activates/modulates innate and adaptive immune responses / host cell plasma membrane / virion membrane / エンベロープ (ウイルス) / symbiont-mediated suppression of host type I interferon-mediated signaling pathway / virion attachment to host cell / SARS-CoV-2 activates/modulates innate and adaptive immune responses / host cell plasma membrane / virion membrane /  細胞膜 / 細胞膜 /  extracellular space / extracellular exosome / zinc ion binding / extracellular region / extracellular space / extracellular exosome / zinc ion binding / extracellular region /  生体膜 / identical protein binding / 生体膜 / identical protein binding /  細胞膜 細胞膜類似検索 - 分子機能 | |||||||||
| 生物種 |   Homo sapiens (ヒト) / Homo sapiens (ヒト) /   Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) | |||||||||
| 手法 |  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.7 Å クライオ電子顕微鏡法 / 解像度: 3.7 Å | |||||||||
 データ登録者 データ登録者 | Ren J / Stuart DI / Duyvesteyn HME | |||||||||
| 資金援助 |  英国, 2件 英国, 2件
| |||||||||
 引用 引用 |  ジャーナル: To Be Published ジャーナル: To Be Publishedタイトル: The emerging BA.2.86 Omicron sub-lineage balances antibody escape and ACE2 affinity 著者: Stuart DI / Liu C #1:  ジャーナル: Acta Crystallogr., Sect. D: Biol. Crystallogr. ジャーナル: Acta Crystallogr., Sect. D: Biol. Crystallogr.年: 2019 タイトル: Macromolecular structure determination using X-rays, neutrons and electrons: recent developments in Phenix 著者: Liebschner D / Afonine PV #2:  ジャーナル: Nature Methods / 年: 2017 ジャーナル: Nature Methods / 年: 2017タイトル: cryoSPARC: algorithms for rapid unsupervised cryo-EM structure determination 著者: Punjani A / Rubinstein JL | |||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18639.map.gz emd_18639.map.gz | 79.1 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18639-v30.xml emd-18639-v30.xml emd-18639.xml emd-18639.xml | 22 KB 22 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| 画像 |  emd_18639.png emd_18639.png | 56.8 KB | ||
| Filedesc metadata |  emd-18639.cif.gz emd-18639.cif.gz | 7.1 KB | ||
| その他 |  emd_18639_half_map_1.map.gz emd_18639_half_map_1.map.gz emd_18639_half_map_2.map.gz emd_18639_half_map_2.map.gz | 77.7 MB 77.7 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18639 http://ftp.pdbj.org/pub/emdb/structures/EMD-18639 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18639 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18639 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|---|
| 「今月の分子」の関連する項目 |
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18639.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18639.map.gz / 形式: CCP4 / 大きさ: 83.7 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Sharpened refined map of BA-2.86 spike with ACE2. | ||||||||||||||||||||||||||||||||||||
| 投影像・断面図 | 画像のコントロール
画像は Spider により作成 | ||||||||||||||||||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.46 Å | ||||||||||||||||||||||||||||||||||||
| 密度 |
| ||||||||||||||||||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-ハーフマップ: Half map A of BA-2.86 spike with ACE2.
| ファイル | emd_18639_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map A of BA-2.86 spike with ACE2. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: Half map B of BA-2.86 spike with ACE2.
| ファイル | emd_18639_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Half map B of BA-2.86 spike with ACE2. | ||||||||||||
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : BA-2.86 variant SARS-CoV2 S protein complexed with angiotensin co...
| 全体 | 名称: BA-2.86 variant SARS-CoV2 S protein complexed with angiotensin converting enzyme 2 (ACE2) |
|---|---|
| 要素 |
|
-超分子 #1: BA-2.86 variant SARS-CoV2 S protein complexed with angiotensin co...
| 超分子 | 名称: BA-2.86 variant SARS-CoV2 S protein complexed with angiotensin converting enzyme 2 (ACE2) タイプ: complex / ID: 1 / 親要素: 0 / 含まれる分子: #1-#2 詳細: Spike protein recombinantly expressed using sequence of human-derived BA-2.86 variant of SARS-CoV2. Angiotensin converting enzyme 2 (ACE2) recombinantely expressed. |
|---|
-超分子 #2: Angiotensin converting enzyme 2 (ACE2)
| 超分子 | 名称: Angiotensin converting enzyme 2 (ACE2) / タイプ: complex / ID: 2 / 親要素: 1 / 含まれる分子: #2 詳細: Human Angiotensin converting enzyme 2 (hACE2) recombinantely expressed |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-超分子 #3: BA-2.86 variant SARS-CoV2 S protein
| 超分子 | 名称: BA-2.86 variant SARS-CoV2 S protein / タイプ: complex / ID: 3 / 親要素: 1 / 含まれる分子: #1 詳細: Spike protein recombinantly expressed using sequence of human-derived BA-2.86 variant of SARS-CoV2. |
|---|---|
| 由来(天然) | 生物種:   Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) |
-分子 #1: Spike protein S2'
| 分子 | 名称: Spike protein S2' / タイプ: protein_or_peptide / ID: 1 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) Severe acute respiratory syndrome coronavirus 2 (SARSコロナウイルス2) |
| 分子量 | 理論値: 22.159053 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: VTNLCPFHEV FNATRFASVY AWNRTRISNC VADYSVLYNF APFFAFKCYG VSPTKLNDLC FTNVYADSFV IKGNEVSQIA PGQTGNIAD YNYKLPDDFT GCVIAWNSNK LDSKHSGNYD YWYRLFRKSK LKPFERDIST EIYQAGNKPC KGKGPNCYFP L QSYGFRPT ...文字列: VTNLCPFHEV FNATRFASVY AWNRTRISNC VADYSVLYNF APFFAFKCYG VSPTKLNDLC FTNVYADSFV IKGNEVSQIA PGQTGNIAD YNYKLPDDFT GCVIAWNSNK LDSKHSGNYD YWYRLFRKSK LKPFERDIST EIYQAGNKPC KGKGPNCYFP L QSYGFRPT YGVGHQPYRV VVLSFELLHA PATVCGP UniProtKB:  スパイクタンパク質 スパイクタンパク質 |
-分子 #2: Processed angiotensin-converting enzyme 2
| 分子 | 名称: Processed angiotensin-converting enzyme 2 / タイプ: protein_or_peptide / ID: 2 / コピー数: 1 / 光学異性体: LEVO |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 分子量 | 理論値: 69.982562 KDa |
| 組換発現 | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
| 配列 | 文字列: STIEEQAKTF LDKFNHEAED LFYQSSLASW NYNTNITEEN VQNMNNAGDK WSAFLKEQST LAQMYPLQEI QNLTVKLQLQ ALQQNGSSV LSEDKSKRLN TILNTMSTIY STGKVCNPDN PQECLLLEPG LNEIMANSLD YNERLWAWES WRSEVGKQLR P LYEEYVVL ...文字列: STIEEQAKTF LDKFNHEAED LFYQSSLASW NYNTNITEEN VQNMNNAGDK WSAFLKEQST LAQMYPLQEI QNLTVKLQLQ ALQQNGSSV LSEDKSKRLN TILNTMSTIY STGKVCNPDN PQECLLLEPG LNEIMANSLD YNERLWAWES WRSEVGKQLR P LYEEYVVL KNEMARANHY EDYGDYWRGD YEVNGVDGYD YSRGQLIEDV EHTFEEIKPL YEHLHAYVRA KLMNAYPSYI SP IGCLPAH LLGDMWGRFW TNLYSLTVPF GQKPNIDVTD AMVDQAWDAQ RIFKEAEKFF VSVGLPNMTQ GFWENSMLTD PGN VQKAVC HPTAWDLGKG DFRILMCTKV TMDDFLTAHH EMGHIQYDMA YAAQPFLLRN GANEGFHEAV GEIMSLSAAT PKHL KSIGL LSPDFQEDNE TEINFLLKQA LTIVGTLPFT YMLEKWRWMV FKGEIPKDQW MKKWWEMKRE IVGVVEPVPH DETYC DPAS LFHVSNDYSF IRYYTRTLYQ FQFQEALCQA AKHEGPLHKC DISNSTEAGQ KLFNMLRLGK SEPWTLALEN VVGAKN MNV RPLLNYFEPL FTWLKDQNKN SFVGWSTDWS PYADHHHHHH UniProtKB:  アンジオテンシン変換酵素2 アンジオテンシン変換酵素2 |
-分子 #3: 2-acetamido-2-deoxy-beta-D-glucopyranose
| 分子 | 名称: 2-acetamido-2-deoxy-beta-D-glucopyranose / タイプ: ligand / ID: 3 / コピー数: 6 / 式: NAG |
|---|---|
| 分子量 | 理論値: 221.208 Da |
| Chemical component information | 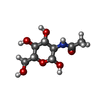 ChemComp-NAG: |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 |  単粒子再構成法 単粒子再構成法 |
| 試料の集合状態 | particle |
- 試料調製
試料調製
| 緩衝液 | pH: 7.4 |
|---|---|
| 凍結 | 凍結剤: ETHANE |
| 詳細 | BA-2.86 Spike with angiotensin converting enzyme 2 (ACE2) at a 6-fold excess of S protein. |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 50.0 µm / 照射モード: OTHER / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.6 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 165000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 2.6 µm / 最小 デフォーカス(公称値): 0.8 µm / 倍率(公称値): 165000 |
| 特殊光学系 | エネルギーフィルター - 名称: TFS Selectris X / エネルギーフィルター - スリット幅: 10 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) 撮影したグリッド数: 1 / 実像数: 9818 / 平均電子線量: 50.0 e/Å2 / 詳細: Images were collected in EER format. |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
- 画像解析
画像解析
| 粒子像選択 | 選択した数: 504319 |
|---|---|
| 初期モデル | モデルのタイプ: PDB ENTRY PDBモデル - PDB ID: 詳細: Initial RBD-Spike from related deposition. ACE2 model. Also compared with 8IOV. |
| 初期 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC |
| 最終 3次元分類 | クラス数: 5 / ソフトウェア - 名称: cryoSPARC (ver. v.4.3.1) 詳細: Following classification into 8 classes, classification without alignment, focussing on RBD/ACE2 region. |
| 最終 角度割当 | タイプ: MAXIMUM LIKELIHOOD / ソフトウェア - 名称: cryoSPARC (ver. v.4.3.1) |
| 最終 再構成 | 想定した対称性 - 点群: C1 (非対称) / 解像度のタイプ: BY AUTHOR / 解像度: 3.7 Å / 解像度の算出法: FSC 0.143 CUT-OFF / ソフトウェア - 名称: cryoSPARC (ver. v.4.3.1) / 詳細: CryoSPARC. / 使用した粒子像数: 15883 |
-原子モデル構築 1
| 初期モデル |
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 詳細 | Phenix real space refinement with manual checks using coot. | ||||||||
| 精密化 | 空間: REAL / プロトコル: OTHER | ||||||||
| 得られたモデル |  PDB-8qsq: |
 ムービー
ムービー コントローラー
コントローラー
















 Z (Sec.)
Z (Sec.) Y (Row.)
Y (Row.) X (Col.)
X (Col.)







































