+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | 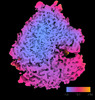 | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Human 80S ribosome structure from pFIB-lamellae reprocessed with TomoBEAR | |||||||||||||||
 マップデータ マップデータ | Focused refinement of LSU, filtered to local resolution, calibrated apix 1.9 | |||||||||||||||
 試料 試料 |
| |||||||||||||||
 キーワード キーワード | cryo-ET /  tomography (トモグラフィー) / StA / subtomogram averaging / tilt series / subnanometer resolution / pFIB / FIB-milling / plasma FIB / cell / tomography (トモグラフィー) / StA / subtomogram averaging / tilt series / subnanometer resolution / pFIB / FIB-milling / plasma FIB / cell /  RIBOSOME (リボソーム) RIBOSOME (リボソーム) | |||||||||||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | |||||||||||||||
| 手法 | サブトモグラム平均法 /  クライオ電子顕微鏡法 / 解像度: 6.2 Å クライオ電子顕微鏡法 / 解像度: 6.2 Å | |||||||||||||||
 データ登録者 データ登録者 | Balyschew N / Yushkevich A / Mikirtumov V / Sanchez RM / Sprink T / Kudryashev M | |||||||||||||||
| 資金援助 |  ドイツ, 4件 ドイツ, 4件
| |||||||||||||||
 引用 引用 |  ジャーナル: Nat Commun / 年: 2023 ジャーナル: Nat Commun / 年: 2023タイトル: Streamlined structure determination by cryo-electron tomography and subtomogram averaging using TomoBEAR. 著者: Nikita Balyschew / Artsemi Yushkevich / Vasilii Mikirtumov / Ricardo M Sanchez / Thiemo Sprink / Mikhail Kudryashev /  要旨: Structures of macromolecules in their native state provide unique unambiguous insights into their functions. Cryo-electron tomography combined with subtomogram averaging demonstrated the power to ...Structures of macromolecules in their native state provide unique unambiguous insights into their functions. Cryo-electron tomography combined with subtomogram averaging demonstrated the power to solve such structures in situ at resolutions in the range of 3 Angstrom for some macromolecules. In order to be applicable to the structural determination of the majority of macromolecules observable in cells in limited amounts, processing of tomographic data has to be performed in a high-throughput manner. Here we present TomoBEAR-a modular configurable workflow engine for streamlined processing of cryo-electron tomographic data for subtomogram averaging. TomoBEAR combines commonly used cryo-EM packages with reasonable presets to provide a transparent ("white box") approach for data management and processing. We demonstrate applications of TomoBEAR to two data sets of purified macromolecular targets, to an ion channel RyR1 in a membrane, and the tomograms of plasma FIB-milled lamellae and demonstrate the ability to produce high-resolution structures. TomoBEAR speeds up data processing, minimizes human interventions, and will help accelerate the adoption of in situ structural biology by cryo-ET. The source code and the documentation are freely available. #1:  ジャーナル: bioRxiv / 年: 2023 ジャーナル: bioRxiv / 年: 2023タイトル: Streamlined Structure Determination by Cryo-Electron Tomography and Subtomogram Averaging using TomoBEAR 著者: Balyschew N / Yushkevich A / Mikirtumov V / Sanchez RM / Sprink T / Kudryashev M | |||||||||||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 添付画像 |
|---|
- ダウンロードとリンク
ダウンロードとリンク
-EMDBアーカイブ
| マップデータ |  emd_18505.map.gz emd_18505.map.gz | 13.4 MB |  EMDBマップデータ形式 EMDBマップデータ形式 | |
|---|---|---|---|---|
| ヘッダ (付随情報) |  emd-18505-v30.xml emd-18505-v30.xml emd-18505.xml emd-18505.xml | 18.1 KB 18.1 KB | 表示 表示 |  EMDBヘッダ EMDBヘッダ |
| FSC (解像度算出) |  emd_18505_fsc.xml emd_18505_fsc.xml | 6.5 KB | 表示 |  FSCデータファイル FSCデータファイル |
| 画像 |  emd_18505.png emd_18505.png | 147.1 KB | ||
| マスクデータ |  emd_18505_msk_1.map emd_18505_msk_1.map | 22.2 MB |  マスクマップ マスクマップ | |
| Filedesc metadata |  emd-18505.cif.gz emd-18505.cif.gz | 4.8 KB | ||
| その他 |  emd_18505_half_map_1.map.gz emd_18505_half_map_1.map.gz emd_18505_half_map_2.map.gz emd_18505_half_map_2.map.gz | 11 MB 11 MB | ||
| アーカイブディレクトリ |  http://ftp.pdbj.org/pub/emdb/structures/EMD-18505 http://ftp.pdbj.org/pub/emdb/structures/EMD-18505 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18505 ftp://ftp.pdbj.org/pub/emdb/structures/EMD-18505 | HTTPS FTP |
-関連構造データ
- リンク
リンク
| EMDBのページ |  EMDB (EBI/PDBe) / EMDB (EBI/PDBe) /  EMDataResource EMDataResource |
|---|
- マップ
マップ
| ファイル |  ダウンロード / ファイル: emd_18505.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) ダウンロード / ファイル: emd_18505.map.gz / 形式: CCP4 / 大きさ: 22.2 MB / タイプ: IMAGE STORED AS FLOATING POINT NUMBER (4 BYTES) | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 注釈 | Focused refinement of LSU, filtered to local resolution, calibrated apix 1.9 | ||||||||||||||||||||
| ボクセルのサイズ | X=Y=Z: 1.9 Å | ||||||||||||||||||||
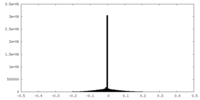
| 密度 |
| ||||||||||||||||||||
| 対称性 | 空間群: 1 | ||||||||||||||||||||
| 詳細 | EMDB XML:
|
-添付データ
-マスク #1
| ファイル |  emd_18505_msk_1.map emd_18505_msk_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
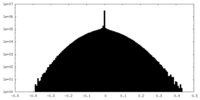
| 密度ヒストグラム |
-ハーフマップ: #1
| ファイル | emd_18505_half_map_1.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
-ハーフマップ: #2
| ファイル | emd_18505_half_map_2.map | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 投影像・断面図 |
| ||||||||||||
| 密度ヒストグラム |
- 試料の構成要素
試料の構成要素
-全体 : 80S human ribosome
| 全体 | 名称: 80S human ribosome |
|---|---|
| 要素 |
|
-超分子 #1: 80S human ribosome
| 超分子 | 名称: 80S human ribosome / タイプ: cell / ID: 1 / 親要素: 0 |
|---|---|
| 由来(天然) | 生物種:   Homo sapiens (ヒト) Homo sapiens (ヒト) |
-実験情報
-構造解析
| 手法 |  クライオ電子顕微鏡法 クライオ電子顕微鏡法 |
|---|---|
 解析 解析 | サブトモグラム平均法 |
| 試料の集合状態 | cell |
- 試料調製
試料調製
| 緩衝液 | pH: 5.75 |
|---|---|
| グリッド | モデル: UltrAuFoil R2/2 / 材質: GOLD / メッシュ: 200 / 支持フィルム - 材質: GOLD / 支持フィルム - トポロジー: HOLEY ARRAY |
| 凍結 | 凍結剤: ETHANE / 装置: FEI VITROBOT MARK IV 詳細: 24 h post infection cells were plunge-frozen using the Vitrobot (Thermo Fisher) offsetting the blotting pad to favour back blotting. Just prior the PF, the cells media has been replaced with ...詳細: 24 h post infection cells were plunge-frozen using the Vitrobot (Thermo Fisher) offsetting the blotting pad to favour back blotting. Just prior the PF, the cells media has been replaced with the complete media with 10% glycerol (v/v)... |
- 電子顕微鏡法
電子顕微鏡法
| 顕微鏡 | FEI TITAN KRIOS |
|---|---|
| 電子線 | 加速電圧: 300 kV / 電子線源:  FIELD EMISSION GUN FIELD EMISSION GUN |
| 電子光学系 | C2レンズ絞り径: 100.0 µm / 最大 デフォーカス(補正後): 5.0 µm / 最小 デフォーカス(補正後): 2.5 µm / 倍率(補正後): 64000 / 照射モード: FLOOD BEAM / 撮影モード: BRIGHT FIELD Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 2.5 µm / 倍率(公称値): 64000 Bright-field microscopy / Cs: 2.7 mm / 最大 デフォーカス(公称値): 5.0 µm / 最小 デフォーカス(公称値): 2.5 µm / 倍率(公称値): 64000 |
| 特殊光学系 | エネルギーフィルター - 名称: TFS Selectris / エネルギーフィルター - スリット幅: 20 eV |
| 試料ステージ | 試料ホルダーモデル: FEI TITAN KRIOS AUTOGRID HOLDER ホルダー冷却材: NITROGEN |
| 撮影 | フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) デジタル化 - サイズ - 横: 4096 pixel / デジタル化 - サイズ - 縦: 4096 pixel / 平均電子線量: 5.0 e/Å2 |
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
 ムービー
ムービー コントローラー
コントローラー




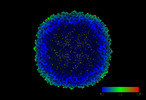


 Z
Z Y
Y X
X