+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 8f39 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Yeast ATP synthase in conformation-2, at pH 6 | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード |  MEMBRANE PROTEIN (膜タンパク質) / F-type ATP synthase / MEMBRANE PROTEIN (膜タンパク質) / F-type ATP synthase /  yeast (酵母) / yeast (酵母) /  mitochondrial (ミトコンドリア) mitochondrial (ミトコンドリア) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報cristae formation / mitochondrial proton-transporting ATP synthase, central stalk / Formation of ATP by chemiosmotic coupling / Cristae formation /  Mitochondrial protein degradation / mitochondrial proton-transporting ATP synthase complex assembly / mitochondrial proton-transporting ATP synthase, stator stalk / mitochondrial proton-transporting ATP synthase, catalytic core / mitochondrial proton-transporting ATP synthase complex, coupling factor F(o) / mitochondrial proton-transporting ATP synthase complex ...cristae formation / mitochondrial proton-transporting ATP synthase, central stalk / Formation of ATP by chemiosmotic coupling / Cristae formation / Mitochondrial protein degradation / mitochondrial proton-transporting ATP synthase complex assembly / mitochondrial proton-transporting ATP synthase, stator stalk / mitochondrial proton-transporting ATP synthase, catalytic core / mitochondrial proton-transporting ATP synthase complex, coupling factor F(o) / mitochondrial proton-transporting ATP synthase complex ...cristae formation / mitochondrial proton-transporting ATP synthase, central stalk / Formation of ATP by chemiosmotic coupling / Cristae formation /  Mitochondrial protein degradation / mitochondrial proton-transporting ATP synthase complex assembly / mitochondrial proton-transporting ATP synthase, stator stalk / mitochondrial proton-transporting ATP synthase, catalytic core / mitochondrial proton-transporting ATP synthase complex, coupling factor F(o) / mitochondrial proton-transporting ATP synthase complex / mitochondrial proton-transporting ATP synthase complex, catalytic sector F(1) / mitochondrial nucleoid / proton motive force-driven mitochondrial ATP synthesis / proton motive force-driven ATP synthesis / proton transmembrane transporter activity / proton-transporting ATP synthase complex, catalytic core F(1) / Mitochondrial protein degradation / mitochondrial proton-transporting ATP synthase complex assembly / mitochondrial proton-transporting ATP synthase, stator stalk / mitochondrial proton-transporting ATP synthase, catalytic core / mitochondrial proton-transporting ATP synthase complex, coupling factor F(o) / mitochondrial proton-transporting ATP synthase complex / mitochondrial proton-transporting ATP synthase complex, catalytic sector F(1) / mitochondrial nucleoid / proton motive force-driven mitochondrial ATP synthesis / proton motive force-driven ATP synthesis / proton transmembrane transporter activity / proton-transporting ATP synthase complex, catalytic core F(1) /  ATP合成酵素 / proton transmembrane transport / proton-transporting ATPase activity, rotational mechanism / proton-transporting ATP synthase activity, rotational mechanism / ATP合成酵素 / proton transmembrane transport / proton-transporting ATPase activity, rotational mechanism / proton-transporting ATP synthase activity, rotational mechanism /  ADP binding / ADP binding /  ミトコンドリア / protein-containing complex assembly / ミトコンドリア / protein-containing complex assembly /  ミトコンドリア内膜 / ミトコンドリア内膜 /  lipid binding / lipid binding /  ATP hydrolysis activity / ATP hydrolysis activity /  ミトコンドリア / ミトコンドリア /  ATP binding / identical protein binding / ATP binding / identical protein binding /  細胞質基質 細胞質基質類似検索 - 分子機能 | ||||||
| 生物種 |   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) | ||||||
| 手法 |  電子顕微鏡法 / 電子顕微鏡法 /  単粒子再構成法 / 単粒子再構成法 /  クライオ電子顕微鏡法 / 解像度: 3.5 Å クライオ電子顕微鏡法 / 解像度: 3.5 Å | ||||||
 データ登録者 データ登録者 | Sharma, S. / Patel, H. / Luo, M. / Mueller, D.M. / Liao, M. | ||||||
| 資金援助 |  米国, 1件 米国, 1件
| ||||||
 引用 引用 |  ジャーナル: Nat Struct Mol Biol / 年: 2024 ジャーナル: Nat Struct Mol Biol / 年: 2024タイトル: Conformational ensemble of yeast ATP synthase at low pH reveals unique intermediates and plasticity in F-F coupling. 著者: Stuti Sharma / Min Luo / Hiral Patel / David M Mueller / Maofu Liao /    要旨: Mitochondrial adenosine triphosphate (ATP) synthase uses the proton gradient across the inner mitochondrial membrane to synthesize ATP. Structural and single molecule studies conducted mostly at ...Mitochondrial adenosine triphosphate (ATP) synthase uses the proton gradient across the inner mitochondrial membrane to synthesize ATP. Structural and single molecule studies conducted mostly at neutral or basic pH have provided details of the reaction mechanism of ATP synthesis. However, pH of the mitochondrial matrix is slightly acidic during hypoxia and pH-dependent conformational changes in the ATP synthase have been reported. Here we use single-particle cryo-EM to analyze the conformational ensemble of the yeast (Saccharomyces cerevisiae) ATP synthase at pH 6. Of the four conformations resolved in this study, three are reaction intermediates. In addition to canonical catalytic dwell and binding dwell structures, we identify two unique conformations with nearly identical positions of the central rotor but different catalytic site conformations. These structures provide new insights into the catalytic mechanism of the ATP synthase and highlight elastic coupling between the catalytic and proton translocating domains. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  8f39.cif.gz 8f39.cif.gz | 849.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb8f39.ent.gz pdb8f39.ent.gz | 表示 |  PDB形式 PDB形式 | |
| PDBx/mmJSON形式 |  8f39.json.gz 8f39.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/f3/8f39 https://data.pdbj.org/pub/pdb/validation_reports/f3/8f39 ftp://data.pdbj.org/pub/pdb/validation_reports/f3/8f39 ftp://data.pdbj.org/pub/pdb/validation_reports/f3/8f39 | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-ATP synthase subunit ... , 13種, 26分子 GZ76USTKLMNOPQRXJYABCDEFHI
| #1: タンパク質 |  ATP合成酵素 ATP合成酵素分子量: 29042.393 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P38077 Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P38077 | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| #2: タンパク質 |  ATP合成酵素 ATP合成酵素分子量: 17575.994 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P05626 Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P05626 | ||||||||||||||
| #3: タンパク質 |  ATP合成酵素 ATP合成酵素分子量: 19509.188 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P30902 Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P30902 | ||||||||||||||
| #4: タンパク質 |  ATP合成酵素 ATP合成酵素分子量: 10090.904 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: Q12349 Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: Q12349 | ||||||||||||||
| #5: タンパク質 |  ATP合成酵素 ATP合成酵素分子量: 9383.837 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: Q06405 Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: Q06405 | ||||||||||||||
| #6: タンパク質 |  ATP合成酵素 / Lipid-binding protein / Oligomycin resistance protein 1 ATP合成酵素 / Lipid-binding protein / Oligomycin resistance protein 1分子量: 7663.243 Da / 分子数: 10 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P61829 Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P61829#8: タンパク質 | |  ATP合成酵素 / F-ATPase protein 6 ATP合成酵素 / F-ATPase protein 6分子量: 25073.230 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P00854 Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P00854#9: タンパク質・ペプチド | |  ATP合成酵素 / ATPase synthase I subunit ATP合成酵素 / ATPase synthase I subunit分子量: 4145.884 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P81450 Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P81450#10: タンパク質 | |  ATP合成酵素 / ATP synthase chain 5 / Oligomycin sensitivity conferral protein / OSCP ATP合成酵素 / ATP synthase chain 5 / Oligomycin sensitivity conferral protein / OSCP分子量: 17826.584 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P09457 Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P09457#11: タンパク質 |  ATP合成酵素 ATP合成酵素分子量: 54748.148 Da / 分子数: 3 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P07251 Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P07251#12: タンパク質 |  ATP合成酵素 ATP合成酵素分子量: 50752.641 Da / 分子数: 3 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P00830, Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P00830,  ATP合成酵素 ATP合成酵素#13: タンパク質 | |  ATP合成酵素 / F-ATPase delta subunit ATP合成酵素 / F-ATPase delta subunit分子量: 14080.876 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: Q12165 Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: Q12165#14: タンパク質 | |  ATP合成酵素 / ATPase subunit epsilon ATP合成酵素 / ATPase subunit epsilon分子量: 6388.076 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P21306 Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P21306 |
-タンパク質・ペプチド , 1種, 1分子 8
| #7: タンパク質・ペプチド |  ATP合成酵素 ATP合成酵素分子量: 5046.215 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)   Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P00856 Saccharomyces cerevisiae (パン酵母) / 参照: UniProt: P00856 |
|---|
-非ポリマー , 2種, 9分子 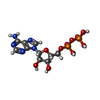


| #15: 化合物 | ChemComp-ADP /  アデノシン二リン酸 アデノシン二リン酸#16: 化合物 | ChemComp-MG / |
|---|
-詳細
| 研究の焦点であるリガンドがあるか | Y |
|---|
-実験情報
-実験
| 実験 | 手法:  電子顕微鏡法 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法:  単粒子再構成法 単粒子再構成法 |
- 試料調製
試料調製
| 構成要素 | 名称: ATP synthase ATP合成酵素 / タイプ: COMPLEX / Entity ID: #1-#14 / 由来: NATURAL ATP合成酵素 / タイプ: COMPLEX / Entity ID: #1-#14 / 由来: NATURAL |
|---|---|
| 由来(天然) | 生物種:   Saccharomyces cerevisiae (パン酵母) Saccharomyces cerevisiae (パン酵母) |
| 緩衝液 | pH: 6 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色 : NO / 凍結 : NO / 凍結 : YES : YES |
急速凍結 | 凍結剤: ETHANE |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Titan Krios / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI TITAN KRIOS |
| 電子銃 | 電子線源 : :  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD Bright-field microscopy / 最大 デフォーカス(公称値): 1500 nm / 最小 デフォーカス(公称値): 700 nm Bright-field microscopy / 最大 デフォーカス(公称値): 1500 nm / 最小 デフォーカス(公称値): 700 nm |
| 撮影 | 電子線照射量: 52 e/Å2 フィルム・検出器のモデル: FEI FALCON IV (4k x 4k) |
- 解析
解析
| ソフトウェア | 名称: PHENIX / バージョン: 1.20.1_4487: / 分類: 精密化 | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
CTF補正 | タイプ: PHASE FLIPPING AND AMPLITUDE CORRECTION | ||||||||||||||||||||||||
3次元再構成 | 解像度: 3.5 Å / 解像度の算出法: FSC 0.143 CUT-OFF / 粒子像の数: 99780 / 対称性のタイプ: POINT | ||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー


















 PDBj
PDBj




