+検索条件
-Structure paper
| タイトル | Quantifying how single dose Ad26.COV2.S vaccine efficacy depends on Spike sequence features. |
|---|---|
| ジャーナル・号・ページ | Nat Commun, Vol. 15, Issue 1, Page 2175, Year 2024 |
| 掲載日 | 2024年3月11日 |
 著者 著者 | Craig A Magaret / Li Li / Allan C deCamp / Morgane Rolland / Michal Juraska / Brian D Williamson / James Ludwig / Cindy Molitor / David Benkeser / Alex Luedtke / Brian Simpkins / Fei Heng / Yanqing Sun / Lindsay N Carpp / Hongjun Bai / Bethany L Dearlove / Elena E Giorgi / Mandy Jongeneelen / Boerries Brandenburg / Matthew McCallum / John E Bowen / David Veesler / Jerald Sadoff / Glenda E Gray / Sanne Roels / An Vandebosch / Daniel J Stieh / Mathieu Le Gars / Johan Vingerhoets / Beatriz Grinsztejn / Paul A Goepfert / Leonardo Paiva de Sousa / Mayara Secco Torres Silva / Martin Casapia / Marcelo H Losso / Susan J Little / Aditya Gaur / Linda-Gail Bekker / Nigel Garrett / Carla Truyers / Ilse Van Dromme / Edith Swann / Mary A Marovich / Dean Follmann / Kathleen M Neuzil / Lawrence Corey / Alexander L Greninger / Pavitra Roychoudhury / Ollivier Hyrien / Peter B Gilbert /        |
| PubMed 要旨 | In the ENSEMBLE randomized, placebo-controlled phase 3 trial (NCT04505722), estimated single-dose Ad26.COV2.S vaccine efficacy (VE) was 56% against moderate to severe-critical COVID-19. SARS-CoV-2 ...In the ENSEMBLE randomized, placebo-controlled phase 3 trial (NCT04505722), estimated single-dose Ad26.COV2.S vaccine efficacy (VE) was 56% against moderate to severe-critical COVID-19. SARS-CoV-2 Spike sequences were determined from 484 vaccine and 1,067 placebo recipients who acquired COVID-19. In this set of prespecified analyses, we show that in Latin America, VE was significantly lower against Lambda vs. Reference and against Lambda vs. non-Lambda [family-wise error rate (FWER) p < 0.05]. VE differed by residue match vs. mismatch to the vaccine-insert at 16 amino acid positions (4 FWER p < 0.05; 12 q-value ≤ 0.20); significantly decreased with physicochemical-weighted Hamming distance to the vaccine-strain sequence for Spike, receptor-binding domain, N-terminal domain, and S1 (FWER p < 0.001); differed (FWER ≤ 0.05) by distance to the vaccine strain measured by 9 antibody-epitope escape scores and 4 NTD neutralization-impacting features; and decreased (p = 0.011) with neutralization resistance level to vaccinee sera. VE against severe-critical COVID-19 was stable across most sequence features but lower against the most distant viruses. |
 リンク リンク |  Nat Commun / Nat Commun /  PubMed:38467646 / PubMed:38467646 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) |
| 解像度 | 2.7 - 3.2 Å |
| 構造データ | EMDB-43658, PDB-8vye: EMDB-43659, PDB-8vyf: EMDB-43660, PDB-8vyg: |
| 化合物 | 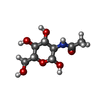 ChemComp-NAG: |
| 由来 |
|
 キーワード キーワード |  VIRAL PROTEIN/IMMUNE SYSTEM (ウイルス性) / VIRAL PROTEIN/IMMUNE SYSTEM (ウイルス性) /  sars-cov-2 (SARSコロナウイルス2) / sars-cov-2 (SARSコロナウイルス2) /  coronavirus (オルトコロナウイルス亜科) / coronavirus (オルトコロナウイルス亜科) /  lambda (Λ) / s309 / s2l20 / s2x303 / C.37 / lambda (Λ) / s309 / s2l20 / s2x303 / C.37 /  antibody (抗体) / antibody (抗体) /  Fab / Fab /  S protein / spike / S protein / spike /  fusion protein (融合タンパク質) / fusion protein (融合タンパク質) /  Structural Genomics (構造ゲノミクス) / Seattle Structural Genomics Center for Infectious Disease / SSGCID / Structural Genomics (構造ゲノミクス) / Seattle Structural Genomics Center for Infectious Disease / SSGCID /  VIRAL PROTEIN (ウイルスタンパク質) / VIRAL PROTEIN (ウイルスタンパク質) /  VIRAL PROTEIN-IMMUNE SYSTEM complex (ウイルス性) VIRAL PROTEIN-IMMUNE SYSTEM complex (ウイルス性) |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について