+検索条件
-Structure paper
| タイトル | Structural and functional insights into Escherichia coli α2-macroglobulin endopeptidase snap-trap inhibition. |
|---|---|
| ジャーナル・号・ページ | Proc Natl Acad Sci U S A, Vol. 112, Issue 27, Page 8290-8295, Year 2015 |
| 掲載日 | 2015年7月7日 |
 著者 著者 | Irene Garcia-Ferrer / Pedro Arêde / Josué Gómez-Blanco / Daniel Luque / Stephane Duquerroy / José R Castón / Theodoros Goulas / F Xavier Gomis-Rüth /   |
| PubMed 要旨 | The survival of commensal bacteria requires them to evade host peptidases. Gram-negative bacteria from the human gut microbiome encode a relative of the human endopeptidase inhibitor, α2- ...The survival of commensal bacteria requires them to evade host peptidases. Gram-negative bacteria from the human gut microbiome encode a relative of the human endopeptidase inhibitor, α2-macroglobulin (α2M). Escherichia coli α2M (ECAM) is a ∼ 180-kDa multidomain membrane-anchored pan-peptidase inhibitor, which is cleaved by host endopeptidases in an accessible bait region. Structural studies by electron microscopy and crystallography reveal that this cleavage causes major structural rearrangement of more than half the 13-domain structure from a native to a compact induced form. It also exposes a reactive thioester bond, which covalently traps the peptidase. Subsequently, peptidase-laden ECAM is shed from the membrane and may dimerize. Trapped peptidases are still active except against very large substrates, so inhibition potentially prevents damage of large cell envelope components, but not host digestion. Mechanistically, these results document a novel monomeric "snap trap." |
 リンク リンク |  Proc Natl Acad Sci U S A / Proc Natl Acad Sci U S A /  PubMed:26100869 / PubMed:26100869 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) / X線回折 |
| 解像度 | 1.6 - 17.0 Å |
| 構造データ | EMDB-3016, PDB-5a42:  EMDB-3017:  EMDB-3018:  PDB-4ziq:  PDB-4ziu:  PDB-4zjg:  PDB-4zjh: |
| 化合物 |  ChemComp-GOL:  ChemComp-CL:  ChemComp-HOH:  ChemComp-NI: 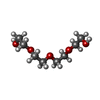 ChemComp-PG4: 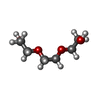 ChemComp-PGE:  ChemComp-1PE:  ChemComp-PEG: 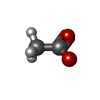 ChemComp-ACT: |
| 由来 |
|
 キーワード キーワード |  MEMBRANE PROTEIN (膜タンパク質) / Bacterial pan-proteinase inhibitor / MEMBRANE PROTEIN (膜タンパク質) / Bacterial pan-proteinase inhibitor /  MEMBRANE PROTEIN/INHIBITOR (生体膜) / MEMBRANE PROTEIN/INHIBITOR (生体膜) /  membrane protein and inhibitor complex (生体膜) / membrane protein and inhibitor complex (生体膜) /  MEMBRANE PROTEIN-INHIBITOR complex (生体膜) / HYDROLASE INHIBITOR / MEMBRANE PROTEIN-INHIBITOR complex (生体膜) / HYDROLASE INHIBITOR /  PEPTIDASE INHIBITOR (プロテアーゼ阻害剤 (生物学)) PEPTIDASE INHIBITOR (プロテアーゼ阻害剤 (生物学)) |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について






