+検索条件
-Structure paper
| タイトル | Dissecting the conformational complexity and mechanism of a bacterial heme transporter. |
|---|---|
| ジャーナル・号・ページ | Nat Chem Biol, Vol. 19, Issue 8, Page 992-991003, Year 2023 |
| 掲載日 | 2023年4月24日 |
 著者 著者 | Di Wu / Ahmad R Mehdipour / Franziska Finke / Hojjat G Goojani / Roan R Groh / Tamara N Grund / Thomas M B Reichhart / Rita Zimmermann / Sonja Welsch / Dirk Bald / Mark Shepherd / Gerhard Hummer / Schara Safarian /      |
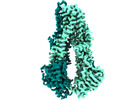
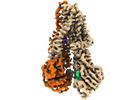
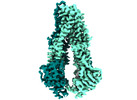
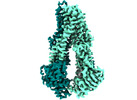
| PubMed 要旨 | Iron-bound cyclic tetrapyrroles (hemes) are redox-active cofactors in bioenergetic enzymes. However, the mechanisms of heme transport and insertion into respiratory chain complexes remain unclear. ...Iron-bound cyclic tetrapyrroles (hemes) are redox-active cofactors in bioenergetic enzymes. However, the mechanisms of heme transport and insertion into respiratory chain complexes remain unclear. Here, we used cellular, biochemical, structural and computational methods to characterize the structure and function of the heterodimeric bacterial ABC transporter CydDC. We provide multi-level evidence that CydDC is a heme transporter required for functional maturation of cytochrome bd, a pharmaceutically relevant drug target. Our systematic single-particle cryogenic-electron microscopy approach combined with atomistic molecular dynamics simulations provides detailed insight into the conformational landscape of CydDC during substrate binding and occlusion. Our simulations reveal that heme binds laterally from the membrane space to the transmembrane region of CydDC, enabled by a highly asymmetrical inward-facing CydDC conformation. During the binding process, heme propionates interact with positively charged residues on the surface and later in the substrate-binding pocket of the transporter, causing the heme orientation to rotate 180°. |
 リンク リンク |  Nat Chem Biol / Nat Chem Biol /  PubMed:37095238 / PubMed:37095238 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) |
| 解像度 | 2.71 - 3.89 Å |
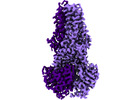
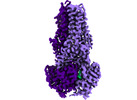
| 構造データ | EMDB-14636, PDB-7zd5:  EMDB-14638: IF(heme/confined) conformation of CydDC (Dataset-1) EMDB-14639, PDB-7zda: EMDB-14640, PDB-7zdb:  EMDB-14641: IF(apo/as isolated) conformation of CydDC in ADP(CydD) bound state (Dataset-3) EMDB-14642, PDB-7zdc: EMDB-14643, PDB-7zde: EMDB-14644, PDB-7zdf: EMDB-14645, PDB-7zdg:  EMDB-14646: IF(heme/confined) conformation of CydDC in AMP-PNP(CydD) bound state (Dataset-6)  EMDB-14647: IF(apo/asym) conformation of CydDC (Dataset-7) 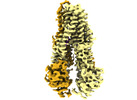 EMDB-14649: IF(heme/coordinated) conformation of CydDC (Dataset-7) EMDB-14652, PDB-7zdk: EMDB-14653, PDB-7zdl:  EMDB-14654: IF(heme/coordinated) conformation of CydDC in AMP-PNP(CydC)/AMP-PNP(CydD) bound state (Dataset-9)  EMDB-14655: IF(apo/as isolated) conformation of CydDC in AMP-PNP(CydD) bound state (Dataset-10) 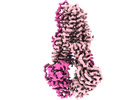 EMDB-14656: IF(heme/confined) conformation of CydDC in AMP-PNP(CydD) bound state (Dataset-10)  EMDB-14657: IF(apo/as isolated) conformation of CydDC in AMP-PNP(CydD) bound state (Dataset-11) 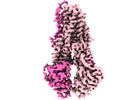 EMDB-14659: IF(heme/confined) conformation of CydDC in AMP-PNP(CydD) bound state (Dataset-12)  EMDB-14660: IF(apo/asym) conformation of CydDC in AMP-PNP(CydC)/AMP-PNP(CydD) bound state (Dataset-13)  EMDB-14662: IF(heme/coordinated) conformation of CydDC in AMP-PNP(CydC)/AMP-PNP(CydD) bound state (Dataset-13)  EMDB-14663: IF(heme/coordinated) conformation of CydDC in AMP-PNP(CydC)/AMP-PNP(CydD) bound state (Dataset-14)  EMDB-14665: IF(heme/confined) conformation of CydDC in AMP-PNP(CydD) bound state (Dataset-14) EMDB-14667, PDB-7zdr: EMDB-14668, PDB-7zds: EMDB-14669, PDB-7zdt: EMDB-14670, PDB-7zdu: EMDB-14671: IF(apo/as isolated) conformation of CydDC mutant (E500Q.C) in AMP-PNP(CydD) bound state (Dataset-19)  EMDB-14672: Occ(apo/return) conformation of CydDC mutant (E500Q.C) in ATP(CydC)/AMP-PNP(CydD) bound state (Dataset-20) 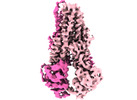 EMDB-14673: IF(heme/confined) conformation of CydDC mutant (E500Q.C) (Dataset-21)  EMDB-14674: Occ(apo/return) conformation of CydDC mutant (E500Q.C) in ATP(CydC) bound state (Dataset-21) EMDB-14675, PDB-7zdw:  EMDB-14676: Occ(apo/return) conformation of CydDC mutant (E500Q.C) in ATP(CydC)/AMP-PNP(CydD) bound state (Dataset-22) EMDB-14684, PDB-7ze5: EMDB-14689, PDB-7zec:  EMDB-15264: IF(apo/as isolated) conformation of CydDC mutant (H85A.C) in AMP-PNP(CydD) bound state (Dataset-17)  EMDB-15265: IF(heme/coordinated) conformation of CydDC mutant (E500Q.C) in AMP-PNP(CydC)/AMP-PNP(CydD) bound state (Dataset-23) |
| 化合物 |  ChemComp-MG: 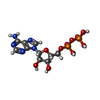 ChemComp-ADP:  ChemComp-PO3: 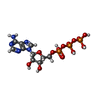 ChemComp-ATP:  ChemComp-HEB: 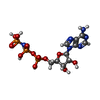 ChemComp-ANP:  ChemComp-ISW: |
| 由来 |
|
 キーワード キーワード |  MEMBRANE PROTEIN (膜タンパク質) / MEMBRANE PROTEIN (膜タンパク質) /  ABC transporter / CydDC / ABC transporter / CydDC /  Heme transporter Heme transporter |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について