+検索条件
-Structure paper
| タイトル | The antigenic anatomy of SARS-CoV-2 receptor binding domain. |
|---|---|
| ジャーナル・号・ページ | Cell, Vol. 184, Issue 8, Page 2183-2200.e22, Year 2021 |
| 掲載日 | 2021年4月15日 |
 著者 著者 | Wanwisa Dejnirattisai / Daming Zhou / Helen M Ginn / Helen M E Duyvesteyn / Piyada Supasa / James Brett Case / Yuguang Zhao / Thomas S Walter / Alexander J Mentzer / Chang Liu / Beibei Wang / Guido C Paesen / Jose Slon-Campos / César López-Camacho / Natasha M Kafai / Adam L Bailey / Rita E Chen / Baoling Ying / Craig Thompson / Jai Bolton / Alex Fyfe / Sunetra Gupta / Tiong Kit Tan / Javier Gilbert-Jaramillo / William James / Michael Knight / Miles W Carroll / Donal Skelly / Christina Dold / Yanchun Peng / Robert Levin / Tao Dong / Andrew J Pollard / Julian C Knight / Paul Klenerman / Nigel Temperton / David R Hall / Mark A Williams / Neil G Paterson / Felicity K R Bertram / C Alistair Siebert / Daniel K Clare / Andrew Howe / Julika Radecke / Yun Song / Alain R Townsend / Kuan-Ying A Huang / Elizabeth E Fry / Juthathip Mongkolsapaya / Michael S Diamond / Jingshan Ren / David I Stuart / Gavin R Screaton /     |
| PubMed 要旨 | Antibodies are crucial to immune protection against SARS-CoV-2, with some in emergency use as therapeutics. Here, we identify 377 human monoclonal antibodies (mAbs) recognizing the virus spike and ...Antibodies are crucial to immune protection against SARS-CoV-2, with some in emergency use as therapeutics. Here, we identify 377 human monoclonal antibodies (mAbs) recognizing the virus spike and focus mainly on 80 that bind the receptor binding domain (RBD). We devise a competition data-driven method to map RBD binding sites. We find that although antibody binding sites are widely dispersed, neutralizing antibody binding is focused, with nearly all highly inhibitory mAbs (IC < 0.1 μg/mL) blocking receptor interaction, except for one that binds a unique epitope in the N-terminal domain. Many of these neutralizing mAbs use public V-genes and are close to germline. We dissect the structural basis of recognition for this large panel of antibodies through X-ray crystallography and cryoelectron microscopy of 19 Fab-antigen structures. We find novel binding modes for some potently inhibitory antibodies and demonstrate that strongly neutralizing mAbs protect, prophylactically or therapeutically, in animal models. |
 リンク リンク |  Cell / Cell /  PubMed:33756110 / PubMed:33756110 /  PubMed Central PubMed Central |
| 手法 | EM (単粒子) / X線回折 |
| 解像度 | 2.04 - 7.3 Å |
| 構造データ | EMDB-12274, PDB-7nd3: EMDB-12275, PDB-7nd4: EMDB-12276, PDB-7nd5: EMDB-12277, PDB-7nd6: EMDB-12278, PDB-7nd7: EMDB-12279, PDB-7nd8: EMDB-12280, PDB-7nd9: EMDB-12281, PDB-7nda: EMDB-12282, PDB-7ndb: EMDB-12283, PDB-7ndc: EMDB-12284, PDB-7ndd:  PDB-7beh:  PDB-7bei:  PDB-7bej:  PDB-7bek:  PDB-7bel:  PDB-7bem:  PDB-7ben:  PDB-7beo:  PDB-7bep: |
| 化合物 |  ChemComp-GOL:  ChemComp-TRS:  ChemComp-HOH:  ChemComp-NO3:  ChemComp-CL:  ChemComp-FMT:  ChemComp-PEG:  ChemComp-SO4:  ChemComp-ACT:  ChemComp-NAG: 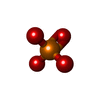 ChemComp-PO4: 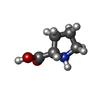 ChemComp-PRO:  ChemComp-IMD:  ChemComp-BR:  ChemComp-PG6:  ChemComp-IOD:  ChemComp-GLU: 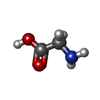 ChemComp-GLY: 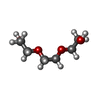 ChemComp-PGE:  ChemComp-PG4: |
| 由来 |
|
 キーワード キーワード |  VIRAL PROTEIN/IMMUNE SYSTEM (ウイルス性) / VIRAL PROTEIN/IMMUNE SYSTEM (ウイルス性) /  SARS-CoV-2 (SARSコロナウイルス2) / SARS-CoV-2 (SARSコロナウイルス2) /  antibody (抗体) / antibody (抗体) /  germline / V-gene / receptor-binding-domain / spike / neutralisation / germline / V-gene / receptor-binding-domain / spike / neutralisation /  protection / protection /  glycosylation (グリコシル化) / valency / glycosylation (グリコシル化) / valency /  VIRAL PROTEIN (ウイルスタンパク質) / VIRAL PROTEIN (ウイルスタンパク質) /  IMMUNE SYSTEM (免疫系) / IMMUNE SYSTEM (免疫系) /  VIRAL PROTEIN-IMMUNE SYSTEM complex (ウイルス性) VIRAL PROTEIN-IMMUNE SYSTEM complex (ウイルス性) |
 ムービー
ムービー コントローラー
コントローラー 構造ビューア
構造ビューア 万見文献について
万見文献について



























